Selection and Sequence Alignment of PCR Primers for Identifying Zizhi Strain
-
摘要:目的 紫芝S2(品种名:武芝2号)是近年来在福建及周边省份推广应用的紫芝栽培新品种,为了避免与遗传背景不同的栽培菌株混杂而建立有效的分子标记。方法 采用PCR扩增筛选条带清晰稳定、呈现多态性的引物,根据菌株间UPMGA聚类构建系统发生树,通过遗传距离确定主要栽培菌株间的亲缘关系,并与菌株间的拮抗反应结果相映证,进而与‘紫芝S2’基因组序列进行比对验证。结果 筛选得到能清晰且稳定地扩增出特异性或多态性条带的2个RAPD-PCR引物和3个ISSR-PCR引物,比对发现这5个引物短序列与‘紫芝S2’基因组序列完全匹配上的位点与数目不同。结论 基于紫芝S2基因组序列比对,确认其中3个引物(ISSR13、S1326和S1506)可以有效地用于紫芝栽培菌株鉴定。Abstract:Objective The molecular markers of the cultivated strain of Ganoderma sinense, Zizhi S2 (aka Wu-Zhi No. 2), recently popularized in Fujian and surrounding provinces were studied to facilitate the authentication of the medicinal fungus.Method Relevant primers of Zizhi S2 showing clear and stable bands and polymorphism were screened using PCR. Phylogenetic tree of UPMGA clustering analysis on the verified authentic cultivars was constructed to determine their relationship by genetic distance as well as antagonistic reaction. Subsequently, sequences of the selected primers were blasted on the genome of Zizhi S2 to validate the methodology.Result There were 2 RAPD-PCR and 3 ISSR-PCR primers found to clearly and stably amplify the specific or polymorphic bands. However, the sites and numbers on the scaffolds of the Zizhi S2 genome that matched the sequences of the 5 primers were not same.Conclusion It was confirmed that three primers(ISSR13, S1326, and S1506)could be effectively used for identification of Zizhi cultivated strains based on sequences alignment with the genome of G. sinense strain Zizhi S2.
-
Keywords:
- Ganoderma sinense /
- cultivated strain /
- molecular markers /
- PCR primers /
- sequence alignment
-
0. 引言
【研究意义】灵芝的商品化栽培(规范化种植)起源于20世纪90年代,福建省曾大力推广以赤芝6号等为代表的优良栽培菌株[1-2],成为国内最大的灵芝生产与出口基地。但2000年以来,福建灵芝种植业受国家林业政策等影响,栽培用材短缺难以维持商品化种植的需求(人工培育灵芝专用林的成材期长达10—12年),栽培量一落千丈。同时,长期以来人工栽培和加工利用的灵芝种类(品种)单一,难以满足多样化的市场需要,产业发展缺乏新的增长点。而野生紫芝种质资源的开发利用及其人工驯化栽培技术的推广应用,可再次促进福建省灵芝产业的发展,能成为山区扶贫致富的好项目。福建省灵芝科真菌资源中灵芝属的紫芝类(Sect. Phaeonema)有9种[3],其代表性种类是被称为中国灵芝的紫芝(G. sinense)、其近缘种的硬孔灵芝(G. duropora)[4],这2种紫芝常见于闽西北山区。【前人研究进展】紫芝是灵芝科灵芝属(Ganoderma)的重要类群紫芝组(Sect. Phaeonema),其特征是菌肉呈均匀褐色、深褐色至栗褐色;以Ganoderma sinense为中心的紫芝大种群是属于高温多雨气候型,分布在长江以南的8个省区[5]。相较于栽培较广泛的赤芝(G. 1ucidum)与松杉灵芝(G. tsugae),紫芝种质资源的开发利用相对滞后,市场上的灵芝或者灵芝类产品很少以紫芝为主要原料[6]。紫芝作为中药材灵芝来源菌物,自2000年起被《中国药典》收录,但其栽培种质资源与遗传育种方面的研究报道较少,亟待加强。目前,我国人工栽培的紫芝菌种主要还是野外分离和驯化的菌株[7],如闽紫96和紫芝S2[8-9]。这2个栽培菌株在栽培性状(朵型、大小)或子实体发育形态上有着很大的不同,其中闽紫96菌盖小型,菌柄侧生、偏生为主,栽培单产仅有12.70 kg· m−3(干品/菌材),而紫芝S2菌盖大型,菌柄中生、近中生,栽培单产平均可达25.05 kg·m−3(干品/菌材)。国内外学者根据(微观和宏观)形态特征上的不同,早在2005年就描述了紫芝(G. sinense)的表型可塑性[10],且基于系统发育分析,已有研究者提出将硬孔灵芝(G. duropora)与紫芝(G. sinense)归类为一个新的复合种——紫芝(Ganoderma duroporum comb. nov.)[11]。最近,来自中国境内不同产地的具有高度表型可塑性的20份紫芝标本,从多基因序列比对结果也被证实为同一种[12]。【本研究切入点】经多年多点区域试验和Finlay稳定性分析,紫芝S2受栽培环境因素影响较小(稳定性好,适应性强),于2012年获得新品种认定(品种认定号:闽认菌2012002),定名为武芝2号[13]。紫芝S2适于林下种植与大棚栽培,已在福建省闽西北推广应用多年,年产量可达300 t;近年来,紫芝S2已经被引种至广东、广西、江西和浙江等省份,辽宁、黑龙江也有星零种植。为了避免与其他来源(遗传背景不同)栽培菌的株系混杂,本研究采用RAPD-PCR、ISSR-PCR技术构建紫芝S2(Zizhi S2)的分子标记。【拟解决的关键问题】应用Primer-Blast在线设计,将筛选得到的特异性PCR引物短序列与紫芝S2基因组全序列进行Blast比对,反向验证其有效性,为紫芝菌株鉴别提供参考。
1. 材料与方法
1.1 供试菌株
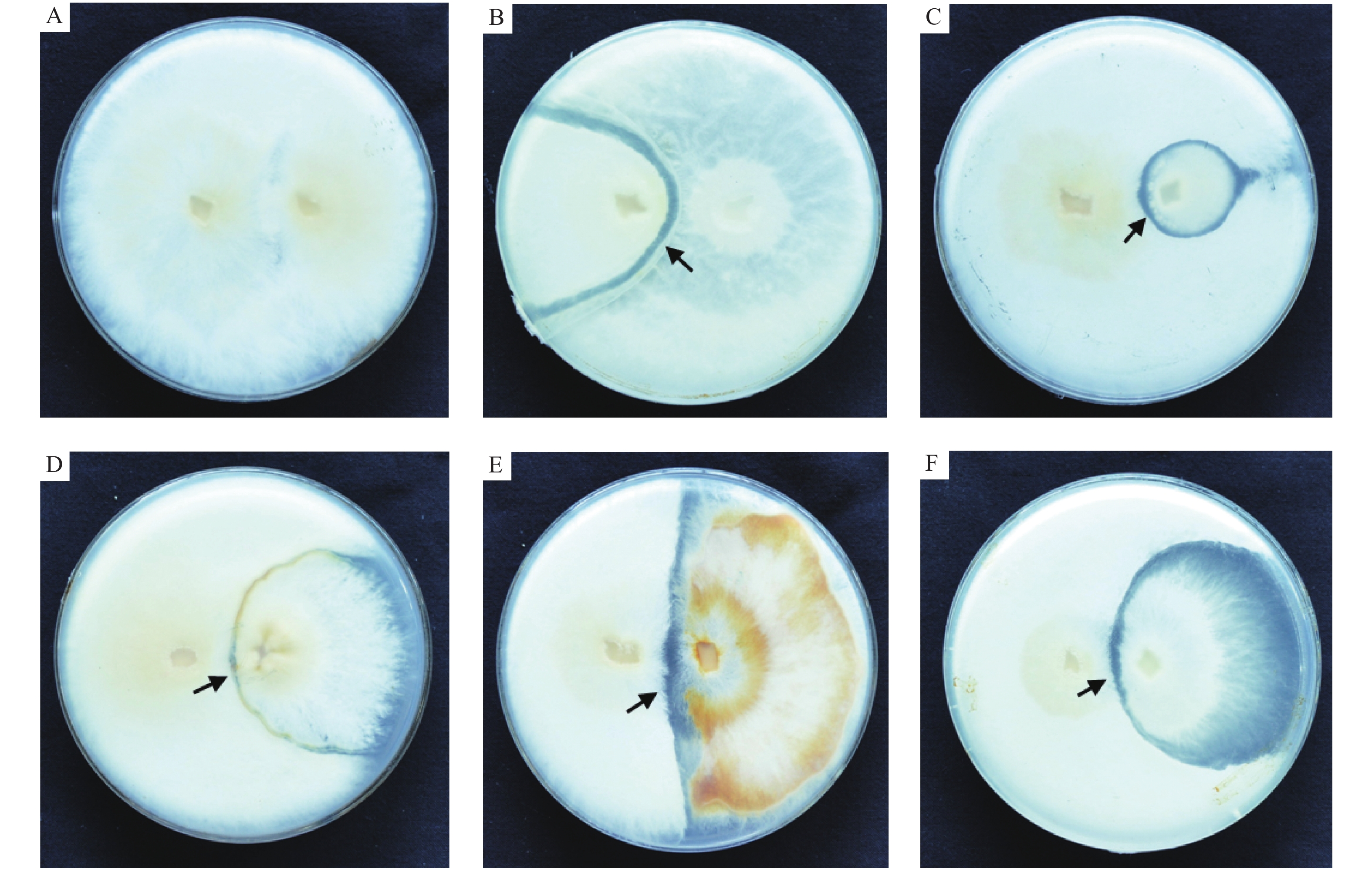
从不同单位得到的6个紫芝栽培常用紫菌株(表1)。其中,1号菌株紫芝S2和2号菌株紫芝X5均由福建省武平县食用菌技术推广服务站选育提供。紫芝S2与紫芝X5、紫芝1、紫芝2、紫芝3、紫芝4等其他5个菌株间均存在不同度的拮抗现象,菌丝表面形成带状拮抗线(图1)。
表 1 供试紫芝菌株信息Table 1. Strains of cultivated Zizhi used in study编号
No.菌株名
Strain name来源
Source1 紫芝 S2 福建省农作物品种审定委员会认定品种(武芝2号) Zizhi S2 New varieties identified by Fujian Crop Variety Approval Committee(Wuzhi No.2) 2 紫芝 X5 福建省武平县食用菌技术推广服务站 Zizhi X5 Fujian Wuping County Edible Fungi Technology Extension Service Statio 3 紫芝1 华中农业大学菌种实验中心 Zizhi 1 Microbial Species Laboratory Center of Huazhong Agricultural Universit 4 紫芝2 上海市农业科学院食用菌研究所 Zizhi 2 Institute of Edible Fungi, Shanghai Academy of Agricultural Sciences 5 紫芝3 江苏省江都天达食用菌研究所 Zizhi 3 Jiangdu Tianda Institute of Edible Fungi, Jiangsu Province 6 紫芝4 辽宁省锦州市亚泰食用菌研究所 Zizhi4 Yatai Institute of Edible Fungi, Jinzhou City, Liaoning Province ![]() 图 1 紫芝菌株间的拮抗现象注:A:紫芝S2-紫芝S2;B:紫芝S2-紫芝X5;C:紫芝S2-紫芝1;D:紫芝S2-紫芝2;E:紫芝S2-紫芝3;F:紫芝S2-紫芝4。Figure 1. Antagonism between different Zizhi strainsNote: A: Zizhi S2 vs. Zizhi S2; B: Zizhi S2 vs. Zizhi X5; C: Zizhi S2 vs. Zizhi 1; D: Zizhi S2 vs. Zizhi 2; E: Zizhi S2 vs. Zizhi 3; and, F: Zizhi S2 vs. Zizhi 4.
图 1 紫芝菌株间的拮抗现象注:A:紫芝S2-紫芝S2;B:紫芝S2-紫芝X5;C:紫芝S2-紫芝1;D:紫芝S2-紫芝2;E:紫芝S2-紫芝3;F:紫芝S2-紫芝4。Figure 1. Antagonism between different Zizhi strainsNote: A: Zizhi S2 vs. Zizhi S2; B: Zizhi S2 vs. Zizhi X5; C: Zizhi S2 vs. Zizhi 1; D: Zizhi S2 vs. Zizhi 2; E: Zizhi S2 vs. Zizhi 3; and, F: Zizhi S2 vs. Zizhi 4.1.2 DNA样品提取与检测
将供试菌株分别接种到贴有玻璃纸的PDA平板中,封口倒置、于27 ℃培养,待菌丝长满平板收集菌丝,于预冷研钵中加入液氮研磨成细粉,采用改良CTAB法提取和纯化总DNA[14]。采用0.75%琼脂糖凝胶电泳(100 V直流1 h,5~80 kb脉冲16 h)检测DNA样品符合建库合格,Nanodrop检测DNA纯度(161.6 ng·μL−1),Qubit快速荧光定量(154.0 ng·μL−1)。
1.3 RAPD、ISSR扩增分析比较
采用Eppendorf Mastercyeter Personal PCR扩增仪,PCR试剂购自TaKaRa有限公司(大连),候选RAPD、ISSR扩增引物均从(上海)生工生物工程股份有限公司提供的引物产品中筛选,PCR反应体系及扩增程序按照参考文献[14]。反应结束后,各取5 μL PCR产物,加1 μL溴酚蓝(6×)混匀,在1.0%琼脂糖凝胶(含Goldview TMDNA染料0.5 μg·mL−1)上电泳,使用Marker为DL2000,于4~5 V·cm−1电压下电泳约1.0 h,在JS-680全自动数码凝胶成像系统上观察并拍照保存。
1.4 引物序列与基因组序列的比对
采用PacBio Sequel三代测序平台完成紫芝S2全基因组测序,组装得到72条Scaffold(其中核基因组71条,另文报道);Blast比对(BLAST, v2.2.26)检测每条Scaffold上是否包含RAPD、ISSR引物的短序列,并统计不同错配比对结果。
1.5 数据统计与处理方法
多态位点百分率计算公式为P =(k/n)×100%,k为多态位点数,n为检测位点总数;遗传相似距离计算采用Nei氏公式S = 2Nxy/(Nx + Ny),其中Nxy为菌株X和Y共有片段数,Nx、Ny分别为X和Y菌株扩增片段总数。采用NTSYSpc2.0软件对菌株RAPD、ISSR分子标记结果进行UPGMA聚类分析,生成供试菌株的遗传聚类关系图。选取比对允许空位数为0的Blast结果(比对分数值Score越大越好,e值越小越好),采用Excel 2010软件进行数据统计,分成比对允许错配为0和1(即Gap=0;Mismatch=0, 1)两类结果。
2. 结果与分析
2.1 鉴别菌株的分子标记短序列
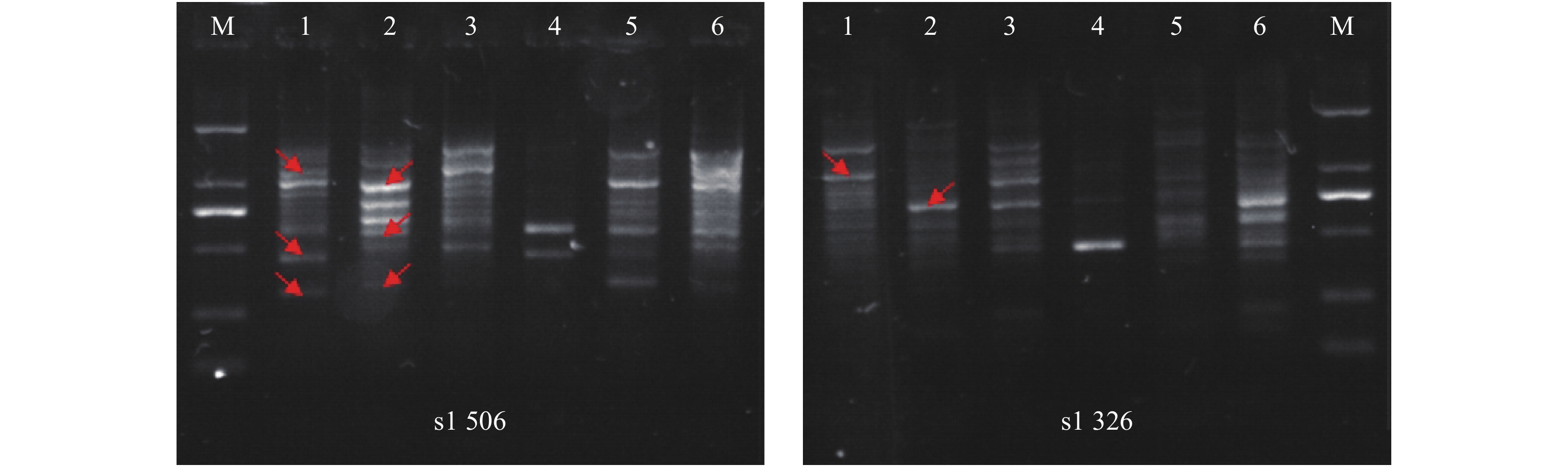
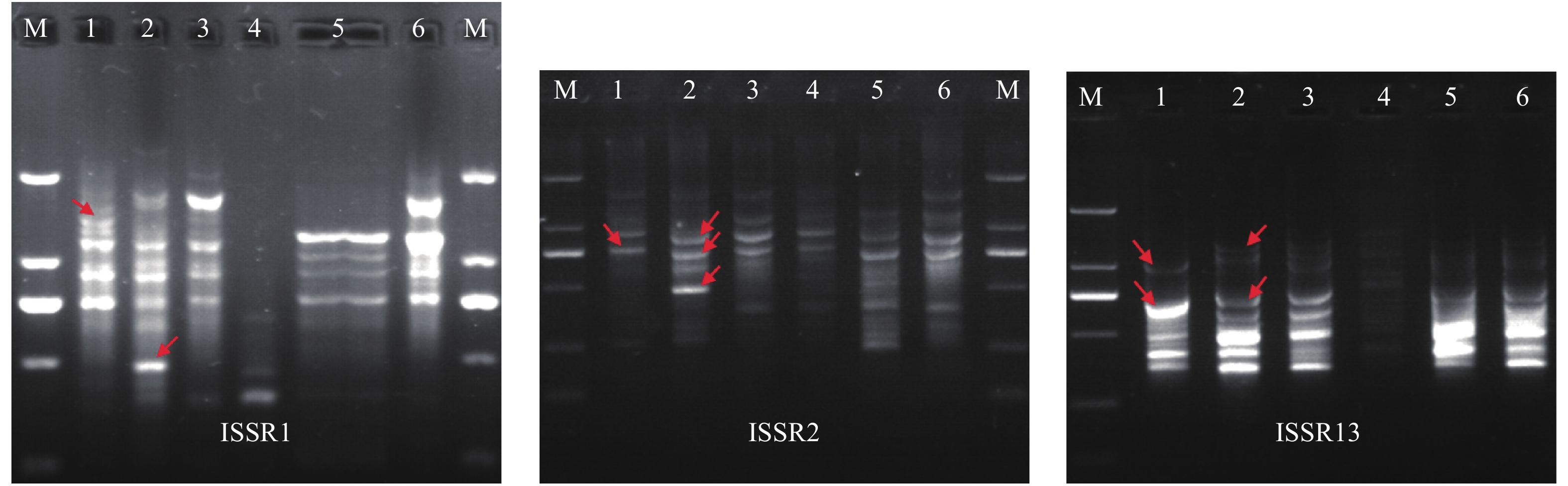
筛选到2个RAPD引物:S1506(GGTGGCCAAG)和S1326(AGGCATCGTG),其扩增效果较好、重复性高,能稳定地扩增出特异性的条带;其中,在紫芝S2、X5的图谱中分别呈现明显区别的1~3特异性条带,并且与4个对照菌株的条带均能明显差异(图2)。同时,还筛选到3个ISSR引物:ISSR1(CACCACACACACACACA)、ISSR2(AGAGACAGAGAGAGAGT)和ISSR13(TTCCCTTCCTTCCC),其扩增效果好、条带清晰且稳定,呈现多态性;共扩增出87条带,其中表现出多态性的条带为59条,多态位点百分率为67.82%。其中,紫芝S2、X5彼此间呈现1~3条特异性条带,也能与4个对照菌株的条带明显区别开来(图3)。分析结果表明:采用S1506、S1326、ISSR1、ISSR2、ISSR13共5个短序列引物,扩增得到RAPD和ISSR图谱,均能够简单而有效地将紫芝S2与其他紫芝菌株鉴别,可以考虑用于分子标记。
2.2 不同紫芝菌株间的遗传距离
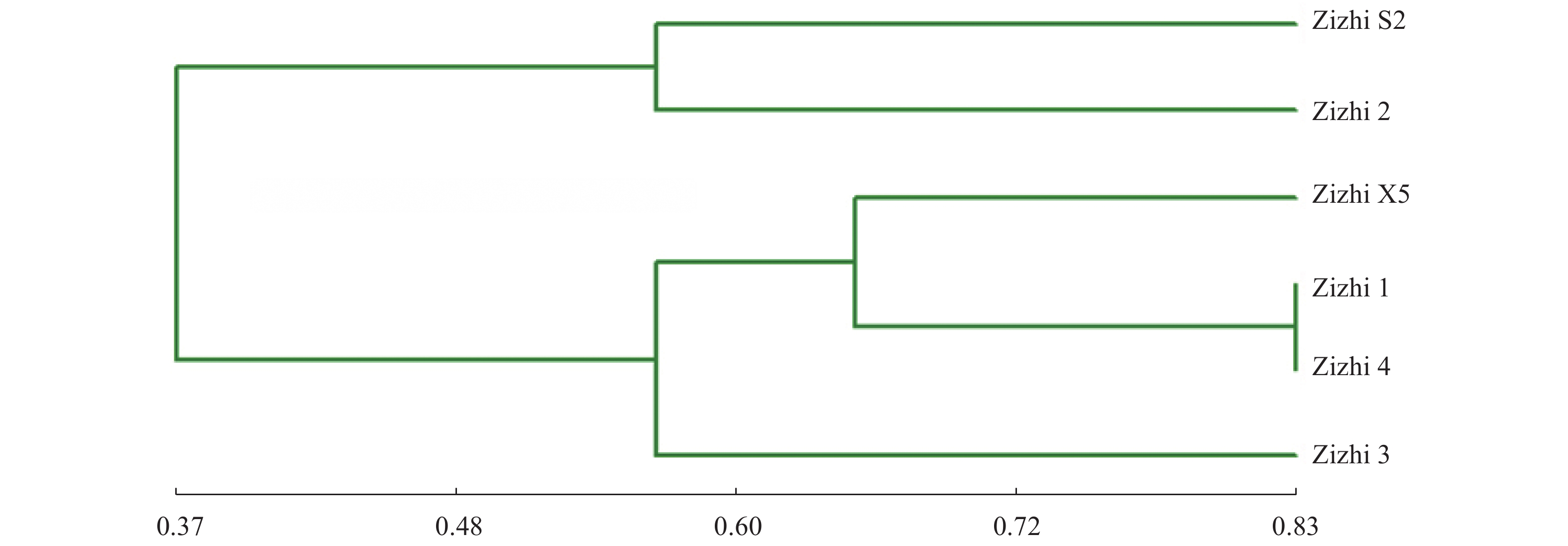
通过聚类分析建立遗传相似距离矩阵,得到聚类图(图4)。结果表明,6株菌株相互间的遗传相似距离在0.564~0.836;其中,1号菌株(紫芝S2)与4号菌株(紫芝2)之间的亲缘关系最近,而与5号菌株(紫芝3)之间的亲缘关系最远。已知紫芝S2与紫芝2之间拮抗不明显,而与紫芝3之间拮抗明显(图1),这2个试验结果相印证。
2.3 RAPD和ISSR引物序列与基因组的比对结果
将筛选得到5个引物短序列(标为*_碱基数:S1506_10、S132610、ISSR1-17、ISSR2_17和ISSR13_14)与紫芝S2的基因组序列进行Blast比对,结果发现:除ISSR1_17外,其他4个引物都能比对上,均在基因组中不同Scaffold上的多个位点找到与引物相匹配的序列片段(表2)。在允许一个(单碱基)错配或空位情况下,在18条Scaffold上有31个片段(17 bp)与ISSR-PCR引物ISSR2_17相匹配(Identity=93.75~94.12),同时在18条Scaffold上找到31个片段(14 bp)与ISSR13_14(完全匹配Identity=100),在48条Scaffold上找到158个片段(10 bp)与S1326_10匹配(Identity=100%),在47条Scaffold上找到172个片段(10 bp)与S1506_10(Identity=100%)相匹配。如不允许错配或空位,则在5条Scaffold上有6个片段(14 bp)与引物ISSR13_14完全匹配(Identity=100%),在48条Scaffold上发现共有158个片段(10 bp)与引物S1326_10完全匹配,在47条Scaffold上共有172个位点片段(10碱基)与S1506_10完全匹配。因此,本研究最终推荐ISSR13、S1326和S1506这3个PCR引物作为鉴别紫芝菌株的分子标记。
表 2 PCR引物与基因组比对结果Table 2. Blast result of PCR primer sequences on genomes引物名称_碱基数
Primer name _Base number比对上引物序列的 Scaffold 数目
All scaffold number与引物匹配的位点数
All primer number比对相似度
Identity(%)不允许错配(空位为0)No mismatches allowed(Gap=0) ISSR13_14 5 6 100 S1326_10 48 158 100 S1506_10 47 172 100 允许一个错配(空位为0)Allow one mismatch(Gap=0) ISSR2_17 11 14 93.75~94.12 ISSR13_14 18 31 100 S1326_10 48 158 100 S1506_10 47 172 100 注:引物ISSR13短序列出现在Scaffold_1上2个位点,引物S1326出现在Scaffold_1上15个位点;S1506引物出现在Scaffold_1上17个位点,出现在Scaffold_60上5个位点。
Note: Short sequence of ISSR 13 appeared at two sites on Scaffold No. 1; S1326 at 15 sites on Scaffold No. 1; and S1506 at 17 sites on Scaffold No. 1 and 5 sites on Scaffold No. 60.3. 讨论与结论
已知在灵芝属药用真菌的遗传多样性研究中可以通过拮抗反应进行初步分类,然后再通过分子生物学的方法进行深入研究[15~17]。本研究发现,紫芝S2菌株与其他紫芝菌株之间存在拮抗反应,遗传背景明显不同的个体在菌丝交界区会形成明显的拮抗线,可以据此进行少量紫芝菌株的初步分类与鉴别;但当菌株数量众多时,工作量大且无法量化而不适用。本研究通过筛选到有价值的2~3个RAPD/ISSR引物,PCR扩增得到的DNA片段呈现条带清晰稳定、多态性和特异性的电泳指纹图谱,可用于紫芝新品种武芝二号(紫芝S2)与其他5个紫芝菌株的鉴别。在此基础上,通过遗传距离确定其亲缘关系,与菌株间的拮抗反应相一致;进一步通过与紫芝S2菌株基因组序列比对,确认利用其中3个引物(ISSR13、S1326和S1506)作为鉴别紫芝栽培品种或菌株简单有效的分子标记物。
2012年以来公布的几种灵芝核基因组[18~21],其中组装质量高和注释信息较完整的有赤芝基因组全序列总长43.29 Mb(82条Scaffolds, 16113个蛋白质编码基因)[19],紫芝基因组全序列总长48.96 Mb(69条Scaffolds, 15688个蛋白质编码基因)[20];本研究同期完成紫芝S2三代测序,组装得到的核基因组序列全长56.76 Mb,包含71条Scaffolds,注释出16681蛋白质编码基因(另文报道)。在此基础上,既可以基于灵芝基因组序列筛选出有效SSR位点用于快捷、准确地标记区分灵芝菌株间亲缘关系(另文报道),也可以将筛选得到的有效鉴别紫芝菌株的RAPD/ISSR-PCR引物(短序列)与紫芝基因组序列进行Blast比对验证。
致谢:本研究依托特色食用菌繁育与栽培国家地方(福建省农业科学院)联合工程研究中心、福建农林大学菌物研究中心、福建省农业科学院药用植物研究中心的科研平台完成。
-
图 1 紫芝菌株间的拮抗现象
注:A:紫芝S2-紫芝S2;B:紫芝S2-紫芝X5;C:紫芝S2-紫芝1;D:紫芝S2-紫芝2;E:紫芝S2-紫芝3;F:紫芝S2-紫芝4。
Figure 1. Antagonism between different Zizhi strains
Note: A: Zizhi S2 vs. Zizhi S2; B: Zizhi S2 vs. Zizhi X5; C: Zizhi S2 vs. Zizhi 1; D: Zizhi S2 vs. Zizhi 2; E: Zizhi S2 vs. Zizhi 3; and, F: Zizhi S2 vs. Zizhi 4.
表 1 供试紫芝菌株信息
Table 1 Strains of cultivated Zizhi used in study
编号
No.菌株名
Strain name来源
Source1 紫芝 S2 福建省农作物品种审定委员会认定品种(武芝2号) Zizhi S2 New varieties identified by Fujian Crop Variety Approval Committee(Wuzhi No.2) 2 紫芝 X5 福建省武平县食用菌技术推广服务站 Zizhi X5 Fujian Wuping County Edible Fungi Technology Extension Service Statio 3 紫芝1 华中农业大学菌种实验中心 Zizhi 1 Microbial Species Laboratory Center of Huazhong Agricultural Universit 4 紫芝2 上海市农业科学院食用菌研究所 Zizhi 2 Institute of Edible Fungi, Shanghai Academy of Agricultural Sciences 5 紫芝3 江苏省江都天达食用菌研究所 Zizhi 3 Jiangdu Tianda Institute of Edible Fungi, Jiangsu Province 6 紫芝4 辽宁省锦州市亚泰食用菌研究所 Zizhi4 Yatai Institute of Edible Fungi, Jinzhou City, Liaoning Province 表 2 PCR引物与基因组比对结果
Table 2 Blast result of PCR primer sequences on genomes
引物名称_碱基数
Primer name _Base number比对上引物序列的 Scaffold 数目
All scaffold number与引物匹配的位点数
All primer number比对相似度
Identity(%)不允许错配(空位为0)No mismatches allowed(Gap=0) ISSR13_14 5 6 100 S1326_10 48 158 100 S1506_10 47 172 100 允许一个错配(空位为0)Allow one mismatch(Gap=0) ISSR2_17 11 14 93.75~94.12 ISSR13_14 18 31 100 S1326_10 48 158 100 S1506_10 47 172 100 注:引物ISSR13短序列出现在Scaffold_1上2个位点,引物S1326出现在Scaffold_1上15个位点;S1506引物出现在Scaffold_1上17个位点,出现在Scaffold_60上5个位点。
Note: Short sequence of ISSR 13 appeared at two sites on Scaffold No. 1; S1326 at 15 sites on Scaffold No. 1; and S1506 at 17 sites on Scaffold No. 1 and 5 sites on Scaffold No. 60. -
[1] 李开本, 陈体强, 何修金, 等. 短段木灵芝优质高产菌株“赤芝6号”研究 [J]. 福建省农科院学报, 1996, 11(3):1−6. LI K B, CHEN T Q, HE X J, et al. Study on“Red Reishi No. 6”, a high-yield and fine-quality strain of Ganoderma lucidum for woodlqg cultivation [J]. Fujian Journal of Agricultural Sciences, 1996, 11(3): 1−6.(in Chinese)
[2] 林兴生, 李开本, 陈体强, 等. 28个灵芝菌株栽培特性比较 [J]. 食用菌学报, 2001, 8(3):45−49. DOI: 10.3969/j.issn.1005-9873.2001.03.009 LIN X S, LI K B, CHEN T Q, et al. A comparative study on the cultivation characteristics of 28 Ganoderma lucidum strains [J]. Acta Edulis Fungi, 2001, 8(3): 45−49.(in Chinese) DOI: 10.3969/j.issn.1005-9873.2001.03.009
[3] 陈体强, 林兴生, 赵健, 等. 福建灵芝科真菌资源及担孢子形态结构数据库研究 [J]. 福建农业学报, 2002, 17(1):40−44. DOI: 10.3969/j.issn.1008-0384.2002.01.011 Chen T Q, Lin X S, ZHAO J, et al. Resource of Ganodermataceae in Fujian Province and its morphology and structure database of basidospores [J]. Fujian Journal of Agricultural Sciences, 2002, 17(1): 40−44.(in Chinese) DOI: 10.3969/j.issn.1008-0384.2002.01.011
[4] 赵继鼎. 中国灵芝科的分类研究: XI.灵芝亚属灵芝组 [J]. 真菌学报, 1989(1):25−34. ZHAO J D. Studies on the taxonomy of Ganodermataceae in China vi [J]. Mycosystema, 1989(1): 25−34.(in Chinese)
[5] 赵继鼎, 张小青. 中国真菌志(第十八卷)·灵芝科[M]. 北京: 科学出版社, 2000, 70: 86-87&103-106. [6] 李钦艳, 钟莹莹, 陈逸湘, 等. 我国灵芝种质资源及生产技术研究进展 [J]. 中国食用菌, 2016, 35(1):8−12, 20. LI Q Y, ZHONG Y Y, CHEN Y X, et al. Research advances on the production technology of Ganoderma in China [J]. Edible Fungi of China, 2016, 35(1): 8−12, 20.(in Chinese)
[7] 陈逸湘, 宋斌, 李挺, 等. 紫芝研究进展 [J]. 广东农业科学, 2011, 38(24):36−39. DOI: 10.3969/j.issn.1004-874X.2011.24.014 CHEN Y X, SONG B, LI T, et al. Research on advances of Ganoderma sinense [J]. Guangdong Agricultural Sciences, 2011, 38(24): 36−39.(in Chinese) DOI: 10.3969/j.issn.1004-874X.2011.24.014
[8] 陈体强, 吴锦忠, 李晔, 等. 福建野生紫芝资源开发利用Ⅱ.“闽紫96”(中国灵芝) [J]. 菌物研究, 2006, 4(4):27−32. DOI: 10.3969/j.issn.1672-3538.2006.04.005 CHEN T Q, WU J Z, LI Y, et al. Exploitation and utilization of wild Zhizi resources(sect. phaeonema of Ganoderma) in Fujian (Ⅱ)/. G. sinense “minzi 96” [J]. Journal of Fungal Research, 2006, 4(4): 27−32.(in Chinese) DOI: 10.3969/j.issn.1672-3538.2006.04.005
[9] 钟礼义, 陈体强, 邱福平. 不同紫芝菌株特性比较及边材栽培研究初报 [J]. 食用菌, 2005, 27(1): 26-27. ZHONG L Y, CHEN T Q, QIU F P, et al. Primary study on cultivation of Zizhi with broad-leaved sapwood and comparative characteristics of different strains[J]. Edible Fungi, 2005, 27(1): 26-27. (in Chinese)
[10] 吴兴亮, 戴玉成. 中国灵芝图鉴 [M]. 北京: 科学出版社, 2005. [11] 王新存. 灵芝科系统发育研究 [D]. 北京: 中国科学院大学, 2012. WANG X C. Phylogenetic studies of Ganodermataceae [D]. Beijing: University of Chinese Academy of Sciences, 2012.(in Chinese)
[12] HAPUARACHCHI K K. High phenotypic plasticity of Ganoderma sinense (ganodermataceae, polyporales) in China [J]. Asian Journal of Mycology, 2019, 2(1): 1−47. DOI: 10.5943/ajom/2/1/1
[13] 钟礼义. 紫芝新品种武芝2号区域试验 [J]. 中国食用菌, 2013, 32(6):25−27. ZHONG L Y. The regional test on new varieties of Zizhi (Ganoderma) [J]. Edible Fungi of China, 2013, 32(6): 25−27.(in Chinese)
[14] 应正河. RAPD、SRAP和ISSR标记在香菇种质资源的应用及其SCAR标记的建立 [D]. 福州: 福建农林大学. 2006. YIN Z H. Application of RAPD, SRAP and ISSR markers in germplasm resources of Lentinus edodes and establishment of SCAR markers [D]. Fuzhou: Fujian Agriculture and Forestry University, 2005. (in Chinese)
[15] 唐传红, 张劲松, 陈明杰, 等. 利用拮抗试验和RAPD对灵芝属菌株进行分类研究 [J]. 微生物学通报, 2005, 32(5):72−76. DOI: 10.3969/j.issn.0253-2654.2005.05.015 TANG C H, ZHANG J S, CHEN M J, et al. Study on classification of strains of Ganoderma by anatagonistic effect and rapd [J]. Microbiology, 2005, 32(5): 72−76.(in Chinese) DOI: 10.3969/j.issn.0253-2654.2005.05.015
[16] 王锦锋, 李晶, I.L.Datti, et al 利用拮抗、ITS和RAPD技术对灵芝属菌株分类的研究 [J]. 西南农业学报, 2017, 30(1):26−33. WANG J F, LI J, DATTI I, et al. Study on classification of 21 strains of Ganoderma by antagonistic effect, ITS and RAPD technology [J]. Southwest China Journal of Agricultural Sciences, 2017, 30(1): 26−33.(in Chinese)
[17] 徐凯, 唐传红, 王天娇, 等. 基于SRAP、ISSR和RAPD分析灵芝G0130菌株单核体多态性 [J]. 工业微生物, 2014, 44(4):39−45. DOI: 10.3969/j.issn.1001-6678.2014.04.008 XU K, TANG C H, WANG T J, et al. Polymorphism of monokaryons of Ganoderma lucidum G0130 based on SRAP, ISSR and RAPD analysis [J]. Industrial Microbiology, 2014, 44(4): 39−45.(in Chinese) DOI: 10.3969/j.issn.1001-6678.2014.04.008
[18] LIU D B, GONG J, DAI W K, et al. The genome of Ganoderma lucidum provides insights into triterpenes biosynthesis and wood degradation [J]. PLoS One, 2012, 7(5): e36146. DOI: 10.1371/journal.pone.0036146
[19] CHEN S L, XU J, LIU C, et al. Genome sequence of the model medicinal mushroom Ganoderma lucidum [J]. Nature Communications, 2012, 3: 913. DOI: 10.1038/ncomms1923
[20] ZHU Y J, XU J, SUN C, et al. Chromosome-level genome map provides insights into diverse defense mechanisms in the medicinal fungus Ganoderma sinense [J]. Scientific Reports, 2015, 5: 11087. DOI: 10.1038/srep11087
[21] 龚明, 鲍大鹏, 唐传红, 等. 沪农灵芝1号全基因组分析和演化比较 [J]. 食用菌学报, 2016, 23(2):1−5, 19. GONG M, BAO D P, TANG C H, et al. Genome-wide analysis and evolution comparison of Ganoderma lucidum SH [J]. Acta Edulis Fungi, 2016, 23(2): 1−5, 19.(in Chinese)
-
期刊类型引用(4)
1. 刘琳玲,杨义明,张瑞,闫梅霞. 灵芝分子标记利用研究进展. 特产研究. 2024(01): 163-167 .  百度学术
百度学术
2. 王辰雨,宋帅,陈小红,夏志兰,谢玲. 7个野生云芝基于ITS序列和ISSR的亲缘关系分析. 中国食用菌. 2023(03): 42-49+56 .  百度学术
百度学术
3. 钟礼义,应正河,刘新锐,陈体强. 紫芝栽培新品种武芝2号基于rDNA-ITS和ITS2的分子鉴定与分析. 广东农业科学. 2022(02): 9-15 .  百度学术
百度学术
4. 陈体强,徐晓兰,石林春,钟礼义. 紫芝栽培品种‘武芝2号’(‘紫芝S2’)全基因组测序及分析. 生物技术通报. 2021(11): 42-56 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载: