Transcriptome-based Identification and Expressions of Amaranth TCP Gene Family
-
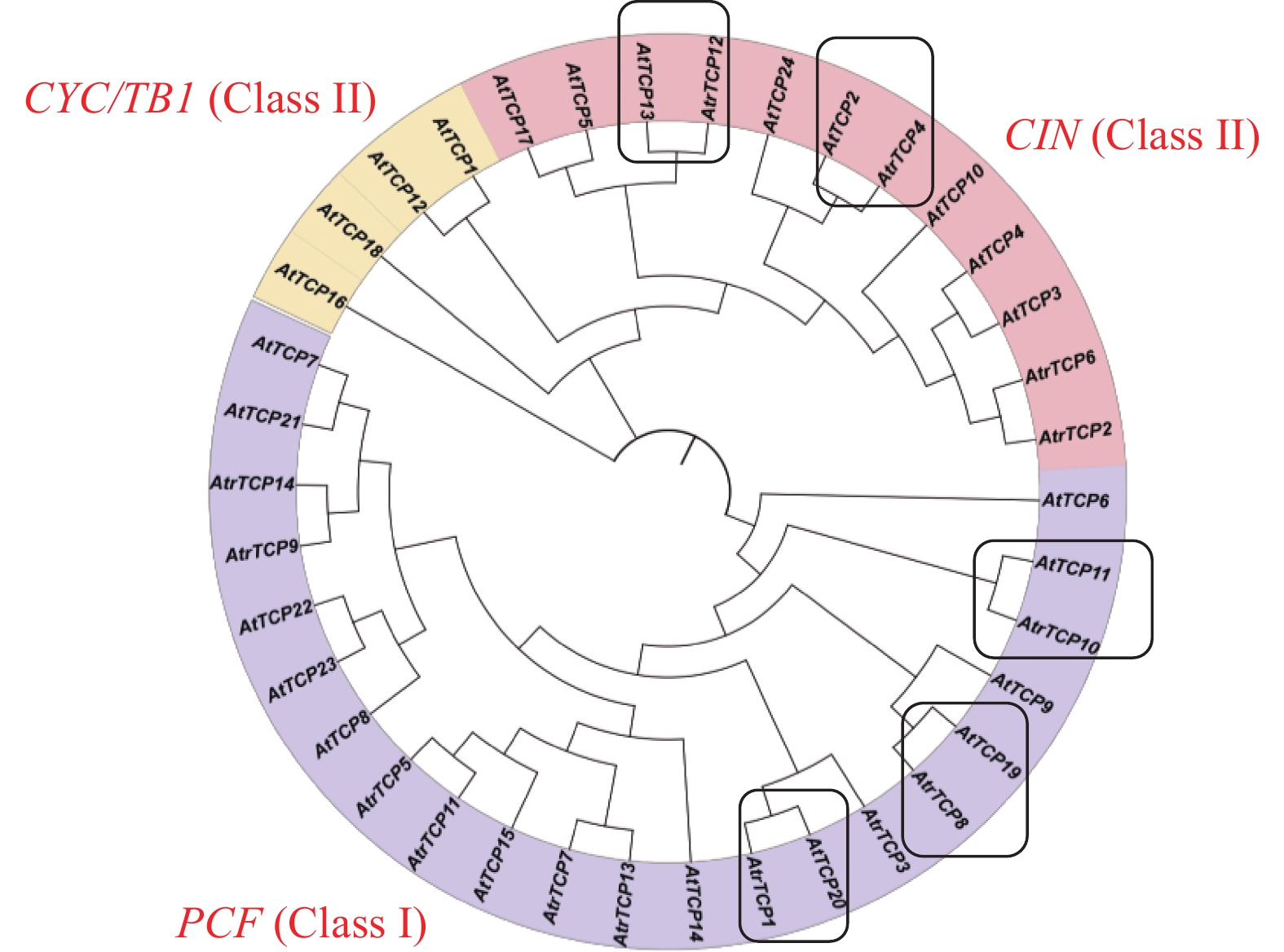
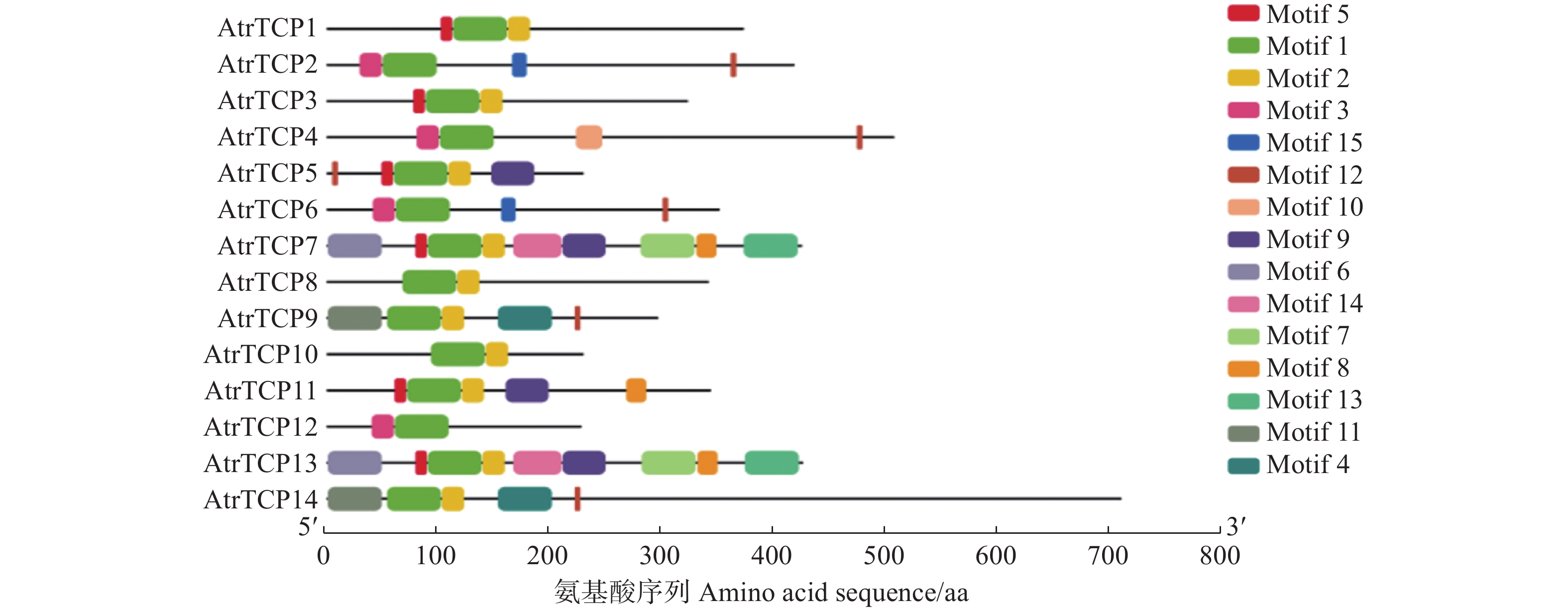
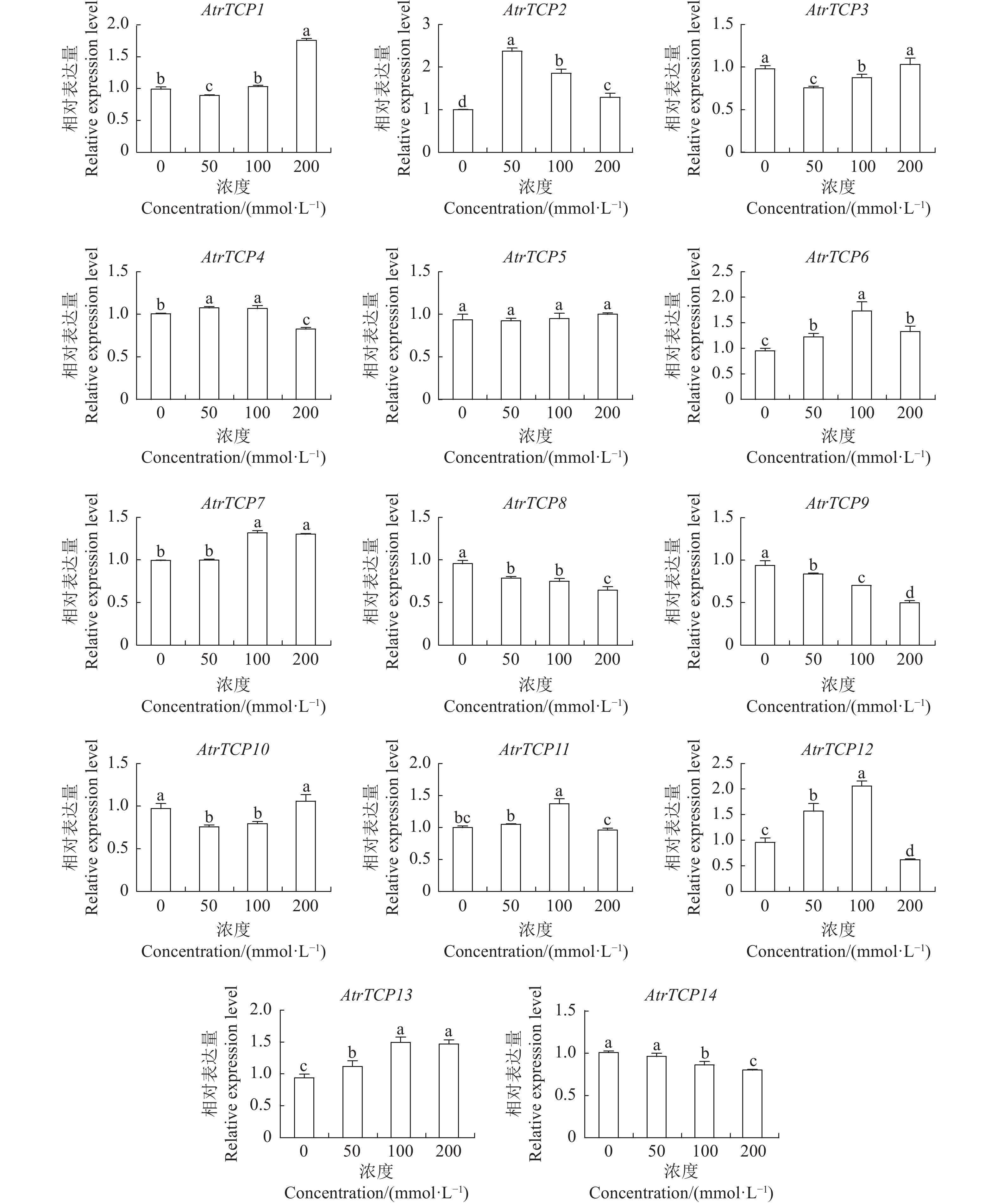
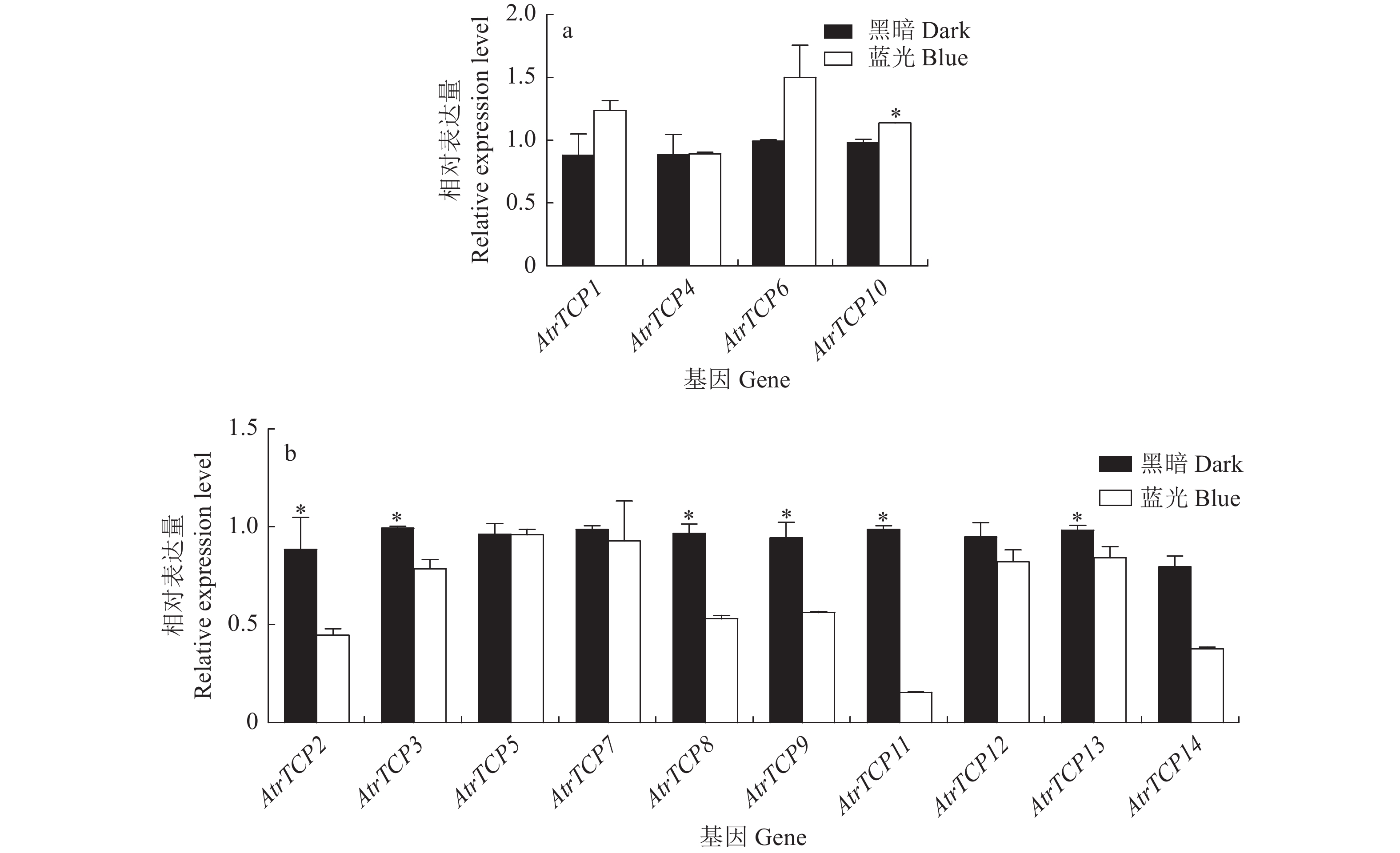
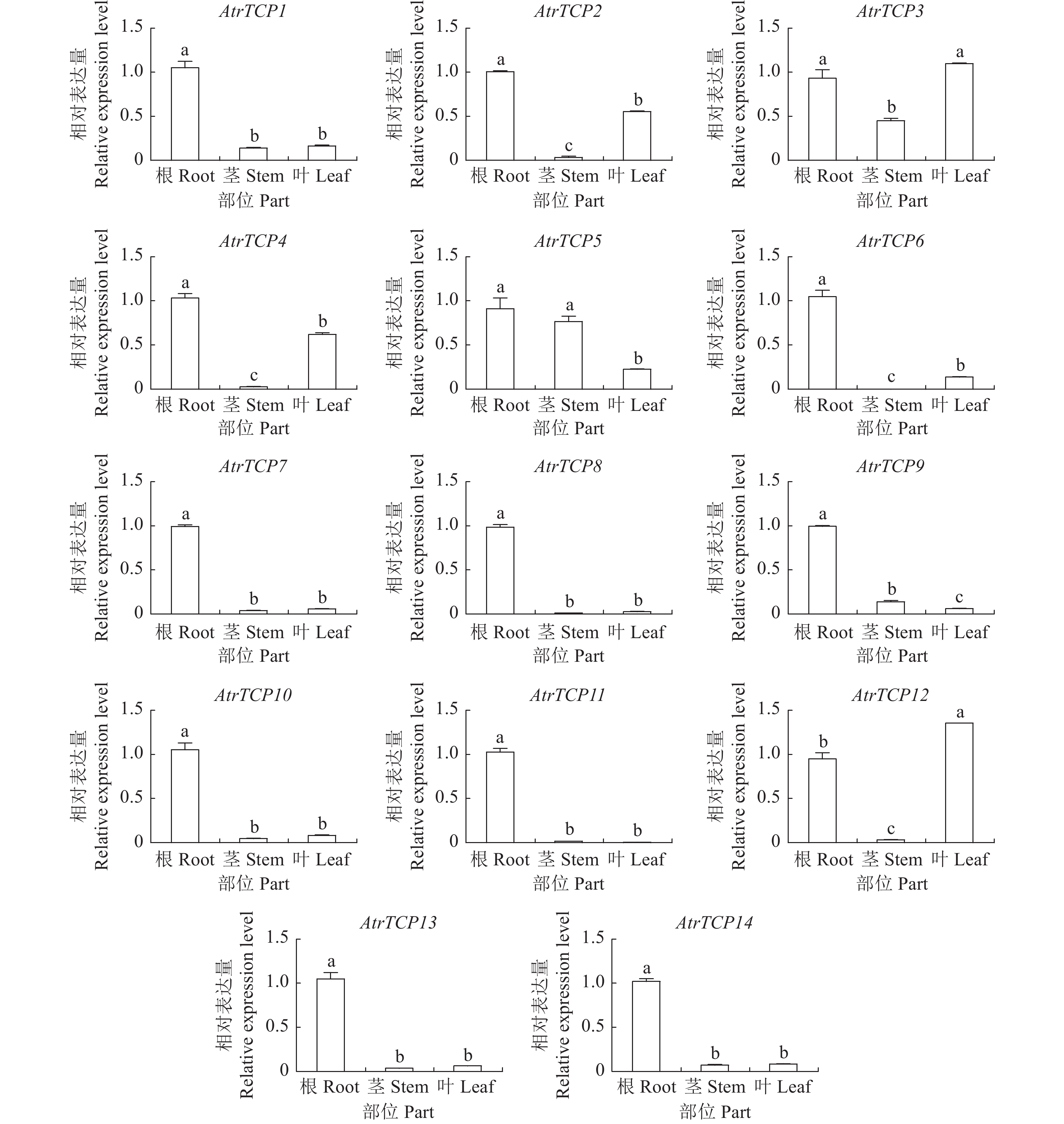
摘要:目的 基于大红苋菜转录组数据库,对苋菜TCP基因家族成员进行筛选鉴定,为深入探讨苋菜TCP基因家族对苋菜生长发育及非生物胁迫响应提供参考。方法 在转录组数据库中,筛选鉴定TCP基因家族成员,并对其进行生物信息学分析;在不同条件处理下培养苋菜幼苗,并利用qRT-PCR技术分析其在不同处理下的表达模式。结果 苋菜TCP基因家族有14个成员,全部定位于细胞核;AtrTCP蛋白质长度在230~721 aa区间内,预测相对分子量为25.25~78.57 kD,等电点为6.15~9.45,包含15个保守基序;除AtrTCP11为稳定蛋白外,其余均为不稳定蛋白;miRNA预测到两个AtrTCP家族成员AtrTCP2和AtrTCP6为miR319靶基因。qRT-PCR结果显示:在盐溶液不同浓度(0、50、100 、200 mmol·L-1)处理下,该基因家族具有不同的表达模式;在蓝光处理下,AtrTCP10基因表达量上调达到了显著水平;AtrTCP2、AtrTCP3、AtrTCP8、AtrTCP9、AtrTCP11和AtrTCP13表达量下调达到了显著水平;在不同铵硝比(0∶0、0∶10、3∶7、5∶5、7∶3、10∶0)处理下,TCP基因家族各成员表达模式不同,其中,AtrTCP3参与整个氮代谢过程;在苋菜各部位进行qRT-PCR分析发现:AtrTCP3、AtrTCP12 在叶片中高表达,其余家族成员均在根部的表达量较高。结论 在不同条件处理下,苋菜TCP基因家族各成员表达模式不同,表明苋菜TCP基因家族各成员可能广泛参与非生物胁迫。Abstract:Objective Members of the amaranth TCP gene family were identified based on the transcriptome database for an in-depth understanding on the roles they associated with the growth, development, and abiotic stress of the plant.Method Members of the TCP gene family were identified by screening the transcriptome database on Dahong amaranth (Amaranthus tricolor L.) for a bioinformatics analysis. Amaranth seedlings were cultured under different conditions and their expressions under different treatments analyzed by qRT-PCR.Results Amaranth TCP gene family has 14 members, all localized in the nucleus. The length of AtrTCP protein was in the range of 230–721 aa, the predicted relative molecular weight was 25.25–78.57 kD, the isoelectric point was 6.15–9.45, and it contained 15 conserved motifs. Except AtrTCP11, which was stable protein, all the other proteins were unstable. Two AtrTCP family members, AtrTCP2 and AtrTCP6, were predicted as miR319 target genes by miRNA. qRT-PCR showed differentiated expressions of the family members under salt solutions of varied concentrations (i.e., 0, 50, 100, and 200 mmol·L−1). The expression of the upregulated AtrTCP10 reached a significant level under blue light, and so did those of the downregulated AtrTCP2, AtrTCP3, AtrTCP8, AtrTCP9, AtrTCP11, and AtrTCP13. Under different concentrations of ammonium nitrate ratio (0∶0, 0∶10, 3∶7, 5∶5, 7∶3, 10∶0) treatment, the expressions of the TCP genes differed. AtrTCP3 was shown to be involved in the entire nitrogen metabolism process. In tissues of amaranth, AtrTCP3 and AtrTCP12 were highly expressed in leaves, while the others in roots.Conclusion Depending on the treatments, the expressions of the amaranth TCP gene family members differed. It suggested a broad spectrum of involvements by the members associated with abiotic stresses on amaranth.
-
Keywords:
- Amaranthus tricolor L. /
- transcription factor /
- gene family /
- bioinformatics analysis /
- expression
-
0. 引言
【研究意义】茶渣是茶饮料、速溶茶和茶单宁产业等加工茶叶后产生的残渣,据统计,仅茶饮料和速溶茶公司每年产生的茶渣就达16万t[1]。研究表明,干基的茶渣粗蛋白质含量为17%~25%,是一种良好的蛋白质饲料资源[2-4]。日本的佐野满昭[5]在饲料中添加3%的茶渣喂养肉鸡,可提高鸡肉的嫩度和脂溶性抗氧化物(VE、VC)含量,降低鸡肉脂质过氧化物含量。徐瑞等[6]研究表明,在日粮中添加2%的茶叶渣能提高育肥猪的生长性能,改善猪肉的色泽和保水性能。吴慧敏[7]研究发现在蛋鸡的日粮中添加1%~2%的茶渣不仅显著增加产蛋数和蛋重观察蛋鸡的生产性能,还能显著提高血清中的GSH-Px,T-SOD,IgA,IgM含量,同时显著的降低血清中的胆固醇含量。马帮军[8]研究表明,猪日粮中添加茶粉1%~3%会降低猪的平均日增重,但显著降低猪背膘厚度,减少脂肪在猪胴体中的沉积。潘发明[9]等则认为饲粮中添加茶渣比例过高,会影响适口性,降低绵羊采食量。主要原因是茶渣粗纤维含量高,畜禽纤维素酶活力低,不利于消化吸收;另一原因,刚出产的茶渣水分达到80%不易储藏和运输,因此仅有小部分的茶渣作为饲料源利用,绝大部分茶渣被丢弃或掩埋;这不仅造成资源浪费,而且造成生态环境污染[10-11]。【前人研究进展】为了更好利用茶渣资源,刘姝等[12]利用木霉等组合微生物发酵茶渣,在30 ℃下发酵4~8 d后发现,饲料中粗蛋白测定含量>25%,比对照提高了20%,其营养价值达到了仔猪配合饲料中粗蛋白的含量。胡桂萍等[13]以提取茶多酚后的茶渣为发酵原料,利用乳杆菌、枯草芽孢杆菌、酵母菌和米曲霉菌进行常温(25~35 ℃)的厌氧固态发酵5~7 d,测定发现发酵产品中粗蛋白含量达29.49%。倪星虹[14]以混合菌种经温度28 ℃发酵7 d后,测定发现茶渣发酵产物中蛋白质含量提高了60.78%。朱飞等[1]利用黑曲霉在添加5%玉米粉的茶渣中进行固态发酵,经自然pH、37 ℃、8 d条件下发酵后,茶渣的营养价值显著提高。贝莱斯芽孢杆菌(Bacillus velezensis)是2005年由Ruiz-Garcia等[15]新命名的一种生防菌,是芽孢杆菌属的一个新种[16-19]。国内外已有学者研究表明Bacillus veiezensis能产生具有广谱抗菌活性的次生代谢产物,包括纤维素酶、蛋白酶以及多种抗菌的活性物质,是用来增加作物产量、维护生态环境和农业生态系统的首选生物药剂[20-26]。贝莱斯芽孢杆也作为水产养殖的益生菌[27]。 Liu X Y等[28]对从海洋微生物中筛选到菌株Bacillus velezensis H3的发酵培养基和拮抗物质进行研究,发现该菌株的活性物质是一种替代性的表面活性素,有较高的研究价值。【本研究切入点】茶渣经发酵后虽然能在一定程度上提高茶渣的营养价值,但发酵温度均不超过37 ℃,发酵效率低,时间长,易被杂菌污染。因此有必要筛选能适合高效分解茶渣的高温菌株。但鲜见贝莱斯芽孢杆菌(Bacillus velezensis)在降解工农业副产物的研究报道。【拟解决的关键问题】本研究从堆积废弃茶渣中分离获得既耐高温又能产生高酶活的贝莱斯芽孢杆菌Fb,对其进行形态学、生理生化以及分子生物学鉴定,同时研究其最适的生长条件,分解茶渣的效果,为后续的茶渣开发研究奠定理论的基础。
1. 材料与方法
1.1 试验材料
LB培养液(L):酵母粉10 g,蛋白胨10 g,牛肉浸膏5 g,NaCl 10 g,pH 5.5~6.0。
纯化固体培养基:LB培养液中时加入20 g琼脂粉。
分离固体培养基:取新鲜茶渣50 g加蒸馏水500 ml蒸煮30 min,过滤后的茶渣液定容到200 ml,加入4 g琼脂粉。
液体发酵培养基:同LB培养液。
茶渣:采集于某地的茶饮料车间。将发酵前后的茶渣烘干,粉碎并过0.25 mm孔径筛,用四分法取样100 g。
主要仪器:LRH-250A生化培养箱,CRY-200恒温摇床,Multiskan MK3酶标仪,FOSS全自动凯氏定氮仪Kjeltec8400,FOSS2010纤维测定仪,menbarPureA300全自动氨基酸分析仪。
试剂:酵母粉、蛋白胨、NaCl等均为AR级、茚三酮染色剂、缓冲液、氨基酸标准液和稀释液均为德国menbarPure公司。
1.2 试验方法
1.2.1 菌株的培养与分离
从某地多年堆积的茶渣处取样品10 g,加入盛有50 mL LB培养液的250 mL锥形瓶中,在42 ℃、120 r·min−1条件下富集培养24 h,取2 mL培养物接种到新鲜的LB培养液,以相同的条件重复培养3次。富集培养物在分离固体培养基上划线培养,42 ℃培养24 h。挑取生长明显的菌落进行复筛。将初筛菌落在纯化固体培养基上划线分离3次,以获得纯培养物,并依据菌种在分离固体培养基的生长情况,挑取有不同形态特征的单菌落,于42 ℃ LB培养基上分别扩繁后加20%甘油混匀,于−80 ℃条件下保存。
1.2.2 菌株产酶能力测定
将菌株活化24 h后挑取1环至液体发酵培养液中,42 ℃、120 r·min−1条件下培养48 h后,测定发酵液的蛋白酶、纤维素酶的活力。采用蒽酮比色法测定纤维素酶(CL)催化羧甲基纤维素钠降解产生的还原糖的含量[29]。每mL样本每分钟催化产生1 μg葡萄糖定义为一个酶活力单位(U)。酸性蛋白酶(ACP)、中性蛋白酶(NP)和碱性蛋白酶(AKP)的测定按照GB/T 23527—2009和相关文献[30-31]执行。
1.2.3 菌落与菌体形态特征观察
菌株平板划线42 ℃分别培养16 h和25 h,在自然光下观察菌落形态;革兰氏染色后,观察菌株形态[32-33]。
1.2.4 菌株生理生化特性
1.2.5 分子鉴定
菌株的分子鉴定用16S rDNA和gyrB基因进行[36]。利用NCBI网站的BLAST功能对所测的16S rDNA和gyrA序列进行同源性分析,确定亲缘关系,使用MEGA 5.0软件Neighbor-Joining[37-38]构建系统发育树,进行1 000次的相似度重复计算。
1.2.6 菌株生长特性
测量菌株的生长曲线,判断最适生长温度[39];对其生长条件进行单因素试验如表1方案,分析不同因素对菌株生长的影响,其他培养条件和培养基相同,间隔2 h取样用酶标仪在波长630 nm测吸光度。每组设3个重复。
表 1 菌株生长条件单因素试验Table 1. Single factor test on growth conditions of bacteria因素
Factors添加水平
Level培养温度
Incubation temperature/℃30、33、36、39、42、45、48、51、54、57、60 装液量
Liquid loading quantity/mL10、15、20、25、30、35、40、45、50、55、60 摇床转速
Shaking speed/r·min−1100、120、140、160、180、200 培养时间
Fermentation time/h2、4、6、8、10、12、14、16、18、20 初始pH值
The initial pH4.0、4.5、5.0、5.5、6.0、6.5、7.0、7.5、8.0、8.5、9.0 盐度
Salinity/%0、0.5、1.0、1.5、2.0、2.5、3.0、3.5、4.0、4.5、5.0、5.5、6.0、6.5、7.0 注:盐度是指海水中溶解物质质量与海水质量的比值。
Note: Salinity refers to the ratio of dissolved substances in water to water.1.2.7 茶渣降解分析
用回接方法,验证菌株对茶渣降解效果:对照组不添加菌剂;发酵组用LB菌株培养液进行扩大培养制成种子,将培养好的种子接入新鲜茶渣中,搅拌均匀使其菌浓度约为1×106 CFU·g−1;对照组和发酵组各装入9个1 L的三角瓶,每瓶装量200 mg。两组同时放入42~45 ℃下的培养箱中;每天摇动三角瓶5 s,发酵7 d结束测定茶渣营养成分的变化。
常规成分测定[40]:粗蛋白、粗纤维和粗灰分分别按GB/T 6432—1994、GB/T 6433—2006和GB/T 6438—2007规定的方法执行。纤维的成分测定[40]:中性洗涤纤维(NDF)、酸性洗涤纤维(ADF)和酸性洗涤木质素(ADL)测定根据GB/T 20806—2006和GB/T 20805—2006方法。氨基酸测定[41]:按GB/T 18246—2000方法,采用全自动氨基酸分析仪(menbarPureA300)进行测定。
1.2.8 数据分析
所得数据用SPSS 16.0软件进行分析,F检验分析;采用氨基酸分析仪自带软件处理数据。
2. 结果与分析
2.1 菌株产酶能力比较
对5株菌的发酵液进行分析,产纤维素酶(CL)活力由高到低分别是DJ、8106、Fb、8116、HLH;酸性蛋白酶(ACP)活力由高到低分别是8106、8116、DJ、HLH、Fb;中性蛋白酶(NP)活力由高到低分别是8106、HLH、8116、DJ、Fb;碱性蛋白酶(AKP)活力由高到低分别是Fb、8116、8106、DJ、HLH(表2)。可见Fb菌产四种酶的活力均较强。
表 2 菌株酶活力比较Table 2. Enzyme activities of selected strains菌号
The
fungusCL活力
(U)
Cellulose
enzymeACP活性
(U)
Acid protease
activityNP活性
(U)
Neutral protease
activityAKP活性
(U)
Alkaline protease
activityDJ 191.58 21.59 40.23 23.35 Fb 198.33 22.90 41.24 44.36 8116 178.46 23.08 28.25 33.06 8106 189.95 24.44 30.90 27.85 HLH 154.34 16.61 54.17 18.76 2.2 菌落与菌体形态特征
Fb菌在自然光下,LB培养基,42 ℃培养16 h,其菌落浅黄色,圆形,表面干燥,不透明,边缘不整齐,如图1。菌体呈杆状,(0.4~0.6) μm×(0.9~4.0) μm,单个或成对排列,菌体形态如图2;芽胞近圆形,近中部生见图3圆圈标记。
2.3 菌株生理生化特性
由表3可知,Fb菌株能水解七叶苷;吲哚试验阳性;可在β-木糖苷酶、苯丙氨酸芳胺酶、α-半乳糖苷酶、丙氨酸-苯丙氨酸-脯氨酸芳胺酶、L-吡咯烷酮芳胺酶等酶中生长;能利用D-甘露醇、D-甘露糖、古老糖、D-海藻糖、D-葡萄糖、D-核糖等多种糖作为碳源生长;但不能利用肌醇、L-鼠李糖、菊粉、环糊精、N-乙酰-D-氨基葡萄糖、D-塔格糖、糖原、麦芽三糖、D-松三糖等。Fb菌株能在低剂量的抗生素中生长,不能在高剂量的卡那霉素耐药(0.2 g·L−1)、竹桃霉素耐药(0.1 g·L−1)、多粘菌素B耐药(0.031 g·L−1)等培养基生长。
表 3 Fb菌株生理生化特性Table 3. Characteristics of Fb项目
Items结果
Result项目
Items结果
Result项目
Items结果
Result项目
Items结果
Resultβ-木糖苷酶
β-xylosidase+ L-赖氨酸芳胺酶
L-lysine aromatase− L-天冬氨酸芳胺酶
L-aspartate aromatase− 亮氨酸芳胺酶
Leucine aromatase− 苯丙氨酸芳胺酶
Phenylalanine aramidase+ L-脯氨酸芳胺酶
L-proline aramidase− β-半乳糖苷酶
β-galactosidase− L-吡咯烷酮芳胺酶
L-pyrrolidone aramidase+ α-半乳糖苷酶
α-Galactosidase+ 丙氨酸芳胺酶
Alanine Aramidase− 酪氨酸芳胺酶
Tyrosine Aramidase− β-N-乙酰氨基葡糖苷酶
β-N-Acetylglucosaminidase− 丙氨酸-苯丙氨酸-脯氨酸芳胺酶
Alanine-phenylalanine-proline arylaminease+ 环糊精
Cyclodextrin− D-半乳糖
D-galactose− 糖原
Glycogen− 肌醇Inositol − 甲基-α-D-吡喃葡萄糖苷酸化
Methyl-α-D-glucopyranosylation+ ELLMAN 试剂 − 甲基-D-木糖苷
Methyl-D-xylosid− α-甘露糖苷酶
α-mannosidase− 麦芽三糖
Maltotriose− 甘氨酸芳胺酶
Glycine arylaminease− D-甘露醇
D-mannitol+ D-甘露糖
D-mannose+ D-松三糖
D-matsutriose− N-乙酰-D-氨基葡萄糖
N-acetyl-D-glucosamine− 古老糖
Ancient sugar+ L-鼠李糖
L-Rhamnose− β-葡糖苷酶
β-glucosidase+ β-甘露糖苷酶
β-mannosidase-phos− 磷酰胆碱
Phorylcholine− 丙酮酸盐
Pyruvate+ α-葡萄糖苷酶+
α-glucosidase− D-塔格糖
D-tagatose− D-海藻糖
D-trehalose+ 菊粉
Inulin− D-葡萄糖
D-glucose+ D-核糖
D-ribose+ 腐胺同化
Putrescine assimilation− 吲哚
Indole+ 卡那霉素耐药
Kanamycin resistance− 竹桃霉素耐药
Oleander resistance− 七叶苷水解
Aescin hydrolysis+ 红四氮唑
Red tetrazolium− 多粘菌素B耐药
Polymyxin B resistance− 注:“+”表示阳性,“-”表示阴性;ELLMAN试剂用于检测游离的巯基(-SH)。
Note: 1: + positive; − negative. 2: ELLMAN reagent for free sulfhydryl (-SH) detection.2.4 菌株的分子鉴定
利用NCBI数据库中的Blast程序分析结果表明,Fb菌株与Bacillus siamensis KCTC 13613T(AJVF01000043)和Bacillus amyloliquefaciens DSM 7T(FN597644)菌株的同源性最高,相似性达到99.93%。采用MEGA 5.0软件,邻位链接法显示Fb菌株与相关种的16S rDNA序列系统发育树(图4)。菌株gyrB测序分析表明,序列长1 176 bp。MEGA 5.0软件分析结果表明,其与Bacillus velezensis BCRC 17467T(DQ903176)的同源性较高,相似性达到100%。同理将该菌株与其他种属明确的10株菌的gyrB基因构建系统发育树(图5)。
结合菌株形态学、生理生化特性以及16S rDNA和gyrB的序列比较分析,将Fb菌株鉴定为贝莱斯芽孢杆菌(Bacillus velezensis)。
2.5 菌株生长特征
2.5.1 Fb菌的生长曲线
从图6可以看出,随着培养温度的提高,菌株的细胞分裂加快,吸光值增加;Fb菌可在30~60 ℃下生长,其最适的生长温度为39~54 ℃。
2.5.2 装液量和摇床转速对Fb菌生长的影响
摇床转速120 r·min−1时,随着装液量的增加,菌液的吸光值升高,至装液量30 mL时,Fb菌的吸光值达最高:0.97;再提高装液量,吸光值逐渐降低(图7)。从图8可知,装液量30 mL时,随着摇床转速的提高,吸光值升高,摇床转速为120 r·min−1时,Fb菌的吸光值达最高:0.98;再提高摇床转速,吸光值逐渐降低。表明Fb菌液体培养最佳装液量和摇床转速分别为30 mL和120 r·min−1。
2.5.3 培养时间对Fb菌生长的影响
图9为Fb菌在培养温度42 ℃下的生长曲线。0~6 h是菌体适应新环境的迟缓期,细胞分裂增殖缓慢;6~16 h为生长繁殖迅速的对数生长期;16 h的菌体吸光值最高0.974;16~18 h为稳定生长期;18 h后由于自溶酶作用或有毒代谢产物积累,细胞进入衰亡期裂解菌体吸光值随之下降。表明Fb菌的最适培养时间为16 h。
2.5.4 初始pH对Fb菌生长的影响
从图10可以看出,培养基初始pH 4.0~5.0时,菌体吸光值随pH的提高而提高;培养基初始pH 5.0~7.0范围内,Fb菌生物量较高且各组间差异不显著,均高0.93;再逐渐提高培养基的初始pH培养时,菌体吸光值随之下降;表明Fb菌的培养基最适初始pH为5.0~7.0。
2.5.5 盐度对Fb菌生长的影响
从图11可以看出,培养液的盐度从0.0增加到1.0%,菌株的吸光值也从0.46增加到0.92;培养液的盐度为1.0%~6.0%,菌株的吸光值均大于0.92,无显著差异;6.0%~7.0%菌株的吸光值下降至0.81。
2.6 菌株对茶渣降解效果
从茶渣常规营养成分分析看出(表4),发酵组比对照组粗蛋白提高14.88%,水分和粗纤维分别下降42.06%和9.69%,均存在显著差异。
表 4 茶渣常规营养成分的变化(n=9)Table 4. Changes in nutritional composition of tea dregs with Fb inoculation (dry base, n=9)(单位:%) 项目 Items 水分 Moisture 粗蛋白 Crude protein 粗纤维 Crude fiber 灰分 Ash 对照组 Control group 63.12±1.96 a 18.42±1.13 b 17.75±1.09 a 5.21±0.79 a 发酵组 Fermentation group 36.57±1.72 b 21.16±1.23 a 16.03±1.43 b 5.17±0.51 a 注:粗蛋白、粗纤维和灰分为干基所测值;同列数据后相同字母表示差异不显著(P>0.05),相邻或相隔字母分别表示差异显著(P<0.05)或差异极显著(P<0.01)。下表同。
Note: Crude protein, crude fiber and ash are measured on a dry basis;the same letter in the same column indicates that the difference is not significant(P>0.05), and the adjacent or spaced letters indicate that the difference is significant(P<0.05)or extremely significant(P<0.01). The same as follows.从茶渣纤维成分分析发现(表5),发酵组比对照组的中性洗涤纤维、酸性洗涤纤维和木质素分别下降10.72%、4.47%和11.37%,均存在显著差异。
表 5 茶渣纤维成分的比较(干基, n=9)Table 5. Change on fiber composition of tea dregs by treatment (dry base, n=9)(单位:%) 项目
Items中性洗涤纤维
Neutral detergent
fiber酸性洗涤纤维
Acid washing
fiber木质素
Lignin对照组
Control group50.75±1.62 a 44.12±1.62 a 2.11±1.62 a 发酵组
Fermentation group45.31±1.38 b 42.15±1.16 b 1.87±1.21 b 从茶渣氨基酸的组分和含量可以看出(表6),除胱氨酸、蛋氨酸和组氨酸发酵前后无差异外,两组均为谷氨酸含量最高,其次天冬氨酸;除胱氨酸、蛋氨酸和组氨酸发酵外,其他14种氨基酸含量均显著提高(P<0.05);氨基酸总量的变化与粗蛋白的变化一致,发酵后其含量提高5.98%,差异显著。
表 6 茶渣处理前后氨基酸含量的比较(干基, n=9)Table 6. Change on amino acid composition of tea dregs by treatment(单位:%) 项目
Items对照组
Control group发酵组
Fermentation group天冬氨酸 Asp 2.14±0.18 b 2.29±0.13 a 苏氨酸 Thr 1.09±0.05 b 1.14±0.03 a 丝氨酸 Ser 1.14±0.11 b 1.20±0.02 a 谷氨酸 Glu 2.76±0.12 b 2.95±0.13 a 甘氨酸 Gly 1.25±0.07 b 1.34±0.02 a 丙氨酸 Ala 1.36±0.04 b 1.43±0.05 a 胱氨酸 Cys 0.11±0.01 a 0.11±0.02 a 缬草氨酸 Val 1.38±0.03 b 1.47±0.03 a 精氨酸 Arg 1.34±0.05 b 1.41±0.06 a 甲硫(蛋)氨酸 Met 0.09±0.01 a 0.11±0.01 a 异亮氨酸 Iso 1.11±0.05 b 1.16±0.04 a 赖氨酸 Lys 1.65±0.02 b 1.77±0.06 a 亮氨酸 Leu 2.14±0.02 b 2.24±0.01 a 酪氨酸 Tyr 0.85±0.02 b 0.91±0.03 a 苯丙氨酸 Phe 1.33±0.03 b 1.38±0.01 a 组氨酸 His 0.05±0.01 a 0.05±0.01 a 脯氨酸 Pro 1.11±0.04 b 1.20±0.03 a 总量 Total 20.92 b 22.17 a 以上结果表明Fb菌能高效分解茶渣,显著提高茶渣的营养价值,更好实现茶渣饲料资源化利用。
3. 讨论与结论
研究表明,在固态发酵中,适宜的水分和原料粒度能够保证发酵基质深层微生物对氧气的需求,疏松多孔的基质能促进微生物的代谢废物及时排出,基质内部的养分也可随着自由水通过原料间隙扩散到基质表面,满足基质表面微生物对营养的需求,有利于微生物正常繁殖并保持高产酶活性状态[1, 42]。对于含水率高的物料,一般选择吸水性强的铺料如麦麸等来降低物料的含水率;或选择中高温菌发酵利于物料中水分蒸发。刘姝等[12]、胡桂萍等[13]、倪星虹[14]和朱飞等[1]均在茶渣中添加不同种类和比例的铺料进行发酵。本研究获得贝莱斯芽孢杆菌Fb属高温菌,在不添加任何铺料条件下,新鲜茶渣茶渣经其7 d发酵后水分下降了42.06%(P<0.05),可有效解决水分过高的问题。这种利用高温菌发酵分解高水分茶渣的方法不仅能降低生产成本,而且能发酵产生的生物热加大物料水分蒸发鲜见报道。
本研究中,利用高温特殊生境分离方法,从废弃茶渣中分离到有分解茶渣作用的高温菌5株,其中Fb菌能产生具有较强酶活力的纤维素酶、酸性蛋白酶、中性蛋白酶和碱性蛋白酶;与胡宝东等[43]从酱香型大曲中分离获得的甲基营养型芽孢杆菌(即贝莱斯芽孢杆菌)FBKL1.0190相似。Fb菌经菌落形态、菌株形态、生理生化特性的研究及分子鉴定为贝莱斯芽孢杆菌(Bacillus velezensis);可耐受55 ℃的高温,最适生长条件为:42~45 ℃、pH 5.0~7.0、16 h、盐度1.0%~6.0%、装液量0.12 mL·min−1、摇床转速120 r·min−1。茶渣经Fb菌株发酵后,与对照组比,粗蛋白提高14.88%(P<0.05);除胱氨酸、蛋氨酸和组氨酸发酵外,其他14种氨基酸含量均显著提高(P<0.05),且氨基酸总量提高5.98%(P<0.05);这与朱飞等[1]的结果一致,与李芳蓉[44]和张沛[45]的结果相近。另,本研究中茶渣经Fb菌株发酵后与对照组相比粗纤维及其中性洗涤纤维、酸性洗涤纤维和木质素分别下降9.69%、10.72%、4.47%和11.37%(P<0.05),这与张楠[46]和陈奕业[47]研究的结果相似。此外,本研究分离的Fb菌株的生长温度均高于他们的发酵菌株,这不仅可减少其他杂菌生长,而且可提高发酵效率。由此可见,Fb菌能有效分解茶渣,提高茶渣的营养价值。但该菌株在发酵分解茶渣的过程中能否产生一些抗菌的活性物质或表面活性素还有待后续的研究。
-
表 1 AtrTCP定量引物
Table 1 Quantitative primers of AtrTCP
基因
Gene正向引物序列(5′-3′)
Forward primer sequences (5′-3′)反向引物序列(5′-3′)
Reverse primer sequences(5′-3′)AtrTCP1 GCTGCTACTGGTGCTTCTATT ACCCATACCCATACCCATC AtrTCP2 GATGAGCAGCAACATTCACT CACCACTTCCACCAACACT AtrTCP3 TCTTCAACAAGCCGAACC ATTTCCCACCTCAATCCC AtrTCP4 GGTTAGTTCAAACGGCAGA ACTGTCTTCCAAATCGGGT AtrTCP5 TTATCCATACCCGAACAGC TTTAGTGTGACGGTCTTTGG AtrTCP6 TTGCTACTGGTAGAGGTGGT CCGGACCCATGTAACATTGA AtrTCP7 AACCTCCACCAAAGACCGT GTTTCTCCATCTGACTTATGCC AtrTCP8 GCCTACAGAAACAGTAACCCTC CCCGTTCAAGAAGCCATT AtrTCP9 TCCACCAAGGTCATCATCTT ATCATCGTCTCTCCGTCCA AtrTCP10 CGCAGAATCAAGAACAAGC TGACTTTGGTATGGCGGT AtrTCP11 TGCTACAGGAACCGGGACTATT GATGGCTTGGATCACTCAACTG AtrTCP12 TGGAGGCAAAGATAGACATAGC TAATGGGCAAAGGAGGGA AtrTCP13 CAACCATCATCGTCCCAA AGTTCCACTTTGCGGCTGT AtrTCP14 ACCACAGCCACAACAAAGT GCGGATACTGAGTCAAAGGA EF1a GGGATGCTGGTATGGTGAA ACGGGTCATTTCTTCTTCTGAG 表 2 AtrTCP基因家族理化性质分析
Table 2 Physicochemical properties of AtrTCP family
基因 ID
Gene ID基因名称
Gene
name蛋白质长度
Protein
length/aa分子式
Formula理化性质
Physicochemical properties相对分子量
Relative molecular
weight/kD等电点
Theoretical
pI不稳定系数
Instability
index平均亲水系数
Grand average of
hydropathicityCL11166.Contig2_All AtrTCP1 377 C1747H2777N535O570S14 40.84 6.61 50.76 −0.712 CL1181.Contig4_All AtrTCP2 423 C1950H3008N608O636S6 45.34 6.64 54.40 −0.805 CL4511.Contig1_All AtrTCP3 326 C1520H2432N462O495S6 35.29 9.17 50.51 −0.540 CL531.Contig1_All AtrTCP4 514 C2309H3640N736O795S14 54.88 8.57 43.25 −0.823 CL7519.Contig2_All AtrTCP5 231 C1096H1711N341O342S9 25.43 9.00 49.08 −0.764 CL7919.Contig2_All AtrTCP6 355 C1671H2575N519O543S9 38.91 6.79 40.47 −0.860 CL9396.Contig1_All AtrTCP7 430 C1951H3036N616O654S9 45.87 6.66 47.14 −0.747 Unigene1202_All AtrTCP8 345 C1544H2522N462O518S9 36.13 9.30 47.05 −0.408 Unigene22459_All AtrTCP9 299 C1373H2214N422O444S4 31.87 9.45 56.13 −0.563 Unigene27038_All AtrTCP10 231 C1115H1771N317O340S6 25.25 9.23 63.06 −0.492 Unigene27184_All AtrTCP11 347 C1606H2483N485O523S10 37.27 8.49 38.62 −0.743 Unigene35431_All AtrTCP12 230 C1118H1787N329O364S4 25.79 8.56 44.38 −0.820 Unigene6554_All AtrTCP13 431 C1955H3042N618O656S9 45.99 6.66 47.05 −0.754 Unigene73441_All AtrTCP14 721 C3395H5434N1006O1105S17 78.57 6.15 58.04 −0.602 表 3 AtrTCP基因家族蛋白质二级结构及亚细胞定位分析
Table 3 Protein secondary structures and subcellular localizations of AtrTCP family
转录因子
Transcription
factorTCP结构域
TCP domain蛋白质二级结构
Secondary protein structure亚细胞定位
Subcellulav
localizationα-螺旋
Alpha helix/%β-转角
Beta turn/%延伸链
Extended strand/%无规卷曲
Random coil/%AtrTCP1 111-377 19.36 5.04 13.79 61.80 Nucleus. AtrTCP2 43-178 14.42 2.36 11.58 71.63 Nucleus. AtrTCP3 86-326 24.23 5.52 15.34 54.91 Nucleus. AtrTCP4 95-253 14.20 2.53 15.18 68.09 Nucleus. AtrTCP5 57-228 13.85 9.52 16.45 60.17 Nucleus. AtrTCP6 55-223 14.08 1.69 14.65 69.58 Nucleus. AtrTCP7 88-290 20.70 4.19 10.23 64.88 Nucleus. AtrTCP8 65-176 15.65 5.22 10.43 68.70 Nucleus. AtrTCP9 51-258 19.73 6.02 18.06 56.19 Nucleus. AtrTCP10 91-231 19.05 4.76 13.42 62.77 Nucleus. AtrTCP11 69-256 11.82 10.95 21.33 55.91 Nucleus. AtrTCP12 54-215 22.61 5.22 13.48 58.70 Nucleus. AtrTCP13 88-291 14.39 5.10 11.37 69.14 Nucleus. AtrTCP14 51-332 20.11 5.83 16.37 57.70 Nucleus. 表 4 苋菜AtrTCP基因家族miRNA的预测及差异表达分析
Table 4 Prediction and differential expressions of AtrTCP family miRNA
基因名称
Gene name对应
miRNA期望值
Expectation抑制
Inhibition多样性
Multiplicity表达量
Expression quantity表达趋势
Express trendAtrTCP6 miR319_1 1.5 切割 Cleavage 1 0.764797327 上调 Up miR319_2 1.5 切割 Cleavage 1 −0.396055889 下调 Down miR319a-3p 1.5 切割 Cleavage 1 −0.134714289 下调 Down miR319a_1 1.5 切割 Cleavage 1 0.179984679 上调 Up miR319a 2.5 切割 Cleavage 1 −0.06725778 下调 Down miR319c_3 2.5 切割 Cleavage 1 0.137854295 上调 Up miR159a_1 3.0 切割 Cleavage 1 0.073891584 上调 Up Total miRNA — — ― 0.558499927 上调 Up AtrTCP2 miR319_1 3.0 切割 Cleavage 1 0.764797327 上调 Up miR319_2 3.0 切割 Cleavage 1 −0.396055889 下调 Down miR319a-3p 3.0 切割 Cleavage 1 −0.134714289 下调 Down miR319a_1 3.0 切割 Cleavage 1 0.179984679 上调 Up Total miRNA — — — 0.414011828 上调 Up AtrTCP11 miR5658 3.0 切割 Cleavage 1 −0.227576991 下调 Down AtrTCP13 miR5658 3.0 切割 Cleavage 1 −0.227576991 下调 Down “Total miRNA”表示基因对应miRNA的总表达趋势;“—”表示无结果。
"Total miRNA" represents the Total expression trend of miRNA corresponding to the gene. "—" indicates no result. -
[1] 仝亚楠, 刘雪, 刘秀洁, 等. 苋菜原生质体和瞬时转化体系的建立及应用 [J]. 分子植物育种, 2021, 19(19):6476−6481. DOI: 10.13271/j.mpb.019.006476 TONG Y N, LIU X, LIU X J, et al. Establishment and application of protoplast and transient transformation system of edible amaranth (Amaranthus spp. ) [J]. Molecular Plant Breeding, 2021, 19(19): 6476−6481.(in Chinese) DOI: 10.13271/j.mpb.019.006476
[2] STRACK D, VOGT T, SCHLIEMANN W. Recent advances in betalain research [J]. Phytochemistry, 2003, 62(3): 247−269. DOI: 10.1016/S0031-9422(02)00564-2
[3] 柳燕, 谢礼洋, 赖钟雄, 等. 苋菜amaAG基因克隆与生物信息学分析 [J]. 江西农业大学学报, 2017, 39(1):168−174. DOI: 10.13836/j.jjau.2017022 LIU Y, XIE L Y, LAI Z X, et al. Cloning and bioinformatics analysis of ama AG in Amaranthus tricolor L [J]. Acta Agriculturae Universitatis Jiangxiensis, 2017, 39(1): 168−174.(in Chinese) DOI: 10.13836/j.jjau.2017022
[4] CUBAS P, LAUTER N, DOEBLEY J, et al. The TCP domain: A motif found in proteins regulating plant growth and development [J]. The Plant Journal, 1999, 18(2): 215−222. DOI: 10.1046/j.1365-313X.1999.00444.x
[5] HUO Y Z, XIONG W D, SU K L, et al. Genome-wide analysis of the TCP gene family in switchgrass (Panicum virgatum L.)[J]. International Journal of Genomics, 2019: https://doaj.org/article/613044632d6d4e2f9f696c52793c0ebd.
[6] PARK H C, KIM M L, KANG Y H, et al. Pathogen- and NaCl-induced expression of the SCaM-4 promoter is mediated in part by a GT-1 box that interacts with a GT-1-like transcription factor [J]. Plant Physiology, 2004, 135(4): 2150−2161. DOI: 10.1104/pp.104.041442
[7] 李佳皓, 谢敏秋, 万传银, 等. 马铃薯转录因子StTCP13基因的原核表达及盐胁迫功能研究 [J]. 华北农学报, 2021, 36(2):33−39. DOI: 10.7668/hbnxb.20191507 LI J H, XIE M Q, WAN C Y, et al. Prokaryotic expression of transcription factor StTCP13 in potato and its function of salt stress [J]. Acta Agriculturae Boreali-Sinica, 2021, 36(2): 33−39.(in Chinese) DOI: 10.7668/hbnxb.20191507
[8] FANG Y J, ZHENG Y Q, LU W, et al. Roles of miR319-regulated TCPs in plant development and response to abiotic stress [J]. The Crop Journal, 2021, 9(1): 17−28. DOI: 10.1016/j.cj.2020.07.007
[9] 郑玲, 白雪婷, 李会云. 高粱TCP基因家族全基因组鉴定及表达分析 [J]. 河南农业科学, 2019, 48(10):30−36. ZHENG L, BAI X T, LI H Y. Genome-wide identification and expression analysis of TCP gene family in Sorghum bicolor L [J]. Journal of Henan Agricultural Sciences, 2019, 48(10): 30−36.(in Chinese)
[10] 李旭娟, 林秀琴, 刘洪博, 等. 甘蔗TB1基因的克隆与生物信息学分析 [J]. 热带作物学报, 2015, 36(11):1978−1985. DOI: 10.3969/j.issn.1000-2561.2015.11.010 LI X J, LIN X Q, LIU H B, et al. Cloning and bioinformatics analysis of the TB1 gene in sugarcane [J]. Chinese Journal of Tropical Crops, 2015, 36(11): 1978−1985.(in Chinese) DOI: 10.3969/j.issn.1000-2561.2015.11.010
[11] 任丽. 白桦BpTCP3基因的功能研究[D]. 哈尔滨: 东北林业大学, 2019. REN L. Function analysis of BpTCP3 gene in Betula platyphylla[D]. Harbin: Northeast Forestry University, 2019. (in Chinese)
[12] DUAN A Q, WANG Y W, FENG K, et al. TCP family genes control leaf development and its responses to gibberellin in celery [J]. Acta Physiologiae Plantarum, 2019, 41(9): 153. DOI: 10.1007/s11738-019-2945-3
[13] RIECHMANN J L, HEARD J, MARTIN G, et al. Arabidopsis transcription factors: Genome-wide comparative analysis among eukaryotes [J]. Science, 2000, 290(5499): 2105−2110. DOI: 10.1126/science.290.5499.2105
[14] 冯建英, 李立芹, 李佳皓, 等. 马铃薯TCP家族转录因子鉴定与表达模式分析 [J]. 基因组学与应用生物学, 2021, 40(S2):2756−2764. DOI: 10.13417/j.gab.040.002756 FENG J Y, LI L Q, LI J H, et al. Identification and expression pattern analysis of TCP family transcription factors in potato [J]. Genomics and Applied Biology, 2021, 40(S2): 2756−2764.(in Chinese) DOI: 10.13417/j.gab.040.002756
[15] 杨婷, 黎成, 申佳瑜, 等. 茄子TCP基因家族全基因组的鉴定与分析[J/OL]. 生物工程学报: 1-21. [2022-07-06]. DOI: 10.13345/j. cjb. 220114. YANG T, LI C, SHEN JY, et al. Identification and analysis of the complete genome of the TCP gene family in Eggplant[J/OL]. Chinese Journal of Bioengineering: 1-21[2022-07-06]. DOI: 10.13345/j.cjb.220114. (in Chinese)
[16] 关紫微, 曹希雅, 张先文, 等. 水稻TCP家族的全基因组鉴定及表达水平分析[J/OL]. 分子植物育种, 2020: 1-18. (2020-12-29). https://kns.cnki.net/kcms/detail/46.1068.S.20201229.0941.002.html. GUAN Z W, CAO X Y, ZHANG X W, et al. Genome-wide identification and expression analysis of TCP gene family in rice(Oryza sativa L. )[J/OL]. Molecular Plant Breeding, 2020: 1-18. (2020-12-29). https://kns.cnki.net/kcms/detail/46.1068.S.20201229.0941.002.html.(in Chinese)
[17] 王景超, 张君, 齐云, 等. 玉米TCP家族基因的表达分析 [J]. 玉米科学, 2022, 30(1):63−68. DOI: 10.13597/j.cnki.maize.science.20220109 WANG J C, ZHANG J, QI Y, et al. Expression analysis on TCP family genes of maize [J]. Journal of Maize Sciences, 2022, 30(1): 63−68.(in Chinese) DOI: 10.13597/j.cnki.maize.science.20220109
[18] 陈何, 王乐, 赵春丽, 等. 氮素和红蓝复合光配比对苋菜幼苗亚硝酸还原酶活性及其基因表达的影响 [J]. 中国农业大学学报, 2021, 26(8):61−71. DOI: 10.11841/j.issn.1007-4333.2021.08.07 CHEN H, WANG L, ZHAO C L, et al. Effects of nitrogen and red and blue light on NiR enzyme activity and gene expression in Amaranthus tricolor L. seedlings [J]. Journal of China Agricultural University, 2021, 26(8): 61−71.(in Chinese) DOI: 10.11841/j.issn.1007-4333.2021.08.07
[19] 王乐, 陈何, 陈家兰, 等. 基于苋菜转录组的ARF基因家族鉴定及表达 [J]. 应用与环境生物学报, 2021, 27(1):167−176. WANG L, CHEN H, CHEN J L, et al. Identification and expression of ARF gene family based on Amaranthus tricolor L. transcriptome [J]. Chinese Journal of Applied and Environmental Biology, 2021, 27(1): 167−176.(in Chinese)
[20] 孙菡笛, 薛江芝, 刘亚洁, 等. 玉米TCP转录因子家族的全基因组鉴定及表达模式分析 [J]. 分子植物育种, 2021, 19(8):2460−2471. DOI: 10.13271/j.mpb.019.002460 SUN H D, XUE J Z, LIU Y J, et al. Genome identification and expression pattern analysis of TCP transcription factor family in maize [J]. Molecular Plant Breeding, 2021, 19(8): 2460−2471.(in Chinese) DOI: 10.13271/j.mpb.019.002460
[21] 丁宁, 李梓彰, 田文, 等. 小麦TCP基因家族的全基因组鉴定和对热胁迫的响应 [J]. 麦类作物学报, 2018, 38(10):1157−1165. DOI: 10.7606/j.issn.1009-1041.2018.10.03 DING N, LI Z Z, TIAN W, et al. Genome-wide identification of the wheat TCP gene family and its response to heat stress [J]. Journal of Triticeae Crops, 2018, 38(10): 1157−1165.(in Chinese) DOI: 10.7606/j.issn.1009-1041.2018.10.03
[22] YIN Z J, LI Y, ZHU W D, et al. Identification, characterization, and expression patterns of TCP genes and microRNA319 in cotton [J]. International Journal of Molecular Sciences, 2018, 19(11): 3655. DOI: 10.3390/ijms19113655
[23] 白永宏, 赵赞延, 魏雄博, 等. 谷子TCP转录因子家族成员鉴定与生物信息学分析 [J]. 南方农业学报, 2020, 51(6):1300−1307. BAI Y H, ZHAO Z Y, WEI X B, et al. Identification and bioinformatics analysis of TCP transcription factor family member in Setaria italic L. (foxtail millet) [J]. Journal of Southern Agriculture, 2020, 51(6): 1300−1307.(in Chinese)
[24] 冯志娟, 徐盛春, 刘娜, 等. 植物TCP转录因子的作用机理及其应用研究进展 [J]. 植物遗传资源学报, 2018, 19(1):112−121. FENG Z J, XU S C, LIU N, et al. Molecular mechanisms and applications of TCP transcription factors in plants [J]. Journal of Plant Genetic Resources, 2018, 19(1): 112−121.(in Chinese)
[25] 张延召, 罗新, 李会云, 等. 牡丹TCP家族基因的鉴定与生物信息学分析 [J]. 分子植物育种, 2022, 20(1):31−37. DOI: 10.13271/j.mpb.020.000031 ZHANG Y Z, LUO X, LI H Y, et al. Identification and bioinformatics analysis of TCP family genes in tree peony [J]. Molecular Plant Breeding, 2022, 20(1): 31−37.(in Chinese) DOI: 10.13271/j.mpb.020.000031
[26] HERVÉ C, DABOS P, BARDET C, et al. In vivo interference with AtTCP20 function induces severe plant growth alterations and deregulates the expression of many genes important for development [J]. Plant Physiology, 2009, 149(3): 1462−1477. DOI: 10.1104/pp.108.126136
[27] 何志敏. 拟南芥转录因子TCP2与CRY1相互作用的遗传学与生化分析[D]. 长沙: 湖南大学, 2016. HE Z M. The molecular genetical and biochemical study of transcription factor TCP2 and CRY1 interaction in Arabidopsis[D]. Changsha: Hunan University, 2016. (in Chinese)
[28] 安新艳, 楼盼盼, 郝娟. 植物TCP转录因子的研究进展 [J]. 安徽农业科学, 2020, 48(15):20−23,27. DOI: 10.3969/j.issn.0517-6611.2020.15.006 AN X Y, LOU P P, HAO J. Research progress on plant TCP transcription factors [J]. Journal of Anhui Agricultural Sciences, 2020, 48(15): 20−23,27.(in Chinese) DOI: 10.3969/j.issn.0517-6611.2020.15.006
[29] 安家兴. 拟南芥TCP11调控维管束的发育[D]. 兰州: 兰州大学, 2012. AN J X. TCP11Regalates the development of vascular bundles in arahidopsis thaliana[D]. Lanzhou: Lanzhou University, 2012. (in Chinese)
[30] KOROLEVA O A, TOMLINSON M L, LEADER D, et al. High-throughput protein localization in Arabidopsis using Agrobacterium-mediated transient expression of GFP-ORF fusions [J]. The Plant Journal:for Cell and Molecular Biology, 2005, 41(1): 162−174.
[31] 张松莲, 曾富华, 喻宁华, 等. 植物miRNA的功能及其作用机制 [J]. 热带亚热带植物学报, 2006, 14(5):444−450. DOI: 10.3969/j.issn.1005-3395.2006.05.015 ZHANG S L, ZENG F H, YU N H, et al. Advances in studies on the function and mechanism of plant microRNA [J]. Journal of Tropical and Subtropical Botany, 2006, 14(5): 444−450.(in Chinese) DOI: 10.3969/j.issn.1005-3395.2006.05.015
[32] 李坤杰, 谭杉杉, 孙勃, 等. 芥菜TCP转录因子家族全基因组鉴定及表达分析 [J]. 四川农业大学学报, 2019, 37(4):459−468. LI K J, TAN S S, SUN B, et al. Genome-wide identification and analysis of TCP transcription factor family in Brassica juncea [J]. Journal of Sichuan Agricultural University, 2019, 37(4): 459−468.(in Chinese)
[33] LIN Y F, CHEN Y Y, HSIAO Y Y, et al. Genome-wide identification and characterization of TCP genes involved in ovule development of Phalaenopsis equestris [J]. Journal of Experimental Botany, 2016, 67(17): 5051−5066. DOI: 10.1093/jxb/erw273
[34] KOYAMA T, SATO F, OHME-TAKAGI M. Roles of miR319 and TCP transcription factors in leaf development [J]. Plant Physiology, 2017, 175(2): 874−885. DOI: 10.1104/pp.17.00732
[35] LIU S C, ZHENG X L, PAN J F, et al. RNA-sequencing analysis reveals betalains metabolism in the leaf of Amaranthus tricolor L [J]. PLoS One, 2019, 14(4): e0216001. DOI: 10.1371/journal.pone.0216001
[36] 王廷芹, 甘秋霞, 李倩茹. 盐胁迫对苋菜种子的发芽及幼苗生长的影响 [J]. 贵州大学学报(自然科学版), 2021, 38(1):10−15,32. DOI: 10.15958/j.cnki.gdxbzrb.2021.01.02 WANG T Q, GAN Q X, LI Q R. Effect of salt stress on seed germination and seedling growth of amaranth [J]. Journal of Guizhou University (Natural Sciences), 2021, 38(1): 10−15,32.(in Chinese) DOI: 10.15958/j.cnki.gdxbzrb.2021.01.02
[37] 曾泳怡, 冯梓晴, 曾晓靖, 等. NaCl胁迫对不同基因型水稻种子萌发和幼苗生长的影响 [J]. 安徽农业科学, 2021, 49(7):25−29. DOI: 10.3969/j.issn.0517-6611.2021.07.007 ZENG Y Y, FENG Z Q, ZENG X J, et al. Effects of NaCl stress on seed germination and seedling growth of different rice genotypes [J]. Journal of Anhui Agricultural Sciences, 2021, 49(7): 25−29.(in Chinese) DOI: 10.3969/j.issn.0517-6611.2021.07.007




 下载:
下载: