Metabolism pathway of malic acid in Saccharomyces cerevisiae JP2 and analysis of its key genes
-
摘要:目的 酿酒酵母JP2(Saccharomyces cerevisiae JP2)是从自然发酵枇杷酒醪中分离得到一株具有较好代谢苹果酸的酵母菌。为深入研究其内在降酸机理,对其进行全基因组测序和基因组序列信息解析,并分析其可能的代谢苹果酸途径,找出影响降酸的基因。方法 基于Illumina Miseq和PacBio测序平台,联用第二代和第三代测序技术对酿酒酵母JP2和模式菌株酿酒酵母S288c进行全基因组测序,采用生物信息学方法对其进行序列组装、基因预测与功能注释,并挖掘JP2可能的苹果酸代谢路径。结果 (1)通过对酿酒酵母JP2基因组组装共得到52个重叠群,整个基因组大小为11.98 Mbp,GC含量 38.07%;JP2和S288c二者的全基因组大小和GC含量差异不大。(2)JP2代谢苹果酸的途径有3个:苹果酸→草酰乙酸→丙酮酸;苹果酸→丙酮酸;苹果酸→富马酸。相关的主要基因分别为maeA、MDH及fumC,其中苹果酸→草酰乙酸→丙酮酸是主要代谢路径,关键基因是maeA。(3)模拟发酵过程苹果酸代谢及其关键基因表达量变化显示,JP2的苹果酸含量下降了35.8%,下降程度显著高于S288c;与S288c相比,JP2的maeA基因呈现表达极显著上调(P<0.01),在发酵后期,其相对表达量达到起始值的130%。结论 JP2在果酒发酵过程中呈现maeA基因高效表达,从而显著提高了苹果酸降解效率。研究结果可为酿酒酵母JP2降酸功能基因的研究以及降酸工程菌的研发提供技术支撑。Abstract:Objective Saccharomyces cerevisiae JP2, isolated from naturally fermented loquat, is a yeast strain with good metabolism of malic acidwine mash. To further investigate its intrinsic acid lowering mechanism, whole genome sequencing and genome sequence information analysis were conducted, and possible metabolic pathways of malic acid were analyzed to identify genes that affect acid lowering.Method Based on the Illumina Miseq and PacBio sequencing platforms, second and third generation sequencing techniques were used to sequence the entire genome of S. cerevisiae JP2 and the type strain S. cerevisiae S288c. Bioinformatics methods were used to assemble their sequences, predict their genes, and annotate their functions, while exploring the possible malic acid metabolism pathways of JP2.Result (1) A total of 52 contigs were obtained by assembling the genome of S. cerevisiae JP2, with a total genome size of 11.98 Mbp and a GC content of 38.07%; There is no significant difference in overall genome size and GC content between JP2 and S288c. (2) There are three pathways for JP2 to metabolize malic acid: malic acid → oxaloacetate → pyruvate; malic acid → pyruvate; and malic acid → fumaric acid. The main genes involved are maeA, MDH, and fumC, among which malic acid → oxaloacetate → pyruvate is the main metabolic pathway with the key gene maeA. (3) The simulated fermentation process showed a decrease of 35.8% in malic acid metabolism and key gene expression levels in JP2, with a significant decrease compared to S288c; Compared with S288c, the maeA gene of JP2 was significantly upregulated (P<0.01), and its relative expression reached 130% of the initial value in the late fermentation stage.Conclusion JP2 exhibits high expression of maeA gene during fruit wine fermentation, significantly improving the efficiency of malic acid degradation. The research results can provide technical support for the study of the acid reducing functional gene of S. cerevisiae JP2 and the development of acid reducing engineering bacteria.
-
0. 引言
【研究意义】酵母菌是葡萄酒等果酒进行酒精发酵所必需的微生物。对于果酸含量较高的水果酿造,必须通过降酸环节来增加酒体柔和度和改善风味,进而提升果酒品质[1-2]。与物理降酸和化学降酸相比,生物降酸被认为是最为合理、有效的降酸途径[3]。目前主要的生物降酸方式是通过乳酸菌作用下的苹果酸-乳酸发酵和酵母菌作用下的有机酸代谢[4]。但是,乳酸菌的发酵特性决定了苹果酸-乳酸发酵过程的降酸幅度是有限的,往往无法满足高酸性果酒的降酸需求[5]。如果选择具有降酸能力的酵母菌用于果酒一次发酵即酒精发酵期的有机酸代谢,再通过二次发酵即苹果酸-乳酸发酵进一步代谢有机酸,即可实现“双效降酸”的效果。【前人研究进展】与乳酸菌相比,酵母菌对酒体环境中酒精、酸度等的耐受性更好[6-7];国内外早有酵母菌降酸方面的研究,但至今仍未得到实际应用。究其原因,主要有两方面:一方面受菌种特性的影响。酿酒酵母降酸能力普遍较差;裂殖酵母(Schizosaccharomyces)降酸能力虽强,但该菌酒精发酵力弱、产不良风味且生长过程受酿酒酵母的明显抑制,因此该菌的降酸过程和酿酒酵母的酒精发酵必需单独进行,工序复杂[8-10],不适宜用于果酒生物降酸。另一方面菌种选育技术还有待突破。利用诱变、原生质体融合和基因工程等对酿酒酵母的改造,虽可以使其兼有酒精发酵和果酸降解能力,但存在稳定性差和食品安全争议等问题,从而都止步于试验阶段[11-12]。本研究前期筛选到的野生型酿酒酵母JP2[13]所具备的高酒精发酵、强果酸代谢能力,成功克服了以上两方面的问题,所以具有重要的研究价值。【本研究切入点】酿酒酵母菌JP2在完成酒精发酵的同时还达到了生物降酸效果,但其降酸的途径尚不明确。【拟解决的关键问题】本研究以酿酒酵母JP2为研究对象,以模式菌株酿酒酵母S288c为对照,采用全基因组、比较基因组及代谢组学方法,解析JP2代谢苹果酸关键基因和代谢路径,旨在于明确酿酒酵母JP2代谢苹果酸过程的通路和关键基因功能,初步解析该菌降酸机制,为制定酿酒酵母生物降酸策略、实现代谢通路的可调可控提供理论指导。
1. 材料与方法
1.1 材料
1.1.1 菌株
酿酒酵母JP2,从自然发酵枇杷酒醪中分离得到,具有较强的酒精发酵能力和降酸特性[13]。模式菌株酿酒酵母S288c,购自美国标准生物品收藏中心(American type culture collection,ATCC)。
1.1.2 培养基
模拟葡萄酒培养基:100 g·L−1 葡萄糖,100 g·L−1果糖,4.5 g·L−1 L-苹果酸,2 g·L−1 (NH4)2SO4,1.7 g·L−1酵母膏,灭菌备用。
1.2 酿酒酵母JP2的培养与基因组DNA的提取
用YPD液体培养基活化酿酒酵母JP2,接种于1 L 模拟酒中,接种量≥1×107 cfu·mL−1,25 ℃下发酵,每天收集发酵过程中的酒样,6000 r·min−1离心10 min收集菌体。
采用基因组提取试剂盒(Bacterial Genomic DNA Isolation Kit,#DP302,TIANGEN)提取酿酒酵母JP2基因组。
1.3 JP2全基因组构建及苹果酸代谢路径分析
采用全基因组鸟枪法策略,构建不同插入片段的文库,利用第二代测序技术,基于Illumina Miseq测序平台,同时利用第三代单分子测序技术,基于PacBio测序平台对文库进行测序。本次测序总共构建了400 bp和20 kp 2个文库。采用AdapterRemoval(v1.5.4)去除测序数据包含的带接头、低质量的reads,以减少后续冗余信息的干扰,保证信息分析质量;采用SOAPec(v2.0)软件基于Kmer频率对所有reads进行质量校正,校正所采用的Kmer设置为17。基因预测采用Glimmer3.0软件,基因功能注释采用本地BLASTP方法将预测的基因序列与KEGG数据库进行比对,得到相应的注释信息。
1.4 酿酒酵母JP2降苹果酸关键基因筛选
将JP2与S288c的蛋白质序列合并建立数据库,同时合并的序列文件作为Query进行All-VS-All BlastP分析。采用orthomcl进行筛选,序列比对的长度不低于任意两条序列的长度的70%,最后对筛选结果进行mcl聚类,其中inflation设置为1.5。
1.5 降酸关键基因分析
将活化后JP2与S288C分别接入模拟酒中进行发酵,收集发酵过程中的酒样。采用高效液相检测方法,检测发酵酒样中的有机酸成分。采用qRT-PCR的方法[11],对影响JP2与S288c代谢苹果酸关键基因进行定量检测,考察两株菌的目标基因表达情况,分析JP2代谢苹果酸关键基因。
1.6 检测方法
2. 结果与分析
2.1 酿酒酵母JP2全基因组构建
采用SOAPdenovo软件组装得到酿酒酵母JP2基因组大小为11982192 bp,最长片段大小为956649 bp,最短片段大小为1086 bp,GC含量38.07%,共51个支架,52个重叠群。
对组装好的序列进行组分分析,采用Augustus(version 3.03)、glimmerHMM(version 3.0.1)和SNAP(version 2006-07-28)软件共预测5205个基因(表1),总长度8044579 bp,平均长度1545 bp,占基因组全长的66.50%。用tRNAscan软件共预测出289个tRNA,用rRNAmmer1.2软件预测出4个rRNA。通过基因组基本信息的比较,发现JP2基因组比S288c小了0.18 Mbp,二者的全基因组GC含量差异不大。
表 1 基因组基本信息Table 1. Basic information of genome特征
Property酿酒酵母JP2
S. cerevisiae JP2酿酒酵母S288c
S. cerevisiae S288c序列总长
Total sequence length/Mbp11.98 12.16 GC含量
GC content/%38.07 38.15 预测基因占比
Predicted gene proportion/%66.50 75.37 预测基因数量
Predicted number of genes5205 5906 预测基因长度
Predicted Gene Length/bp1545 1369 tRNA数量
tRNA quantity289 299 rRNA数量
rRNA quantity4 10 2.2 酿酒酵母JP2苹果酸代谢路径分析
采用本地BLASTP的方法,结合KEGG数据库,对预测基因进行功能注释。
酿酒酵母JP2在KEGG数据库中共有3168个基因得到功能注释,可以分成47个亚分类图,结果见图1,发现在翻译(866个)、折叠分类降解(628个)、复制和修复(442个)得到较多的功能注释。将这些注释的基因比对到代谢通路图,最终得到346个代谢通路图。其中,与苹果酸代谢相关代谢路径共3条(图2),分别为:(1)苹果酸→草酰乙酸→丙酮酸→乙酸→乙醛→乙醇;(2)苹果酸→丙酮酸→乙酸→乙醛→乙醇;(3)苹果酸→富马酸。
2.3 酿酒酵母JP2降苹果酸关键基因解析
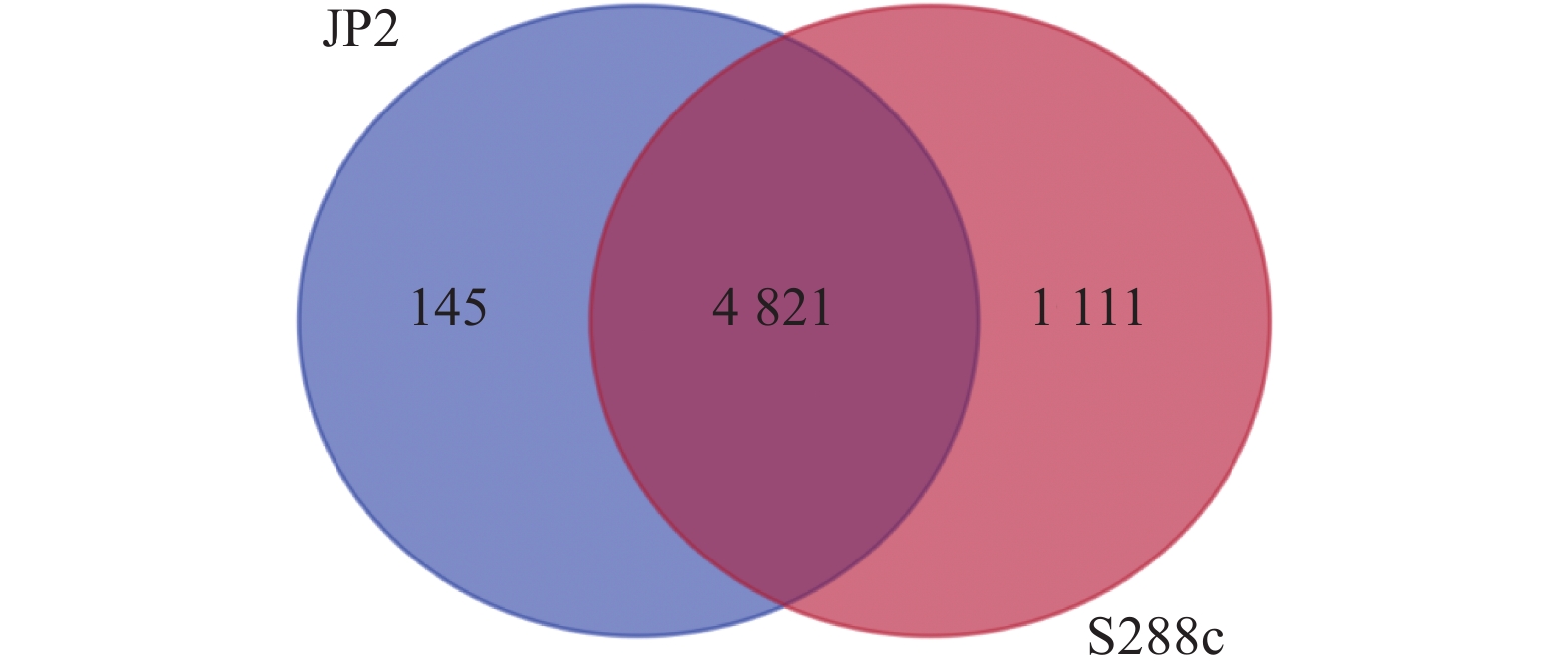
通过酿酒酵母JP2与S288C全基因组比较分析,结果表明(图3、图4),与S288c相比,JP2特有145个基因,缺失1111个基因。在缺失基因中未找到与苹果酸代谢相关的基因,在特有基因中找到与苹果酸代谢密切相关的基因有3个,分别为:(1)maeA基因。该基因是构成苹果酸脱氢酶(EC 1.1.1.37)关键基因,影响的代谢路径为苹果酸→草酰乙酸→丙酮酸。(2)MDH基因。该基因是构成苹果酸脱氢酶(EC 1.1.1.38)关键基因,虽然在苹果酸脱氢酶(EC 1.1.1.38)作用下酵母JP2代谢苹果酸时也产生草酸乙酰盐,但草酸乙酰盐只是苹果酸脱氢酶(EC 1.1.1.38)催化反应过程中的中间产物,其会迅速转化为丙酮酸,因而该酶影响的代谢路径应该为苹果酸→丙酮酸。(3)fumC基因。该基因是构成富马酸水合酶(EC 4.2.1.2)关键基因,代谢路径为苹果酸→富马酸。
2.4 酿酒酵母 JP2发酵过程中代谢苹果酸关键基因的表达变化
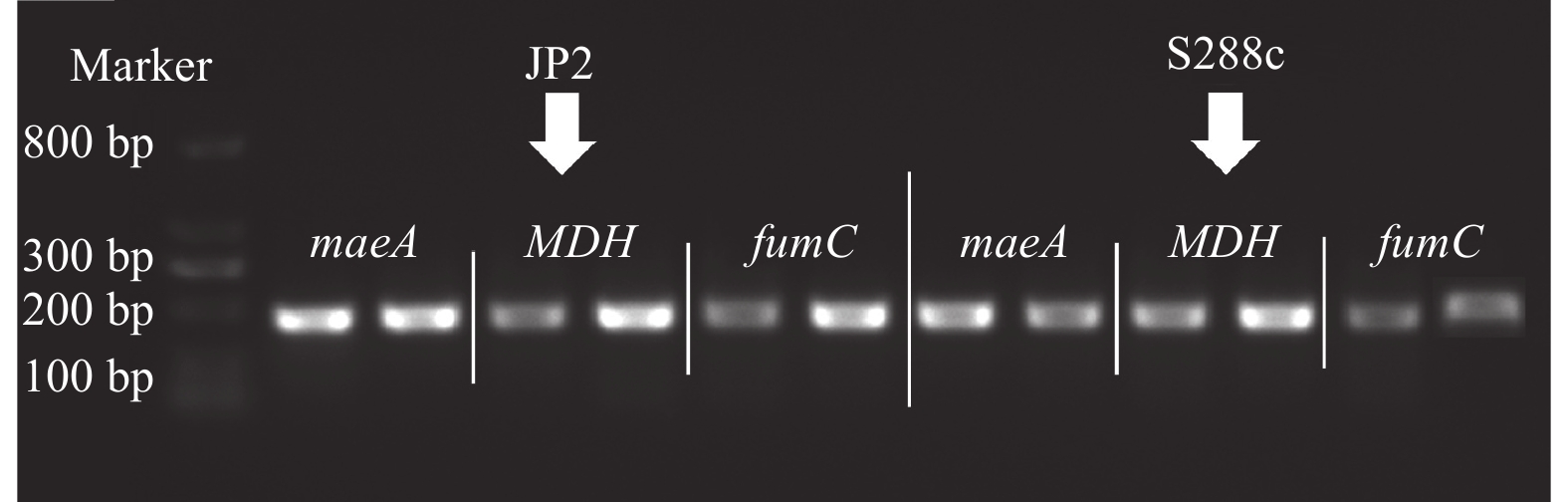
以JP2和S288c为对象,在模拟葡萄酒培养基中发酵13 d,采用高效液相技术检测发酵前后有机酸的变化,采用PCR、qRT-PCR技术对苹果酸代谢的密切相关基因进行存在和表达量研究。由表2可知,JP2试验组中的苹果酸含量下降了35.8%,而S288c试验组的苹果酸含量下降程度显著低于JP2试验组,两个试验组中的其余有机酸均呈不同程度的上升。由图5可知,3个基因在两菌中均存在,在菌株S288c中的基因表达水平弱于JP2菌株,推测为S288c同位点存在序列缺失。由表3可知,与S288c相比,JP2的maeA基因出现表达极显著上调(P<0.01),而MDH、fumC基因表达差异不显著。
表 2 酿酒酵母JP2和S288c发酵过程有机酸代谢Table 2. Organic acid metabolism during the fermentation process of S. cerevisiae JP2 and S. cerevisiae S288c (单位:μg·mL−1)有机酸
Organic acid发酵时间
Fermentation timeJP2试验组
JP2 groupS288c试验组
S288c group草酸 Oxalate 发酵起点 0.00±0.00 0.00±0.00 发酵结束 2.58±0.02 2.76±0.04 酒石酸 Tartaric acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 6.39±0.05 6.81±0.08 苹果酸 Malic acid 发酵起点 4500.00±0.00 4500.00±0.00 发酵结束 2886.87±150.21 4365.36±143.45* 乳酸 Lactic acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 44.19±3.14 47.12±4.86 乙酸 Acetic acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 556.26±25.01 213.14±18.24 富马酸 Fumaric acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 1.71±0.04 1.98±0.06 *:同一发酵时间不同试验组之间差异显著(P<0.05)。
*: Significant difference between different groups at the same fermentation time (P<0.05).表 3 苹果酸代谢关键基因表达量比较Table 3. Comparison of expression of key genes of malic acid metabolism菌株
StrainmaeA基因
maeA geneMDH基因
MDH genefumC基因
fumC geneJP2 1.25** 1.02 0.97 S288C 1.00 1.00 1.00 **:不同菌株之间差异极显著(P<0.01)。
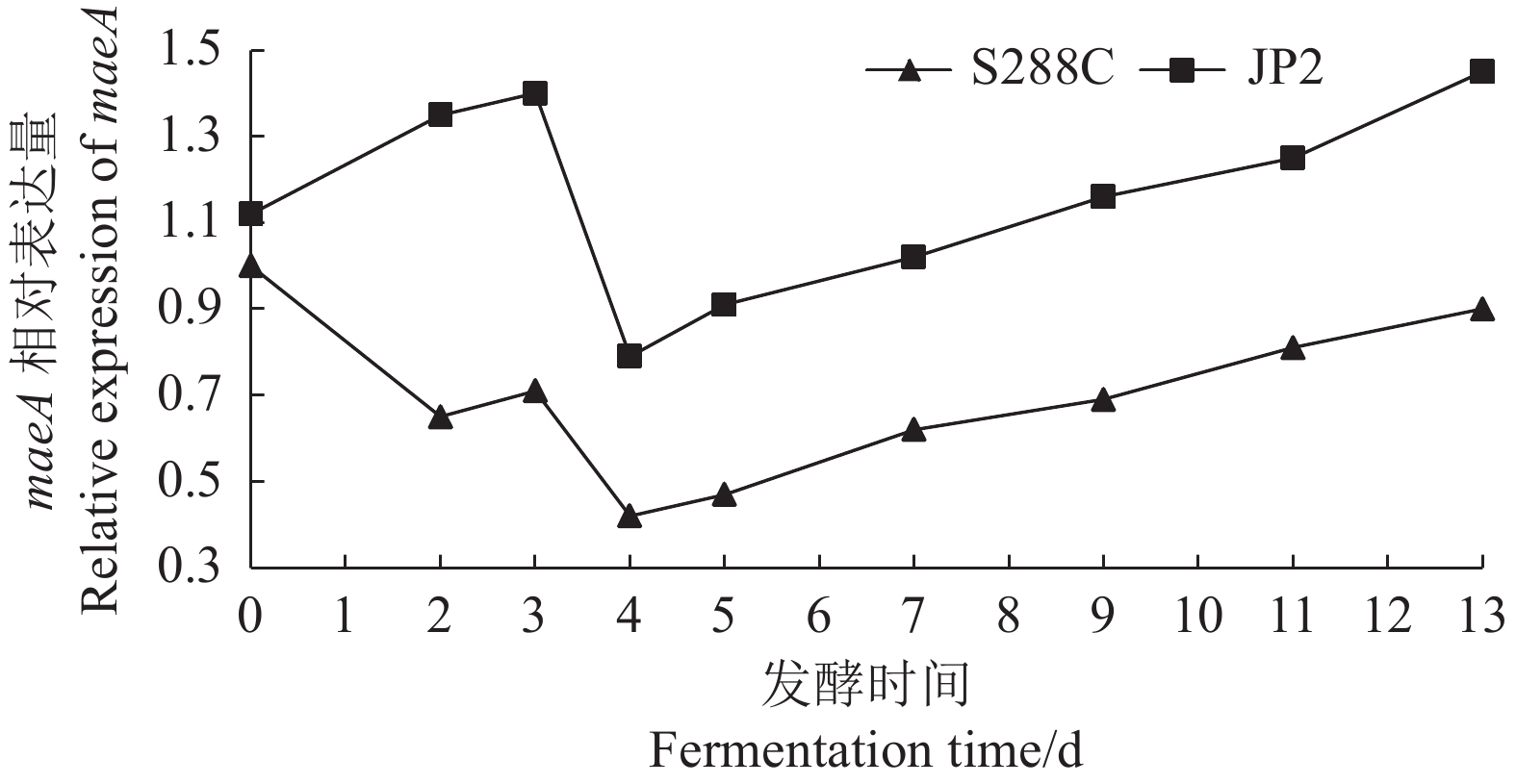
**: Significant difference between different strains (P<0.01).进一步考察两菌maeA基因在模拟发酵液中不同发酵时期的变化,结果见图6。在酒精发酵的前3 d,酿酒酵母JP2的maeA相对表达量上升,而后缓慢下降,在发酵后期,其相对表达量急速上升,达到起始值的130%。而对照酵母菌酿酒酵母S288c的maeA相对表达量先下降后期略微上升,但仍低于起始值。表明苹果酸→草酰乙酸→丙酮酸是JP2代谢苹果酸的主要途径,maeA是其关键基因。
3. 讨论
苹果酸-乙醇发酵被认为是酵母菌代谢苹果酸的主要途径,这一途径有两个关键酶即苹果酸通透酶和苹果酸酶[16]。裂殖酵母在苹果酸通透酶基因mae1表达的H+-苹果酸同向运载蛋白作用下,将胞外苹果酸主动转运进入细胞[14];该基因的表达是组成型的,不受代谢副产物的抑制,保证了苹果酸的高效转运[17]。然而,酿酒酵母没有苹果酸转运基因mae1,苹果酸靠单纯扩散的方式进入细胞质,且该过程可能受到己糖(葡萄糖、果糖)的诱导或抑制,苹果酸转运效率较低[18]。前期研究中,显示JP2主要代谢的有机酸为苹果酸,并通过高效代谢苹果酸以实现其显著降酸功能。通过基因组及代谢组分析,明确酿酒酵母JP2降解苹果酸涉及代谢过程中3个关键基因,与S288c相比,JP2的maeA基因出现表达极显著上调,而MDH、fumC基因表达差异不显著,同时JP2发酵液有机酸中苹果酸显著下降,乙酸显著增加,并生成少量的富马酸,因而代谢路径苹果酸→草酰乙酸→丙酮酸→乙酸→乙醇是JP2代谢苹果酸主要途径,关键基因为maeA;苹果酸→富马酸和苹果酸→丙酮酸→乙酸→乙醇为次要路径。与模式酿酒酵母S288c相比,JP2在果酒发酵过程中maeA基因高效表达,可显著提高苹果酸降解效率。后续研究将采用基因敲除或强表达技术,进一步对maeA基因的降酸功能进行验证。
-
表 1 基因组基本信息
Table 1 Basic information of genome
特征
Property酿酒酵母JP2
S. cerevisiae JP2酿酒酵母S288c
S. cerevisiae S288c序列总长
Total sequence length/Mbp11.98 12.16 GC含量
GC content/%38.07 38.15 预测基因占比
Predicted gene proportion/%66.50 75.37 预测基因数量
Predicted number of genes5205 5906 预测基因长度
Predicted Gene Length/bp1545 1369 tRNA数量
tRNA quantity289 299 rRNA数量
rRNA quantity4 10 表 2 酿酒酵母JP2和S288c发酵过程有机酸代谢
Table 2 Organic acid metabolism during the fermentation process of S. cerevisiae JP2 and S. cerevisiae S288c (单位:μg·mL−1)
有机酸
Organic acid发酵时间
Fermentation timeJP2试验组
JP2 groupS288c试验组
S288c group草酸 Oxalate 发酵起点 0.00±0.00 0.00±0.00 发酵结束 2.58±0.02 2.76±0.04 酒石酸 Tartaric acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 6.39±0.05 6.81±0.08 苹果酸 Malic acid 发酵起点 4500.00±0.00 4500.00±0.00 发酵结束 2886.87±150.21 4365.36±143.45* 乳酸 Lactic acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 44.19±3.14 47.12±4.86 乙酸 Acetic acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 556.26±25.01 213.14±18.24 富马酸 Fumaric acid 发酵起点 0.00±0.00 0.00±0.00 发酵结束 1.71±0.04 1.98±0.06 *:同一发酵时间不同试验组之间差异显著(P<0.05)。
*: Significant difference between different groups at the same fermentation time (P<0.05).表 3 苹果酸代谢关键基因表达量比较
Table 3 Comparison of expression of key genes of malic acid metabolism
菌株
StrainmaeA基因
maeA geneMDH基因
MDH genefumC基因
fumC geneJP2 1.25** 1.02 0.97 S288C 1.00 1.00 1.00 **:不同菌株之间差异极显著(P<0.01)。
**: Significant difference between different strains (P<0.01). -
[1] GRANDVALET C. Oenococcus oeni: Queen of the cellar, nightmare of geneticists [J]. Microbiology, 2017, 163(3): 297−299. DOI: 10.1099/mic.0.000456
[2] JIANG J, SUMBY K M, SUNDSTROM J F, et al. Directed evolution of Oenococcus oeni strains for more efficient malolactic fermentation in a multi-stressor wine environment [J]. Food Microbiology, 2018, 73: 150−159. DOI: 10.1016/j.fm.2018.01.005
[3] FERNÁNDEZ-PÉREZ R, TENORIO RODRÍGUEZ C, RUIZ-LARREA F. Fluorescence microscopy to monitor wine malolactic fermentation [J]. Food Chemistry, 2019, 274: 228−233. DOI: 10.1016/j.foodchem.2018.08.088
[4] VILELA A. Use of nonconventional yeasts for modulating wine acidity [J]. Fermentation, 2019, 5(1): 27. DOI: 10.3390/fermentation5010027
[5] 李华, 刘延琳, 蒋思欣, 等. Oenococcus oeni苹果酸-乳酸发酵关键酶基因在酿酒酵母中的转化与表达 [J]. 农业生物技术学报, 2006, 14(4):606−611. LI H, LIU Y L, JIANG S X, et al. Cloning of two key enzyme genes from Oenococcus oeni for malolactic fermentation and their expressions in Saccharomyces cerevisiae [J]. Journal of Agricultural Biotechnology, 2006, 14(4): 606−611.(in Chinese)
[6] BENITO Á, JEFFARES D, PALOMERO F, et al. Selected Schizosaccharomyces pombe strains have characteristics that are beneficial for winemaking [J]. PLoS One, 2016, 11(3): e0151102. DOI: 10.1371/journal.pone.0151102
[7] PETRUZZI L, CAPOZZI V, BERBEGAL C, et al. Microbial resources and enological significance: Opportunities and benefits [J]. Frontiers in Microbiology, 2017, 8: 995. DOI: 10.3389/fmicb.2017.00995
[8] BENITO S, PALOMERO F, CALDERÓN F, et al. Selection of appropriate Schizosaccharomyces strains for winemaking [J]. Food Microbiology, 2014, 42: 218−224. DOI: 10.1016/j.fm.2014.03.014
[9] MYLONA A E, DEL FRESNO J M, PALOMERO F, et al. Use of Schizosaccharomyces strains for wine fermentation—Effect on the wine composition and food safety [J]. International Journal of Food Microbiology, 2016, 232: 63−72. DOI: 10.1016/j.ijfoodmicro.2016.05.023
[10] MINNAAR P P, JOLLY N P, PAULSEN V, et al. Schizosaccharomyces pombe and Saccharomyces cerevisiae yeasts in sequential fermentations: Effect on phenolic acids of fermented Kei-apple (Dovyalis caffra L. ) juice [J]. International Journal of Food Microbiology, 2017, 257: 232−237. DOI: 10.1016/j.ijfoodmicro.2017.07.004
[11] VAUDANO E, COSTANTINI A, GARCIA-MORUNO E. An event-specific method for the detection and quantification of ML01, a genetically modified Saccharomyces cerevisiae wine strain, using quantitative PCR [J]. International Journal of Food Microbiology, 2016, 234: 15−23. DOI: 10.1016/j.ijfoodmicro.2016.06.017
[12] BENITO Á, CALDERÓN F, BENITO S. Schizosaccharomyces pombe biotechnological applications in winemaking [J]. Methods in Molecular Biology, 2018, 1721: 217−226.
[13] 何志刚, 李维新, 梁璋成, 等. 优良降酸酿酒酵母的分离和鉴定 [J]. 中国食品学报, 2013, 13(5):191−197. DOI: 10.16429/j.1009-7848.2013.05.028 HE Z G, LI W X, LIANG Z C, et al. Separation and identification of selected yeast with deacidification ability for brewing in fruit wine [J]. Journal of Chinese Institute of Food Science and Technology, 2013, 13(5): 191−197.(in Chinese) DOI: 10.16429/j.1009-7848.2013.05.028
[14] 李维新, 魏巍, 何志刚, 等. 酿酒酵母JP2发酵枇杷汁的有机酸代谢及其产香特征分析 [J]. 中国食品学报, 2016, 16(2):251−257. DOI: 10.16429/j.1009-7848.2016.02.034 LI W X, WEI W, HE Z G, et al. Analysis of organic acids metabolism and fragrant characteristic for fermentated loquat juice using Saccharomyces cerevisiae JP2 [J]. Journal of Chinese Institute of Food Science and Technology, 2016, 16(2): 251−257.(in Chinese) DOI: 10.16429/j.1009-7848.2016.02.034
[15] LIANG Z C, LIN X Z, HE Z G, et al. Amino acid and microbial community dynamics during the fermentation of Hong Qu glutinous rice wine [J]. Food Microbiology, 2020, 90: 103467. DOI: 10.1016/j.fm.2020.103467
[16] COMITINI F, AGARBATI A, CANONICO L, et al. Yeast interactions and molecular mechanisms in wine fermentation: A comprehensive review [J]. International Journal of Molecular Sciences, 2021, 22(14): 7754. DOI: 10.3390/ijms22147754
[17] DARBANI B, STOVICEK V, VAN DER HOEK S A, et al. Engineering energetically efficient transport of dicarboxylic acids in yeast Saccharomyces cerevisiae [J]. Proceedings of the National Academy of Sciences of the United States of America, 2019, 116(39): 19415−19420. DOI: 10.1073/pnas.1900287116
[18] BOVO B, NADAI C, VENDRAMINI C, et al. Aptitude of Saccharomyces yeasts to ferment unripe grapes harvested during cluster thinning for reducing alcohol content of wine [J]. International Journal of Food Microbiology, 2016, 236: 56−64. DOI: 10.1016/j.ijfoodmicro.2016.07.022




 下载:
下载: