Transcriptome-based EST-SSR Search for Analysis on Lemon Genetic Diversity
-
摘要:目的
以柠檬转录组数据为基础开发EST-SSR标记并进行遗传多样性分析。
方法利用MISA软件对柠檬转录组进行SSR位点搜索,用Primer 3.0设计和筛选引物,并绘制49份柠檬的聚类图。
结果共鉴定到
40 193 个SSR位点,SSR分布频率为35.67%,平均分布距离为1.39 kb;柠檬转录组中SSR序列以单、三、二核苷酸重复类型为主,分别占80.91%、9.20%和8.67%,其优势基元分别为A/T、AG/CT和AAG/CTT;共设计出4561 对引物,选取其中24对进行有效性验证,筛选到13对多态性引物;扩增得到47个等位基因,有效等位基因均值为3.615;观察杂合度、期望杂合度和引物多态信息含量的均值为0.637、0.516和0.528;在遗传距离0.40时可将供试材料划分为五大类,聚类结果与传统的形态学分类结果大体一致。结论本研究开发的13对EST-SSR引物能将供试材料进行有效区分,与传统分类学相吻合,并揭示了供试材料间具有丰富的遗传多样性,为柠檬种质资源后续开发及利用提供参考依据。
Abstract:ObjectiveEST-SSRs of lemon were searched based on transcriptome data to analyze the genetic diversity of the plant.
MethodSSR loci of lemon transcriptomes were screened using MISA to design primers by Primer 3.0. A clustering of 49 species was secured.
ResultOf the
40193 SSR loci identified, a distribution frequency of 35.67% and an average distribution distance of 1.39 kb were found in the sequences of 80.91% mono-, 9.20% tri-, and 8.67% dinucleotide repeat types with the dominant motifs of A/T, AG/CT, and AAG/CTT, respectively. Twenty-four of the4561 pairs of primers were selected for validity verification, and 13 by being polymorphic. The amplified 47 alleles had a mean effective value of 3.615 with the mean Ho, He, and PIC of 0.637, 0.516, and 0.528, respectively. At the genetic distance of 0.40, the lemon cultivars were clustered into 5 categories, which were generally consistent with that obtained by the traditional morphological classification.ConclusionThirteen pairs of EST-SSR primers were selected to classify 49 lemon specimens. The resulting clustering agreed with traditional taxonomy. The displayed rich genetic diversity rendered these cultivars an adequate germplasm collection for the development and utilization of lemon plants.
-
Keywords:
- lemon /
- transcriptome /
- EST-SSRs /
- cluster analysis
-
0. 引言
【研究意义】柠檬[Citrus limon(L.)Burm.F.]属芸香科(Rutaceae)柑橘属(Citrus)[1]枸橼类(Citrus medica L.)植物[1],起源于印度东北部的阿萨姆邦、缅甸北部及中国南方地区[2],在四川、重庆、云南、广西、海南和福建等地均有种植,已有
1000 多年的栽培史[3,4]。柠檬果形独特,有很高的营养价值与药用价值,不仅具有独特怡人的香味和酸爽口感,还具有多种生理活性功能,广泛应用于食品、美容、保健及医疗等行业[2],市场前景广阔。我国在柠檬种植业上,对传统资源开发的同时也在不断引进国外资源,已先后从美国、意大利、西班牙、澳大利亚等地引进了大量的种质进行栽培[1,3,4],极大地丰富了国内柠檬种质资源。但随着社会需求的增长、栽培面积的扩大、种质不断引进,我国现有柠檬品种或品系达200多种[1,3,4],存在植株性状相似、遗传背景不明、同物异名或同名异物等情况,极大影响了柠檬产业的推广和应用,为此柠檬种质资源鉴定评价及多样性分析等基础工作显得尤其重要。【前人研究进展】有关学者采用简单序列重复区间扩增多态性(inter-simple sequence repeat, ISSR)[5]、简单重复序列(simple sequence repeat, SSR)[6]、目标起始密码子多态性(start codon targeted polymorphism, SCoT)[5]等分子标记和表型性状[7]逐步开展柠檬遗传多样性和种质资源鉴定研究。其中SSR分子标记是检测遗传背景复杂的木本植物的有效工具之一[8],其可分为g-SSR和EST-SSR两类,而EST-SSR具有开发成本低、效率高、通用性好等特点。EST-SSR标记已经在许多果树中得到广泛开发与应用,如橄榄(Canarium album L. Ranuesch.)[8]、扁桃(Amygdalus communis)[9]、油梨(Persea americana Mill.)[10]、葡萄(Vitis vinifera)[11]等。【本研究切入点】目前有关柠檬种质鉴定和遗传多样性评价的系统性研究仍有待深入,基于转录组测序的EST-SSR分子标记在柠檬上的开发及应用研究鲜见报道。【拟解决的关键问题】本研究利用已公布的柠檬转录组数据对其进行SSR位点分布特征分析及引物设计,选取49份柠檬开展遗传多样性评价,以期为柠檬种质资源鉴定与创新应用提供参考依据。1. 材料与方法
1.1 试验材料
49份柠檬种质采集于福建省热带作物科学研究所柠檬种质资源圃(表1),先择其鲜嫩幼叶,装入标记好的保鲜袋中带回实验室,用改良CTAB法提取DNA,经浓度、纯度和完整性检测后调至50 ng·μL−1,−20 ℃保存。
表 1 柠檬材料Table 1. List of lemon specimens编号 Code 名称 Name 学名 Scientific name 编号 Code 名称 Name 学名 Scientific name 1 大果青柠檬 C. limon (L.) Burm. f. 26 海南白花柠檬 Citrus medica L. 2 巴柑柠檬 C. bergamia Risso. 27 白柠檬1号 C. limonia Osb. 3 大果柠檬1号 C. limon (L.) Burm. f. 28 红柠檬2号 C. limonia Osb. 4 粉红花费米耐劳 C. limon (L.) Burm. f. 29 小青柠 C. limon (L.) Burm. f. 5 尤力克 C. limon (L.) Burm. f. 30 红肉柠檬 Citrus medica L. var.sarcodactylis Swingle 6 四倍体费米耐劳 C. limon (L.) Burm. f. 31 安岳柠檬 C. limon (L.) Burm. f. 7 费米耐劳 C. limon (L.) Burm. f. 32 意大利大果 C. limon (L.) Burm. f. 8 台湾柠檬1号 C. limon (L.) Burm. f. 33 红柠檬1号 C. limonia Osb. 9 台湾柠檬2号 C.aurantifolia (Christm.) Swingle 34 改良北京柠檬 C. limon (L.) Burm. f. 10 香水柠檬 C. limon (L.) Burm. f. 35 诏安柠檬 C. limon (L.) Burm. f. 11 泰国中果 C. limon (L.) Burm. f. 36 五峰2号 Citrus medica L. 12 白花费米耐劳 C. limon (L.) Burm. f. 37 云南土柠檬 Citrus medica L. 13 甜柠檬 C. limon (L.) Burm. f. 38 红花柠檬 C. limon (L.) Burm. f. 14 矮化费米耐劳 C. limon (L.) Burm. f. 39 泰国小果 C. limon (L.) Burm. f. 15 粗柠檬 C.jambhiri Lush. 40 北京青柠檬 C. limon (L.) Burm. f. 16 关东柠檬 C. limon (L.) Burm. f. 41 白柠檬2号 C. limon (L.) Burm. f. 17 大果柠檬2号 C. limon (L.) Burm. f. 42 龙岩土柠檬 Citrus medica L. 18 广东青柠檬 C. limon (L.) Burm. f. 43 台湾红肉柠檬 C. limon (L.) Burm. f. 19 北京柠檬 C. limon (L.) Burm. f. 44 沐阳柠檬 C. limon (L.) Burm. f. 20 柠檬变种1号 C. limon (L.) Burm. f. 45 美亚柠檬 C. limon (L.) Burm. f. 21 山东大黄柠檬 C. limon (L.) Burm. f. 46 五峰3号 Citrus medica L. 22 江苏红肉柠檬 C. limon (L.) Burm. f. 47 泰国青柠檬 C. limon (L.) Burm. f. 23 五峰1号 Citrus medica L. 48 大果柠檬3号 C. limon (L.) Burm. f. 24 红柠檬 C. limon (L.) Burm. f. 49 莆田土柠檬 Citrus medica L. 25 莆田四季柠檬 C. limon (L.) Burm. f. 1.2 柠檬转录组SSR位点查找、引物设计与PCR扩增
以发布在NCBI网站上柠檬转录组数据(检索号为PRJNA606613)为基础,利用MISA(http://pgrc.jpk-gatersleben.de/misa/)软件进行SSR位点查找,SSR位点查找标准、引物设计及SSR-PCR反应体系均参考潘丽芹等[12]的方法。完成引物设计后,从中挑选24对SSR引物并交由广州诺赛生物科技有限公司进行引物合成。
1.3 数据统计与分析
运用ABI3730测序仪进行荧光毛细管电泳检测,利用GeneMarker、GenAlEx 6.501和NTSYS-pc2.10e等软件对数据进行处理分析。
2. 结果与分析
2.1 柠檬转录组SSR位点分布特征
从序列总长约为55.73 Gb的柠檬转录组中获得
68 118 条Unigenes,利用MISA软件识别到40193 个SSR位点,分布在24 297 条Unigenes,SSR分布频率为35.67%;其中8000 条Unigenes含有≥2个SSR位点,10 816 条Unigenes为复合SSR位点(表2)。表 2 柠檬转录组中SSR序列分布Table 2. SSR sequences of lemon transcriptomes条目 Items 数量 Number 比例 Percentage/% 检索序列总数
Retrieved sequences68118 碱基总长度
Total base length/Gb55.73 检测到的SSR总数
SSRs detected40193 含SSR 标记的序列数
Number of sequences containing SSR markers24297 35.67 有1 个SSR 的序列数
Number of sequences with 1 SSR5481 8.05 有≥2个SSR 的序列数
Number of sequences with≥ 2 SSRs8000 11.74 复合SSR 的序列数
Number of composite SSR sequences10816 15.88 由表3可知,柠檬转录组中从单核苷酸到六核苷酸重复类型均存在且各类型比例和间距差异显著,位点平均间距为1.39 kp。其中单核苷酸重复占据绝对优势,数量达32 520个(占总SSR的 80.91%),出现频率最高为47.74%,平均间距最小为1.71 kb;三核苷酸(
3696 个,9.20%)和二核苷酸(3484 个,8.67%)分别位居第二、第三,出现频率分别为5.43%和5.11%,对应平均间距为15.08 kb和16.00 kb;五核苷酸重复数量最少(66个,0.16%),出现频率最低,为0.10%,而平均距离最大为844.38 kb;各SSR类型数量和比例与出现频率变化相一致、与平均距离呈相反。表 3 柠檬转录组SSR序列分布情况Table 3. SSR sequence distribution in transcriptomes of lemon specimensSSR类型
SSR type数量
Number比例
Percentage/%出现频率
Distribution
frequency/%平均距离
Average
distance/kb单核苷酸
Mono-nucleotide32520 80.91 47.74 1.71 二核苷酸
Di-nucleotide3484 8.67 5.11 16.00 三核苷酸
Tri-nucleotide3696 9.20 5.43 15.08 四核苷酸
Tetra-nucleotide331 0.82 0.49 168.37 五核苷酸
Penta-nucleotide66 0.16 0.10 844.38 六核苷酸
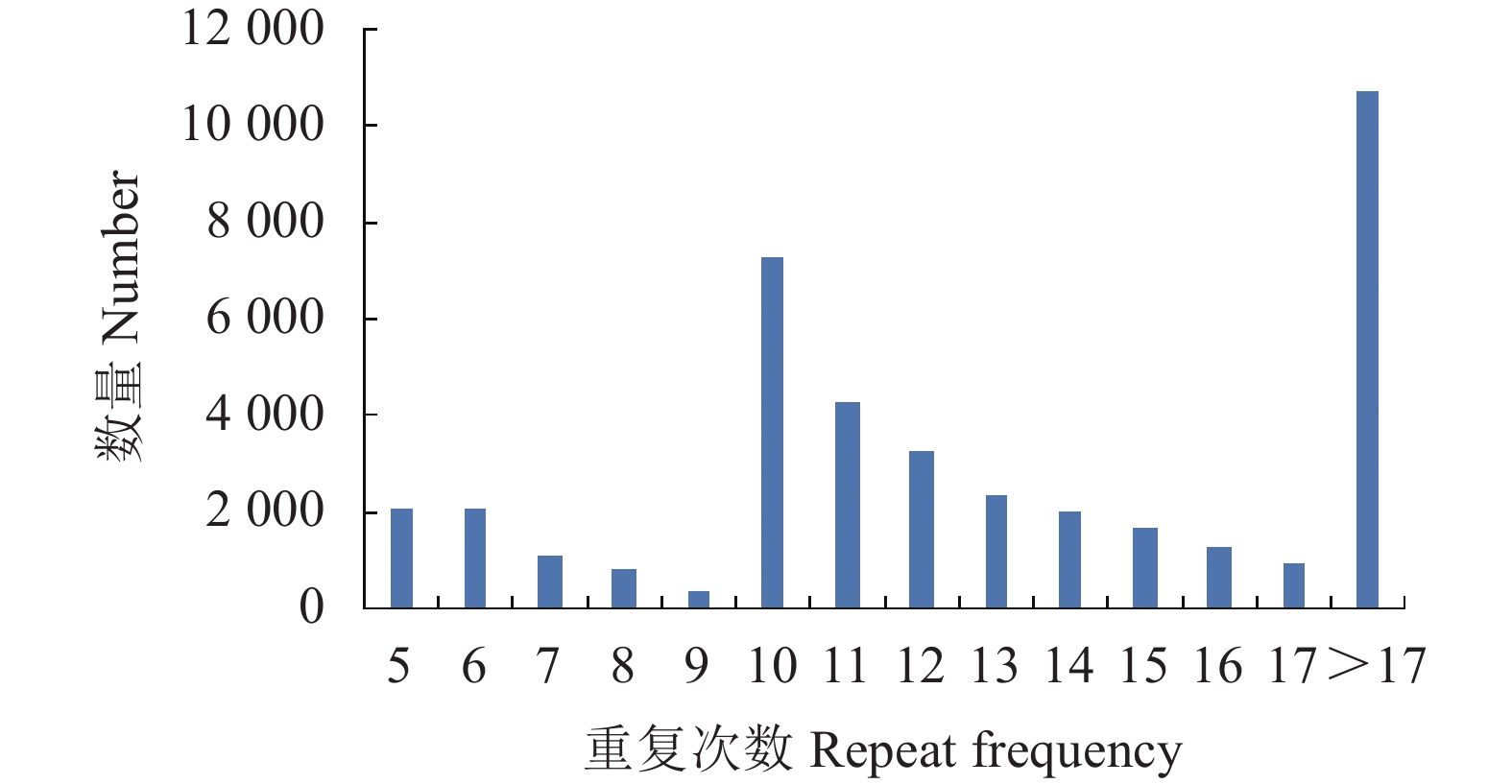
Hexa-nucleotide96 0.24 0.14 580.51 总计 Total 40193 100.00 59.00 1.39 由图1可知,柠檬转录组中SSR的重复次数以10~13次重复为主,占总位点数的42.61%,其中以10次重复数量最多,为
7286 ,占总SSR数的18.13%,其次是11次重复的为4228 个,占比为10.52%,>17次重复的数量有10715 个,占比为26.66%。2.2 转录组中SSR基序重复类型和频率特征
由表4可知,在柠檬转录组中的
40193 个SSR位点检测到90种重复基元,从单核苷酸至六核苷酸重复基元中各有2、4、10、24、15、35种。从柠檬转录组SSR核苷酸基序类型来看,单核苷酸重复基元类型中,A/T为优势重复基元类型,出现30978 次,发生频率为45.48%,占SSR位点总数的77.07%,占单核苷酸基序总数的95.26%,C/G则仅占4.74%。二核苷酸重复基元中AG/CT最多,共2180 次,发生频率为3.20%,占SSR位点总数的5.42%;其次是AC/GT、AT/AT为633次、658次,而CG/CG最少仅出现13次,三者分别占SSR位点总数的1.57%、1.64%、0.03%,表现出明显的偏倚性。三核苷酸重复基元中AAG/CTT为主,共995次,发生频率为1.46%,占SSR位点总数的2.48%;其次是AAT/ATT、AGC/CTG和ATC/ATG,分别占总SSR位点数目的1.71%、1.26%和1.17%。四、五、六核苷酸重复占SSR位点总数比例极低,不足1.50%。表 4 柠檬转录组SSR重复基元分布Table 4. SSR motif distribution in lemon transcriptomes重复类型
Repeat types类型数目
Number of types重复基元
Repeat motifs数目
Number发生频率
Frequency/%比例
Proportion/%单核苷酸
Mononucleotide2 A/T 30978 45.48 77.07 C/G 1542 2.26 3.84 二核苷酸
Dinucleotide4 AG/CT 2180 3.20 5.42 AT/AT 658 0.97 1.64 AC/GT 633 0.93 1.57 CG/CG 13 0.02 0.03 三核苷酸
Trinucleotide10 AAG/CTT 995 1.46 2.48 AAT/ATT 686 1.01 1.71 AGC/CTG 505 0.74 1.26 ATC/ATG 469 0.69 1.17 其他类型
Other types1041 1.44 2.59 四核苷酸
Tetra-nucleotide24 AAAT/ATTT 91 0.13 0.23 AAAG/CTTT 88 0.13 0.22 AAAC/GTTT 22 0.03 0.05 ACTG/AGTC 16 0.02 0.04 其他类型
Other types114 0.17 0.28 五核苷酸
Penta-nucleotide15 AAAAG/CTTTT 19 0.03 0.05 AAAAC/GTTTT 6 0.01 0.01 AAAAT/ATTTT 6 0.01 0.01 AACTC/AGTTG 6 0.01 0.01 AGCTC/AGCTG 6 0.01 0.01 其他类型
Other types23 0.03 0.06 六核苷酸
Hexa-nucleotide35 AAGGAG/CCTTCT 10 0.01 0.02 ACCAGC/CTGGTG 10 0.01 0.02 AAAAAC/GTTTTT 6 0.01 0.01 ACCTCC/AGGTGG 6 0.01 0.01 其他类型
Other types64 0.09 0.16 2.3 SSR引物多态性分析
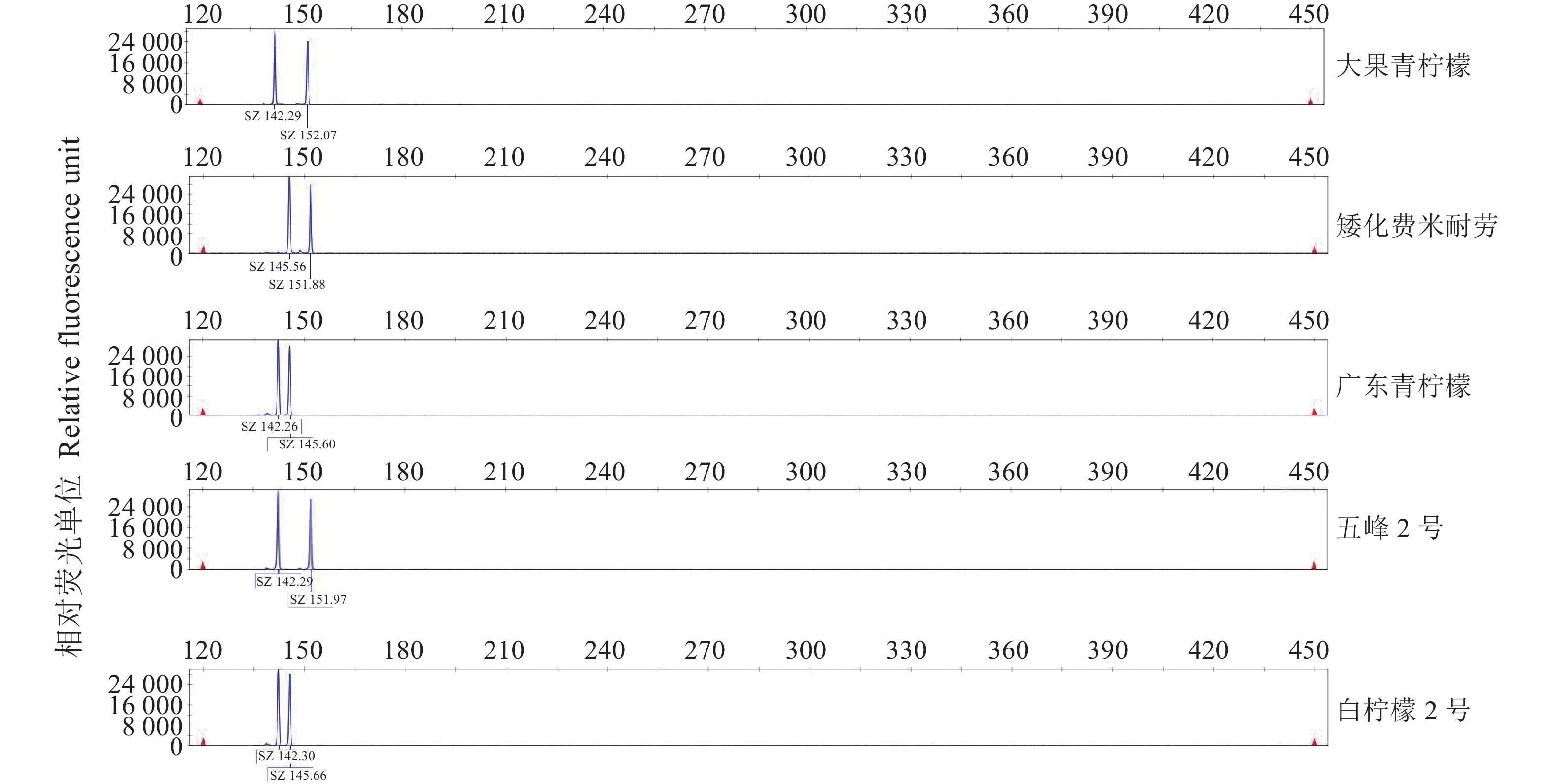
利用Primer3.0软件设计出
4561 对潜在有效性引物,从中选择24对不同重复单元引物进行PCR扩增,筛选出13对引物具有多态性(图2)。利用13对多态性SSR引物分析49份柠檬种质遗传多样性,共检测到47个等位基因;有效等位基因数为2~6,平均为3.615;观察杂合度为0.143~0.905,平均为0.637;期望杂合度为0.187~0.798,平均为0.516,引物的多态信息含量为0.215~0.793,平均为0.528(表5)。表 5 柠檬转录组13对SSR多态性引物信息Table 5. Information on 13 pairs of polymorphic SSR primers in lemon transcriptomes引物编号
Primer No.来源
Gene ID引物系列
Primer sequences(5′-3′)SSR基元
SSR motif有效等位基因
Effective
allele观测杂合度
Observed
heterozygosity期望杂合度
Expected
heterozygosity多态信息含量
Polymorphism
information contentCil-5 TRINITY_DN21626_c0_g1_i1 F:AGGTACGGAACAGGACGATTTC
R:GTCCCACCACCCATGATCATC(TGG) 8 3 0.429 0.351 0.404 Cil-6 TRINITY_DN22713_c0_g1_i3 F:TGGAGGGGAATTGCAGAGAAAT
R:CAAAAGCGTCTGATTCGGATAA(AG) 18 3 0.841 0.664 0.657 Cil-7 TRINITY_DN22780_c0_g1_i1 F:ACAGAGCCGCCGTCGTAATT
R:ACGCTGATGAAGAAGGCTGCTG(ACA) 10 4 0.746 0.566 0.570 Cil-9 TRINITY_DN20983_c0_g1_i1 F:TTGTTTGTTGCAGCTCGATACA
R:GCCGACACCAATTACAGACTTT(GTCT) 6 4 0.317 0.309 0.365 Cil-11 TRINITY_DN21948_c0_g3_i2 F:ACCATCTAAGGGCTTCGTTTTA
R:GTAACGGCTTGCACTCCATCAA(TCGA) 6 2 0.143 0.187 0.215 Cil-12 TRINITY_DN14061_c0_g1_i3 F:CTGCTCGGAGGTAGGGAATTG
R:CGAAGGTGGTGGTTAAGC(TTGA) 7 3 0.762 0.495 0.491 Cil-13 TRINITY_DN39739_c0_g1_i1 F:GCCGTCTCCAGAGCGCAAATT
R:AACACTCACAATCATGCCGCGA(TCT) 8 4 0.905 0.641 0.622 Cil-14 TRINITY_DN12772_c0_g1_i1 F:TCCCCTTTTCTTTGGCGTTCTT
R:TGGGATCATAGCCGGAGTACAA(TCT) 8 4 0.698 0.507 0.507 Cil-15 TRINITY_DN15845_c0_g2_i1 F:GGTTTGTCCCTTGCTGTAAAA
R:CCGGACCTGAAAACTACTCTAT(AC) 10 4 0.746 0.601 0.589 Cil-16 TRINITY_DN22015_c0_g2_i4 F:TGGACACAGCGGCCATTTTGT
R:CGAACCCGTCCCATCTGCATAA(GAGC) 6 2 0.683 0.479 0.474 Cil-17 TRINITY_DN11400_c1_g1_i1 F:AGCCGAATGAGGAACCGAAGAA
R:GCGGTAAATTAGTGGAGGTTGC(TCCCT) 5 5 0.730 0.798 0.793 Cil-19 TRINITY_DN23736_c6_g2_i1 F:CCGACTTTCTTCCCGTCTTCTA
R:GGGATTTAGGCGAGGACAATGA(TCA) 8 3 0.413 0.373 0.436 Cil-24 TRINITY_DN22258_c1_g2_i1 F:GGCGGTGATGTGGGCGAAATA
R:TCCTACTGCTTTGTCACCTTCT(GGA) 8 6 0.873 0.74 0.746 平均值 Average 3.615 0.637 0.516 0.528 2.4 柠檬种质的亲缘关系分析
运用NTsys2.1软件进行UPGMA聚类分析获得49份柠檬种质遗传多样性树状图(图3),以遗传距离0.40为阈值将49份柠檬种质分为5大类群:A类由13份柠檬样品组成,以遗传距离0.20为阈值又可分为2个亚类,A1由10份柠檬类样品组成,分别为大果青柠檬、广东青柠檬、改良北京柠檬、泰国青柠檬、北京柠檬、北京青柠檬、江苏红肉柠檬、诏安柠檬、美亚柠檬、山东大黄柠檬;A2由3份柠檬样品组成,分别为柠檬变种1号、意大利大果柠檬、红花柠檬;B类由18份柠檬样品组成,其中包含1份粗柠檬、2份藜檬、7份柠檬、8份香橼,以遗传距离0.20为阈值又可分为5个亚类,B1由粗柠檬单独组成,为粗柠檬类;B2由2份柠檬样品和3份藜檬类样品组成,分别是关东柠檬、白柠檬1号、白柠檬2号、红柠檬1号、红柠檬2号,B3由1份柠檬样品和1份香橼类样品组成,分别是香水柠檬、红肉柠檬,B4由2份柠檬样品和7份香橼类样品组成,分别为五峰1号、台湾红肉柠檬、莆田土柠檬、龙岩土柠檬、五峰3号、泰国中果柠檬、五峰2号、云南土柠檬、海南白花柠檬,B5由小青柠单独成组成,为柠檬类,来自海南的土柠檬,个头小;C类由15份材料构成,包含1份巴柑柠檬类样品和14份柠檬类样品,分别是巴柑柠檬、大果柠檬1号、莆田四季柠檬、粉红费米耐劳柠檬、四倍体费米耐柠檬、白花费米耐劳柠檬、大果柠檬2号、大果柠檬3号、红柠檬、矮化费米耐劳柠檬、安岳柠檬、沐阳柠檬、尤力克柠檬、费米耐劳柠檬、台湾柠檬1号;D类由2份材料构成,分别是台湾柠檬2号为莱檬类、泰国小果柠檬为柠檬类;E类由甜柠檬单独组成,甜柠檬是柠檬与柑的杂交品种。
3. 讨论与结论
利用MISA软件对55.73 Gb的柠檬转录组进行检索,共识别到
40193 个SSR位点,SSR的发生频率为60.32%,高于橄榄的23.86%[8],油梨的35.05%[10],火龙果(Hylocereus spp.)的6.02%[13],这表明柠檬转录组SSR数量丰富,且不同物种间SSR的发生频率差异大,可能是SSR检索软件、开发SSR的方式及筛选标准和物种不同所导致的。本研究发现,柠檬SSR类型以单、二、三核苷酸重复类型为主,而四、五、六核苷酸重复类型极少,其中以单核苷酸重复类型(A/T)最多,占SSR位点总数的77.07%,这与潘丽芹等[12]、杨仕美等[13]的报道一致,而前人研究表明大多数植物SSR重复类型以二、三核苷酸为主的,产生这种差异可能与SSR检索参数设置和物种本身的基因型差异有关[10,14,15]。本研究中,二核苷酸(AG/CT)和三核苷酸(AAG/CTT)重复类型数量最多,分别占SSR位点总数的5.42%和2.48%,这一结果与扁桃 [9]、油梨[10]、火龙果[13]等植物在转录组开发方面的报道一致。本研究用多态性较好的13对SSR引物对49份柠檬材料进行遗传多样性评价,扩增出47个等位基因,有效等位基因数为2~6个,平均值为3.615,与郭俊等[10]、杨仕美等[13]的研究结果相符,而孙禄娟等[16]对盐生草(Halogeton glomeratus)、杨丹丹等[17]对楸树(Catalpa bungei)的研究结果显示有效等位基因数为3~13个的差异较大,其原因可能与试验群体大小、基因内SSR位点的保守性等[18]有关。本研究中13对多态性引物获得的多态信息含量(polymorphism information content, PIC)平均值为0.528,高于油梨[10]的PIC值0.456,山茶(Camellia japonica)[12]的PIC值0.496,火龙果[13]的PIC值0.45,这可能与本研究所选用材料涵盖丰富的种质类型、来源地广泛及种质资源间遗传背景差异显著有关,说明本研究开发的13对SSR引物用于柠檬遗传多样性检测是可行的,能够真实反映柠檬种质之间丰富的遗传变异类型。
聚类分析结果表明供试的49份材料遗传多样性较为丰富,在遗传距离为0.40时,将供试材料分为5大类,第一类以柠檬类为主,主要以青柠檬作为销售产品,第二类以香橼和藜檬为主,第三类以欧洲栽培柠檬为主如费米耐劳柠檬及其品系构成,第四类由台湾柠檬1号和莱蒙构成,第五类由甜柠檬构成。本研究发现国内品种与国外品种呈现明显的地域聚集特征,但也有部分样品存在跨地域分布现象,如泰国青柠檬、意大利大果柠檬与广东青柠檬等国内品种聚在一起。这种现象在李(Plum)[19]、砂梨(P.pyrifolia)[20],姜(Zingiber officinale)[21]上也有报道,这可能是柠檬在传播过程中长期的自然选择和种间杂交产生基因渗透所致。来源地相同的材料却没有聚为一类,如台湾柠檬、台湾红肉柠檬、香水柠檬和台湾青柠檬均来自台湾,这种现象在栽培大豆(Glycine max L.Merr.)[22]也有报道,说明来源地相同其遗传背景也可能存在差异。尤力克柠檬与费米耐劳及其品系聚成一类,表明尤力克柠檬与费米耐劳柠檬的品系的亲缘关系较近,与前人 [6,23,24]的研究结果相吻合。除少量样品外SSR标记能够将柠檬、莱檬、藜檬、粗柠檬、香橼等有效区分开,聚类结果与传统的形态学分类结果及前人研究成果[3,4,6]大体相同。
综上所述,本研究所开发的13对EST-SSR引物对柠檬及近缘种具有较好区分度和可行性,对柠檬的种质资源评价、鉴定及新品种选育等具有指导意义。
-
表 1 柠檬材料
Table 1 List of lemon specimens
编号 Code 名称 Name 学名 Scientific name 编号 Code 名称 Name 学名 Scientific name 1 大果青柠檬 C. limon (L.) Burm. f. 26 海南白花柠檬 Citrus medica L. 2 巴柑柠檬 C. bergamia Risso. 27 白柠檬1号 C. limonia Osb. 3 大果柠檬1号 C. limon (L.) Burm. f. 28 红柠檬2号 C. limonia Osb. 4 粉红花费米耐劳 C. limon (L.) Burm. f. 29 小青柠 C. limon (L.) Burm. f. 5 尤力克 C. limon (L.) Burm. f. 30 红肉柠檬 Citrus medica L. var.sarcodactylis Swingle 6 四倍体费米耐劳 C. limon (L.) Burm. f. 31 安岳柠檬 C. limon (L.) Burm. f. 7 费米耐劳 C. limon (L.) Burm. f. 32 意大利大果 C. limon (L.) Burm. f. 8 台湾柠檬1号 C. limon (L.) Burm. f. 33 红柠檬1号 C. limonia Osb. 9 台湾柠檬2号 C.aurantifolia (Christm.) Swingle 34 改良北京柠檬 C. limon (L.) Burm. f. 10 香水柠檬 C. limon (L.) Burm. f. 35 诏安柠檬 C. limon (L.) Burm. f. 11 泰国中果 C. limon (L.) Burm. f. 36 五峰2号 Citrus medica L. 12 白花费米耐劳 C. limon (L.) Burm. f. 37 云南土柠檬 Citrus medica L. 13 甜柠檬 C. limon (L.) Burm. f. 38 红花柠檬 C. limon (L.) Burm. f. 14 矮化费米耐劳 C. limon (L.) Burm. f. 39 泰国小果 C. limon (L.) Burm. f. 15 粗柠檬 C.jambhiri Lush. 40 北京青柠檬 C. limon (L.) Burm. f. 16 关东柠檬 C. limon (L.) Burm. f. 41 白柠檬2号 C. limon (L.) Burm. f. 17 大果柠檬2号 C. limon (L.) Burm. f. 42 龙岩土柠檬 Citrus medica L. 18 广东青柠檬 C. limon (L.) Burm. f. 43 台湾红肉柠檬 C. limon (L.) Burm. f. 19 北京柠檬 C. limon (L.) Burm. f. 44 沐阳柠檬 C. limon (L.) Burm. f. 20 柠檬变种1号 C. limon (L.) Burm. f. 45 美亚柠檬 C. limon (L.) Burm. f. 21 山东大黄柠檬 C. limon (L.) Burm. f. 46 五峰3号 Citrus medica L. 22 江苏红肉柠檬 C. limon (L.) Burm. f. 47 泰国青柠檬 C. limon (L.) Burm. f. 23 五峰1号 Citrus medica L. 48 大果柠檬3号 C. limon (L.) Burm. f. 24 红柠檬 C. limon (L.) Burm. f. 49 莆田土柠檬 Citrus medica L. 25 莆田四季柠檬 C. limon (L.) Burm. f. 表 2 柠檬转录组中SSR序列分布
Table 2 SSR sequences of lemon transcriptomes
条目 Items 数量 Number 比例 Percentage/% 检索序列总数
Retrieved sequences68118 碱基总长度
Total base length/Gb55.73 检测到的SSR总数
SSRs detected40193 含SSR 标记的序列数
Number of sequences containing SSR markers24297 35.67 有1 个SSR 的序列数
Number of sequences with 1 SSR5481 8.05 有≥2个SSR 的序列数
Number of sequences with≥ 2 SSRs8000 11.74 复合SSR 的序列数
Number of composite SSR sequences10816 15.88 表 3 柠檬转录组SSR序列分布情况
Table 3 SSR sequence distribution in transcriptomes of lemon specimens
SSR类型
SSR type数量
Number比例
Percentage/%出现频率
Distribution
frequency/%平均距离
Average
distance/kb单核苷酸
Mono-nucleotide32520 80.91 47.74 1.71 二核苷酸
Di-nucleotide3484 8.67 5.11 16.00 三核苷酸
Tri-nucleotide3696 9.20 5.43 15.08 四核苷酸
Tetra-nucleotide331 0.82 0.49 168.37 五核苷酸
Penta-nucleotide66 0.16 0.10 844.38 六核苷酸
Hexa-nucleotide96 0.24 0.14 580.51 总计 Total 40193 100.00 59.00 1.39 表 4 柠檬转录组SSR重复基元分布
Table 4 SSR motif distribution in lemon transcriptomes
重复类型
Repeat types类型数目
Number of types重复基元
Repeat motifs数目
Number发生频率
Frequency/%比例
Proportion/%单核苷酸
Mononucleotide2 A/T 30978 45.48 77.07 C/G 1542 2.26 3.84 二核苷酸
Dinucleotide4 AG/CT 2180 3.20 5.42 AT/AT 658 0.97 1.64 AC/GT 633 0.93 1.57 CG/CG 13 0.02 0.03 三核苷酸
Trinucleotide10 AAG/CTT 995 1.46 2.48 AAT/ATT 686 1.01 1.71 AGC/CTG 505 0.74 1.26 ATC/ATG 469 0.69 1.17 其他类型
Other types1041 1.44 2.59 四核苷酸
Tetra-nucleotide24 AAAT/ATTT 91 0.13 0.23 AAAG/CTTT 88 0.13 0.22 AAAC/GTTT 22 0.03 0.05 ACTG/AGTC 16 0.02 0.04 其他类型
Other types114 0.17 0.28 五核苷酸
Penta-nucleotide15 AAAAG/CTTTT 19 0.03 0.05 AAAAC/GTTTT 6 0.01 0.01 AAAAT/ATTTT 6 0.01 0.01 AACTC/AGTTG 6 0.01 0.01 AGCTC/AGCTG 6 0.01 0.01 其他类型
Other types23 0.03 0.06 六核苷酸
Hexa-nucleotide35 AAGGAG/CCTTCT 10 0.01 0.02 ACCAGC/CTGGTG 10 0.01 0.02 AAAAAC/GTTTTT 6 0.01 0.01 ACCTCC/AGGTGG 6 0.01 0.01 其他类型
Other types64 0.09 0.16 表 5 柠檬转录组13对SSR多态性引物信息
Table 5 Information on 13 pairs of polymorphic SSR primers in lemon transcriptomes
引物编号
Primer No.来源
Gene ID引物系列
Primer sequences(5′-3′)SSR基元
SSR motif有效等位基因
Effective
allele观测杂合度
Observed
heterozygosity期望杂合度
Expected
heterozygosity多态信息含量
Polymorphism
information contentCil-5 TRINITY_DN21626_c0_g1_i1 F:AGGTACGGAACAGGACGATTTC
R:GTCCCACCACCCATGATCATC(TGG) 8 3 0.429 0.351 0.404 Cil-6 TRINITY_DN22713_c0_g1_i3 F:TGGAGGGGAATTGCAGAGAAAT
R:CAAAAGCGTCTGATTCGGATAA(AG) 18 3 0.841 0.664 0.657 Cil-7 TRINITY_DN22780_c0_g1_i1 F:ACAGAGCCGCCGTCGTAATT
R:ACGCTGATGAAGAAGGCTGCTG(ACA) 10 4 0.746 0.566 0.570 Cil-9 TRINITY_DN20983_c0_g1_i1 F:TTGTTTGTTGCAGCTCGATACA
R:GCCGACACCAATTACAGACTTT(GTCT) 6 4 0.317 0.309 0.365 Cil-11 TRINITY_DN21948_c0_g3_i2 F:ACCATCTAAGGGCTTCGTTTTA
R:GTAACGGCTTGCACTCCATCAA(TCGA) 6 2 0.143 0.187 0.215 Cil-12 TRINITY_DN14061_c0_g1_i3 F:CTGCTCGGAGGTAGGGAATTG
R:CGAAGGTGGTGGTTAAGC(TTGA) 7 3 0.762 0.495 0.491 Cil-13 TRINITY_DN39739_c0_g1_i1 F:GCCGTCTCCAGAGCGCAAATT
R:AACACTCACAATCATGCCGCGA(TCT) 8 4 0.905 0.641 0.622 Cil-14 TRINITY_DN12772_c0_g1_i1 F:TCCCCTTTTCTTTGGCGTTCTT
R:TGGGATCATAGCCGGAGTACAA(TCT) 8 4 0.698 0.507 0.507 Cil-15 TRINITY_DN15845_c0_g2_i1 F:GGTTTGTCCCTTGCTGTAAAA
R:CCGGACCTGAAAACTACTCTAT(AC) 10 4 0.746 0.601 0.589 Cil-16 TRINITY_DN22015_c0_g2_i4 F:TGGACACAGCGGCCATTTTGT
R:CGAACCCGTCCCATCTGCATAA(GAGC) 6 2 0.683 0.479 0.474 Cil-17 TRINITY_DN11400_c1_g1_i1 F:AGCCGAATGAGGAACCGAAGAA
R:GCGGTAAATTAGTGGAGGTTGC(TCCCT) 5 5 0.730 0.798 0.793 Cil-19 TRINITY_DN23736_c6_g2_i1 F:CCGACTTTCTTCCCGTCTTCTA
R:GGGATTTAGGCGAGGACAATGA(TCA) 8 3 0.413 0.373 0.436 Cil-24 TRINITY_DN22258_c1_g2_i1 F:GGCGGTGATGTGGGCGAAATA
R:TCCTACTGCTTTGTCACCTTCT(GGA) 8 6 0.873 0.74 0.746 平均值 Average 3.615 0.637 0.516 0.528 -
[1] 高俊燕,周东果,岳建强,等. 费米耐劳柠檬引种研究初报[J]. 西南农业学报,2008,21(3) :760−763. DOI: 10.3969/j.issn.1001-4829.2008.03.047 GAO J Y,ZHOU D G,YUE J Q,et al. Original research on the performance of the Femminello lemon introduced into Dehong state[J]. Southwest China Journal of Agricultural Sciences,2008,21(3) :760−763. (in Chinese) DOI: 10.3969/j.issn.1001-4829.2008.03.047
[2] 李进学,高俊燕,付小猛,等. 云南柠檬产业发展的优势与潜力[J]. 热带农业科学,2017,37(10) :99−104,110. LI J X,GAO J Y,FU X M,et al. Advantages and potential of lemon industry in Yunnan Province[J]. Chinese Journal of Tropical Agriculture,2017,37(10) :99−104,110. (in Chinese)
[3] 沈兆敏,柠檬优质丰产栽培[M]. 北京:金盾出版社,2002:1–8. [4] 周齐铭,彭长江,计长远. 柠檬栽培技术[M]. 成都:四川科学技术出版社,2005:7–8. [5] 马丽丽,解凯东,叶俊丽,等. 基于SSR标记的早花柠檬亲缘关系分析[J]. 果树学报,2013,30(3) :381−385,506. MA L L,XIE K D,YE J L,et al. Phylogenetic relationship for early flowering lemon by SSR markers[J]. Journal of Fruit Science,2013,30(3) :381−385,506. (in Chinese)
[6] 张树伟. 香水柠檬无籽成因和相关基因的分离与鉴定[D]. 南宁:广西大学,2014. ZHANG S W. Causes of Seedless Forming and identifiction of seedless related genes from ‘Xiangshui’ lemon[D]. Nanning:Guangxi University,2014. (in Chinese)
[7] 尤桂春,林文忠,武竞超,等. 柠檬种质资源表型性状遗传多样性分析[J]. 中国南方果树,2020,49(06) :34−39. YOU G C,LIN W Z,WU J C,et al. Analysis genetic diversity of lemon germplasm resources with phenotypic traits[J]. South China Fruits,2020,49(06) :34−39. (in Chinese)
[8] 谢倩,张诗艳,江来,等. 橄榄转录组SSR信息分析及分子标记开发与应用[J]. 园艺学报,2023,50(11) :2350−2364. XIE Q,ZHANG S Y,JIANG L,et al. Analysis of Canarium album transcriptome SSR information and molecular marker development and application[J]. Acta Horticulturae Sinica,2023,50(11) :2350−2364. (in Chinese)
[9] 秦玥,朱高浦,郭欢欢,等. 扁桃EST-SSR分子标记开发及遗传多样性评价[J]. 林业科技通讯,2023(6) :20−25. QIN Y,ZHU G P,GUO H H,et al. Development of EST-SSR molecular marker and evaluation of genetic diversity in Prunus communis[J]. Forest Science and Technology,2023(6) :20−25. (in Chinese)
[10] 郭俊,朱婕,谢尚潜,等. 油梨转录组SSR分子标记开发与种质资源亲缘关系分析[J]. 园艺学报,2020,47(8) :1552–1564. GUO J,ZHU J,XIE S Q,et al. Development of SSR molecular markers based on transcriptome and analysis of genetic relationship of germplasm resources in Avocado[J]. Acta Horticulturae Sinica,2020,47(8) :1552−1564. (in Chinese)
[11] 毛娟,梁国平,卢世雄,等. 葡萄EST-SSR标记的开发及其在遗传多样性分析中的应用[J]. 中外葡萄与葡萄酒,2019(6) :12−19. MAO J,LIANG G P,LU S X,et al. Development of the EST-SSRs marker and application on genetic diversity analysis from grapevine[J]. Sino-Overseas Grapevine & Wine,2019(6) :12–19. (in Chinese)
[12] 潘丽芹,李纪元,李绍翠,等. 基于山茶转录组的SSR标记开发及亲缘关系分析[J]. 北京林业大学学报,2019,41(7) :111–120. PAN L Q,LI J Y,LI S C,et al. Development of SSR markers based on transcriptome of Camellia japonica and analysis of genetic relationship[J]. Journal of Beijing Forestry University,2019,41(7) :111−120. (in Chinese)
[13] 杨仕美,乔光,毛永亚,等. 基于火龙果转录组测序的SSR标记开发及种质亲缘关系分析[J]. 分子植物育种,2018,16(24) :8096−8110. YANG S M,QIAO G,MAO Y Y,et al. SSR markers development and genetic relationship analysis of germplasm based on transcriptomic sequencing of Pitaya[J]. Molecular Plant Breeding,2018,16(24) :8096−8110. (in Chinese)
[14] 饶龙兵,杨汉波,郭洪英,等. 基于桤木属转录组测序的SSR分子标记的开发[J]. 林业科学研究,2016,29(6) :875−882. RAO L B,YANG H B,GUO H Y,et al. Development of SSR molecular markers based on transcriptome sequences of alnus[J]. Forest Research,2016,29(6) :875−882. (in Chinese)
[15] 李太强,刘雄芳,万友名,等. 滇东南濒危植物长梗杜鹃转录组微卫星特征分析[J]. 林业科学研究,2017,30(4) :533−541. LI T Q,LIU X F,WANG Y M,et al. Characteristic analysis of microsatellites in the transcriptome of Rhododendron Longipedicellatum,an endangered species endemic to southeastern Yunnan,china[J]. Forest Research,2017,30(4) :533−541. (in Chinese)
[16] 孙禄娟,何建军,汪军成,等. 基于全长转录组测序的盐生草SSR标记开发及其遗传多样性分析[J]. 草业学报,2022,31(8) :199−210. SUN L J,HE J J,WANG J C,et al. Development of SSR markers based on full-length transcriptome sequencing and genetic diversity analysis of Halogeton glomeratus[J]. Acta Prataculturae Sinica,2022,31(8) :199−210. (in Chinese)
[17] 杨丹丹,马玲玲,李亚,等. 楸树EST-SSR标记开发及种质资源的遗传多样性分析[J]. 分子植物育种,2020,18(4) :1216−1223. YANG D D,MA L L,LI Y,et al. Development of EST-SSR marker and genetic diversity of germplasm sesources in Catalpa bungei[J]. Molecular Plant Breeding,2020,18(4) :1216−1223. (in Chinese)
[18] 杜庆章. 利用连锁与连锁不平衡联合作图解析毛白杨重要性状的等位遗传变异[D]. 北京:北京林业大学,2014. DU Q Z. Dissection of allelic variation underlying important traits in Populus tomentos Carr.by usingjoint linkage and linkage disequilibrium mapping[D]. Beijing:Beijing Forestry University,2014. (in Chinese)
[19] 陈红,杨迤然. 贵州李资源遗传多样性及亲缘关系的ISSR分析[J]. 果树学报,2014,31(2) :175−180. CHEN H,YANG Y R. Genetic diversity and relationship of Plum resources in Guizhou analysed by ISSR markers[J]. Journal of Fruit Science,2014,31(2) :175−180. (in Chinese)
[20] 张起,安华明. 贵州砂梨种质资源遗传多样性的ISSR分析[J]. 果树学报,2015,32(1) :6−12. ZHANG Q,AN H M. Genetic diversity of Pyrus pyrifolia resources distributed in Guizhou province revealed by ISSR markers[J]. Journal of Fruit Science,2015,32(1) :6−12. (in Chinese)
[21] 陈孝赏,汤紫依,何海叶,等. 基于ISSR标记的姜种质资源遗传多样性分析[J]. 分子植物育种,2024,22(20) :6757−6766. CHEN X S,TANG Z Y,HE H Y,et al. Genetic diversity analysis of Ginger (Zingiber officinale) germplasm resources based on ISSR molecular marker[J]. Molecular Plant Breeding,2024,22(20) :6757−6766. (in Chinese)
[22] 宋喜娥,李英慧,常汝镇,等. 中国栽培大豆(Glycine max (L.) Merr.) 微核心种质的群体结构与遗传多样性[J]. 中国农业科学,2010,43(11) :2209−2219. SONG X E,LI Y H,CHANG R Z,et al. Population structure and genetic diversity of mini core collection of Cultivated Soybean (Glycine max (L.) Merr.) in china[J]. Scientia Agricultura Sinica,2010,43(11) :2209−2219. (in Chinese)
[23] 杨晓明. 柑橘亚科植物系统发育基因组学及野生枸橼、宜昌橙谱系地理学研究[D]. 武汉:华中农业大学,2017. YANG X M. The phylogenomic of aurantioideae and phylogeography of Wild Citron(Citrus medica) and Ichang Papeda(Citrus ichangensls) [D]. Wuhan:Huazhong Agricultural University,2017. (in Chinese)
[24] 李小孟. 柑橘及其近缘属植物的分子进化与栽培柑橘的起源研究[D]. 重庆:西南大学,2010. LI X M. Molecular phylogeny of the true citrus fruit trees group (Aurantioideae,Rutaceae) and the origin of cultivated citrus[D]. Chongqing:Southwest University,2010. (in Chinese)




 下载:
下载: