Epidemiology of Waterfowl Circoviral diseases and Characteristics of Genomes in Natural Cross-species Infection on Goose
-
摘要:目的
探明鹅群水禽圆环病毒(waterfowl circovirus, WFCV),包括鹅圆环病毒(goose circovirus, GoCV)和鸭圆环病毒(duck circovirus, DuCV)的感染情况及首例自然共感染病例中两种WFCV(GoCV和DuCV)基因组分子特征。
方法应用三引物PCR方法对2023—2024年自福建、广西和安徽等地养鹅场收集的153份患病鹅组织样品进行WFCV检测,并对发现的一例GoCV和DuCV自然共感染病例中两种病毒全基因组进行序列测定与分析。
结果自153份鹅样品中检测到53份WFCV阳性样品,阳性率为34.6%,其中GoCV单纯阳性样品47份,DuCV单纯阳性5份,1份安徽来源的样品(AH2367)为GoCV和DuCV共感染。AH2367样品中的GoCV(Go-AH2367)基因组全长
1821 nt,与GenBank数据库中的GoCV核苷酸序列同源性在82.9%~99.0%,与DuCV序列同源性在67.0%~70.9%;DuCV(Du-AH2367)基因组全长1992 nt,与GenBank数据库中的DuCV序列同源性在84.4%~99.7%,与GoCV序列同源性在66.1%~68.8%。共感染病毒的两个主要功能蛋白(Rep、Cap)氨基酸序列仅出现零星位点变异,未出现基因重组、位点缺失和插入,蛋白中的重要功能基序均高度保守。遗传进化分析表明,Go-AH2367株与基因 I 型GoCV处于同一进化分支,Du-AH2367株与基因1型DuCV亲缘关系最近,处于同一进化分支。结论鹅群中WFCV感染较为严重,以GoCV为主,伴有DuCV的跨种感染及GoCV和DuCV共感染,呈现病毒生态多样性流行。共感染样品中的两株病毒基因组遗传稳定,未出现基因缺失或重组等明显基因变异。
Abstract:ObjectiveThe prevalence of waterfowl circoviruses (WFCVs) including goose circovirus (GoCV) and duck circovirus (DuCV) at geese farms was surveyed, and molecular characteristics of the GoCV and DuCV genomes in the first case of natural co-infection on geese studied.
MethodFrom the flocks at geese farms in 6 provinces, 153 tissue specimens were collected during 2023–2024. GoCV and DuCV were detected by using a tri-primer PCR method. Genome sequences of the viruses in samples of the first natural co-infection case were determined.
ResultFifty-three WFCVs were identified from the collected specimens at a positive rate of 34.6%. Of which, 47 were positive for GoCV, 5 for DuCV, and one from Anhui Province (named as AH2367) with both GoCV and DuCV. The whole genome sequence of Go-AH2367 was
1821 nt in length with a homology of 82.9%–99.0% with GoCV and 67.0%–70.9% with DuCV, while that of Du-AH2367 was1992 nt long with a homology of 84.4%–99.7% with DuCV and 66.1%–68.8% with GoCV. The two major functional proteins of the Go-AH2367 and Du-AH2367(Rep and Cap) were consisted of highly conserved critical motifs with a few sporadic site variations but free of gene recombination, site deletion or insertion on the amino acid sequences. The phylogenetic tree of Go-AH2367 was clustered into an evolutionary branch of the Genotype 1 GoCV isolates, while that of Du-AH2367 of the Genotype I DuCV isolates in the same evolutionary branch.ConclusionThe WFCV infections in geese occurred in China were severe. They were mostly the GoCV type with only one confirmed cross-species case that could cause a diverse epidemic. The genomes of Go-AH2367 and Du-AH2367 found in the co-infected goose were genetically stable with no apparent variations by deletion or recombination.
-
0. 引言
【研究意义】水禽圆环病毒(waterfowl circovirus, WFCV)主要包括鹅圆环病毒(goose circovirus, GoCV)和鸭圆环病毒(duck circovirus, DuCV)。根据国际病毒分类委员会(International Committee on Taxonomy of Viruses, ICTV)分类建议,WFCV为圆环病毒科(Circoviridae)圆环病毒属(Circovirus)成员,是一种无囊膜 DNA 病毒,基因组为共价闭合环状单链基因组,大小为
1821 ~1996 nt,含有6个主要开放阅读框(open reading frame, ORF)V1~V3和C1~C3,其中ORF V1 编码病毒基因组复制相关的蛋白(Rep),ORF C1编码病毒抗原相关的衣壳蛋白(Cap)[1]。GoCV于1999年首次在德国的鹅群中被发现[2],而DuCV则于2003年首次在德国的鸭群中被发现[3],随后这两种病毒感染在世界各地陆续被报道[4−13],且临床感染率呈上升趋势[14−17],近来还出现了WFCV交叉跨种感染病例[18−22]。深入开展水禽WFCV的感染情况调查,明确病毒基因组的分子特征和变异规律,可为开展该病分子致病机制研究及制定行之有效的疫病防控策略提供科学依据。【前人研究进展】GoCV主要引起鹅生长发育迟缓、羽毛絮乱、腹泻,法氏囊、胸腺和脾脏等组织淋巴细胞减少和组织细胞增生,以及法氏囊中单核细胞可见嗜碱性包涵体的存在[23]。我国大陆于2005年首次报道了鹅群中存在GoCV的感染[8],随后开始逐渐发展流行。尽管调查发现鹅感染的圆环病毒主要为GoCV[24],但陆续有研究发现跨宿主感染现象,赵光伟等[18]2009年首次报道了重庆一例患病鹅中检测到DuCV,Shehata等[19]在2013年波兰境内收集的一批发生鹅细小病毒感染的观赏鸭样品中检测到了GoCV的共感染,Xu等[20,21]在鸭群中发现鹅圆环病毒,随后又在鹅群中检测出到DuCV基因1型和2型。【本研究切入点】鉴于当前鹅群较为复杂的WFCV病毒生态流行状况,亟需对鹅群开展圆环病毒流行病学调查。【拟解决的关键问题】本研究应用三引物检测方法对近年来收集的临床鹅样品开展WFCV感染调查,以期明确我国当前鹅群中GoCV和DuCV两种病毒的流行特点,解析发现的自然共感染病例中的GoCV与DuCV基因组分子特征及变异情况,为全面了解鹅群中WFCV流行规律及科学防治提供依据。1. 材料与方法
1.1 试验材料
1.1.1 病料收集
2023—2024年,自福建、广西和安徽等6个省(自治区)共收集到153份鹅肝脏、脾脏、胸腺、法氏囊等组织样品,这些组织来源于临床上生长发育不良/体况消瘦、羽毛脱落及死亡的鹅,初步怀疑存在水禽圆环病毒感染,样品收集后于−80 ℃保存备用,相关信息见表1。
表 1 收集的鹅组织样品信息Table 1. Information on tissue specimens collected from geese farms收集时间
Collection time组织样品来源与数量 Sample sources and number/份 合计 Total/份 福建 广西 安徽 广东 江西 浙江 2023年 28 21 25 10 8 7 99 2024年 19 13 5 8 5 4 54 合计 Total 47 34 30 18 13 11 153 1.1.2 主要试剂与仪器
Simply P Virus DNA/RNA Extraction Kit购自杭州博日科技股份有限公司,2 × Phanta Max Master Mix购自南京诺唯赞生物科技股份有限公司,DL2000 DNA Marker购自北京宝日医生物技术有限公司,高速冷冻离心机(武汉基因科技有限公司),梯度PCR仪(Bio-Rad公司),高速低温组织研磨仪(武汉赛维尔科技有限公司)。
1.2 试验方法
1.2.1 病料处理与核酸提取
取适量采集的脏器置于研磨管中,加入3倍体积的1×PBS与钢制研磨珠,经高速低温组织研磨仪匀浆后,反复冻融3次,于高速冷冻离心机4 ℃ 12 000 ×g 离心10 min;取200 μL 上清液使用Simply P Virus DNA/RNA Extraction Kit提取纯化核酸,按照试剂盒说明书进行,提取的病毒核酸储存于−80 ℃。
1.2.2 GoCV与DuCV检测
样品GoCV与DuCV的检测参照王梦鸽等[25]的方法,由生工生物工程(上海)股份有限公司合成,可同时检测GoCV与DuCV引物对:CVF 5′-AAY CWC GCG GGA AGT GGT GGG A -3′;DuCV-R1:5′- TTC TAR GCA TAA ACG AGA TC -3′;GoCV-R1:5′- ATA MGA TTC GGA CAA TGG ACT G -3′。以提取的核酸为模板,进行GoCV与DuCV的PCR检测。反应体系:2 × Phanta Max Master Mix 10 μL,CVF(10 μmol·L−1) 0.6 μL,DuCVR (10 μmol·L−1)0.45 μL,GoCVR (10 μmol·L−1)0.3 μL,模板1 μL,RNase Free H2O补足至20 μL。反应条件:95 ℃预变性5 min;95 ℃ 变性30 s,54.6 ℃退火30 s,72 ℃ 延伸30 s,36个循环;72 °C彻底延伸10 min。反应结束后经1%琼脂糖凝胶电泳检测。阳性样品送至生工生物工程(上海)股份有限公司进行测序。
1.2.3 全基因组扩增与序列测序
根据GenBank上公布的GoCV和DuCV全基因序列,应用Primer 3 plus 在线引物设计程序,在王梦鸽等[25]检测引物的基础上,设计反向扩增引物,扩增余下的基因片段。引物对序列包括GoCV-F1:5′-CGT TGT CTT GAA TGG AGT TTA GCC A-3′,GoCV-R2:5′- ATA GCC RTC CCA CCA CTT CCC -3′;DuCV-F1:5′-GGA TTA AAC ACT TGG GCA GC-3′,DuCV-R2:5′-GTA ACG GGT AAC GGT CGG TA-3′,其中GoCV-F1与GoCV-R2用于扩增GoCV基因片段,DuCV-F1与DuCV-R2用于扩增DuCV基因片段。反应体系:2×Phanta Max Master Mix 10 μL,上下游引物(10 μmol·L−1)各0.25 μL,模板1 μL,ddH2O补足至20 μL。反应条件:95 ℃预变性5 min;95 ℃ 变性30 s,55~58 ℃退火30 s,72 ℃ 延伸2 min,35个循环;72 °C彻底延伸10 min。反应结束后经1%琼脂糖凝胶电泳检测扩增结果。将阳性样品送至生工生物工程(上海)股份有限公司进行测序。
1.3 数据分析
将测序结果应用DNAStar软件SeqMan程序进行拼接整理,获得的全基因组序列与GenBank数据库中的GoCV和DuCV基因组序列进行比对分析。应用Lasergene 7.0软件包中的MegAlign程序分别进行GoCV和DuCV全基因组同源性分析,并应用TBtools-Ⅱ 软件Heatmap程序绘制同源性热图[26]。应用 MEGA 11 软件 phylogeny 程序进行建树。采用Neighbor Joining 方法,bootstrap method重复
1000 次,采用默认模型进行全基因组遗传进化分析。将获得共感染毒株的Rep及Cap蛋白氨基酸序列与NCBI数据库中的相应蛋白进行比较。应用MegAlign程序分别进行GoCV和DuCV主要功能蛋白同源性分析。应用在线程序BepiPred 3.0 Server 预测分析共感染毒株的主要功能Cap蛋白 B 细胞抗原表位。
为研究是否存在重组的事件,将采集的自然共感染病例样本与 GoCV 和 DuCV 参考序列进行比较。使用重组检测程序 RDP5 的默认设置对序列进行分析,并应用Simplot 3.5.1进行验证。
2. 结果与分析
2.1 GoCV与DuCV检测
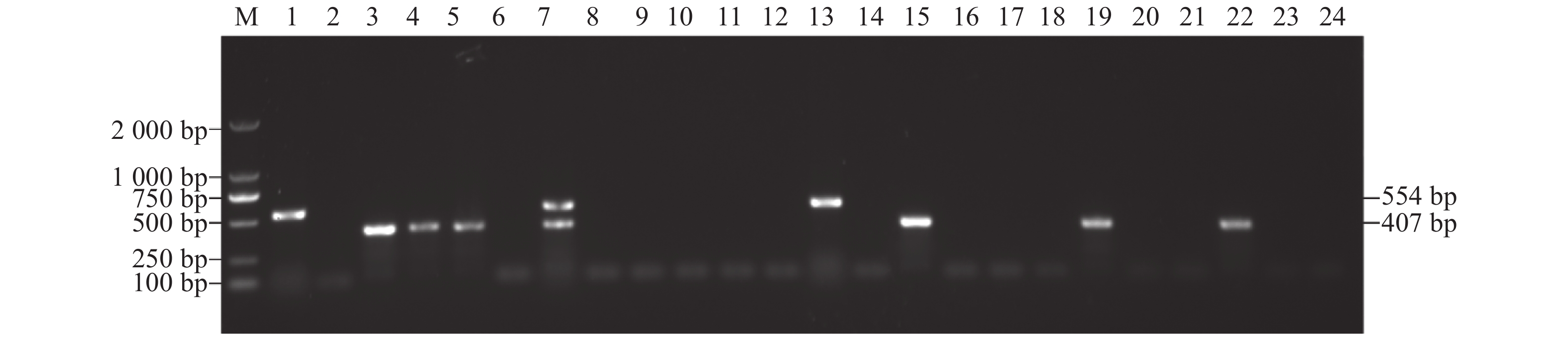
对采集的鹅组织样品进行GoCV与DuCV的PCR检测,检出大小为407 bp和554 bp的两条目的条带,其中407 bp的扩增片段为GoCV,554 bp大小的扩增片段为DuCV,片段大小与预期相符。在153份样品中,共检测出53份WFCV核酸阳性,阳性率达34.6%。其中,GoCV阳性样品47份,阳性率为30.7%(47/153),DuCV阳性样品5份,阳性率为3.3%(5/153),而一份来自安徽的鹅组织中同时检出GoCV与DuCV,部分结果如图1所示。
2.2 共感染病例病毒序列分析
2.2.1 全基因组分子特征及同源性分析
全基因组测序结果显示,共感染病例中的GoCV Go-AH2367株基因组全长1821 nt,ORF V1长882 nt,编码293个氨基酸的Rep蛋白;ORF C1长753 nt,编码250个氨基酸的Cap蛋白。共感染病例中的DuCV Du-AH2367株基因组全长1992 nt,ORF V1长879 nt,编码292个氨基酸Rep蛋白;ORF C1长774 nt,编码257个氨基酸的Cap蛋白。
获得的Go-AH2367株与Du-AH2367株的全基因组序列之间的同源性仅为69%。两株WFCV基因序列与已发布的GoCV和DuCV毒株序列同源性分析表明(图2),GoCV Go-AH2367株与GoCV(KR869727.1~AF536933.1)的全基因组序列同源性在82.9%~99.0%,与DuCV-1(MN078102.1~PP056136.1)的同源性在78.5%~79.5%,与DuCV-2(MN928806.1~ON227557.1)的同源性在79.2%~81.0%,与DuCV-3病毒株(OP432310.1)的同源性仅为70.9%。DuCV病毒株Du-AH2367株全基因序列与DuCV-1(EU344803.1~OR134489.1)的同源性最高,在93.2%~99.7%,与DuCV-2(MK814585.1~MN928807.1)的序列同源性在83.2%~92.9%,而与DuCV-3株的序列同源性较低,仅为73.7%(OP432310.1),与GoCV(KR869727.1~AF536933.1)的同源性仅为66.1%~68.8%。
2.2.2 全基因组遗传进化分析
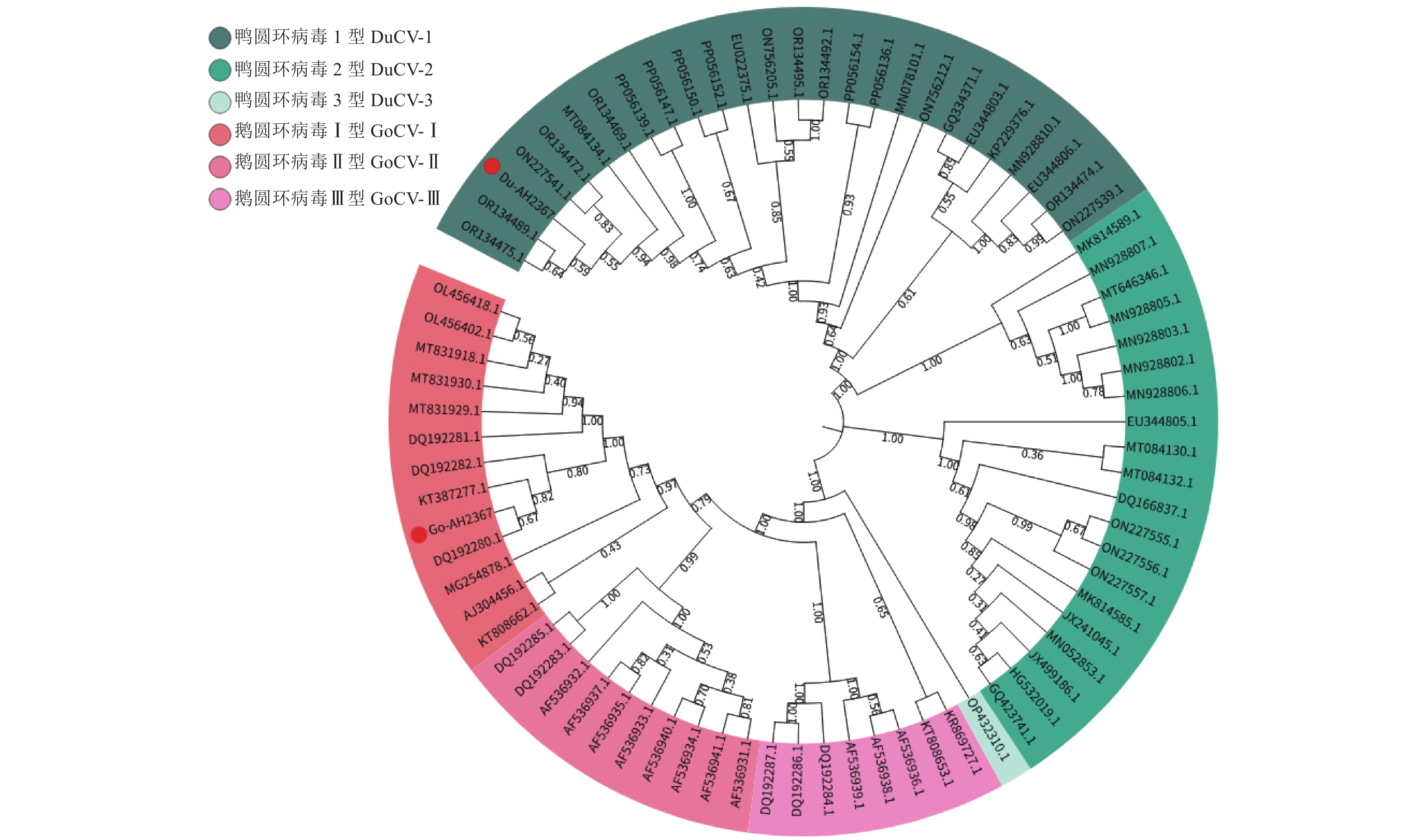
将本研究获得的GoCV Go-AH2367与DuCV Du-AH2367株与GenBank数据库中的30株GoCV、45株DuCV全基因组序列进行遗传进化分析。结果(图3)显示,Go-AH2367株处于GoCV病毒株进化分支中,与2015年山东的鹅源GoCV株 Shandong(KT387277.1)遗传距离最近,与2012年波兰的鹅病毒株2GK(KR869727.1)及2014年波兰的鹅病毒株DG5(KT808653.1)遗传距离较远。DuCV Du-AH2367株处于DuCV大进化分支中,与DuCV-1病毒株处于同一小进化分支,与广西2022年的鸭源DuCV株DuCV-GX51-2022(OR134489.1)和2020年鸭源DuCV株DuCV-GX28-2020(OR134475.1)亲缘关系最近。
2.2.3 共感染病毒基因组重组分析
使用RDP5分别对GoCV Go-AH2367和DuCV Du-AH2367以及GenBank数据库中76株WFCV基因组序列(46株DuCV和30株GoCV )参考序列进行重组分析。结果显示GoCV Go-AH2367和DuCV Du-AH2367与参考序列之间均未发生重组事件。
2.2.4 GoCV Go-AH2367毒株主要功能蛋白变异分析
GoCV Go-AH2367毒株ORF V1基因编码氨基酸序列同源性分析结果表明,GoCV Go-AH2367 ORF V1与GenBank数据库中GoCV ORF V1 同源性为92.15%~100%,与DuCV ORF V1 的同源性在81.2%~84.2%,与2015年山东的鹅源GoCV株 Shandong(ALZ45069.1)氨基酸序列同源性达100%。ORF C1基因编码氨基酸序列同源性分析结果显示,Go-AH2367株ORF C1与GenBank数据库中的GoCV ORF C1 同源性为79.9%~100%,与GenBank数据库中的DuCV-1 ORF C1 序列同源性为45.0%~48.8%,与2015年山东鹅源GoCV株 Shandong(ALZ45071.1)及2005年浙江鹅源GoCV病毒株yk3(ABA39142.1)ORF C1同源性高达100%。
GoCV Go-AH2367毒株ORF V1长882 nt,编码一个由293个氨基酸组成的Rep蛋白,该蛋白上与滚环复制相关的3处功能基序I(FTINNP)、II(TPHLQGF)、III(YCSKE),及病毒dNTP结合域超家族解旋酶基序Walker A(GRPGSGKSR)和B(DDFYGW)均高度保守;ORF C1长753 nt,编码一个由250个氨基酸组成的Cap蛋白,与已发布的基因I型GoCV(GoCV-I)Cap蛋白 8个B细胞抗原表位(18–22 nt、57–65 nt、72–84 nt、94–104 nt、127–168 nt、179–190 nt、195–199 nt、220–231 nt)高度保守,与GoCV-Ⅱ和GoCV-Ⅲ在这些表位分别存在3个和5个位点差异(图4)。
2.2.5 DuCV Du-AH2367毒株主要功能蛋白变异分析
DuCV Du-AH2367毒株ORF V1基因编码氨基酸序列同源性分析结果表明,DuCV Du-AH2367 ORF V1与GenBank数据库中的GoCV ORF V1 序列同源性为82.2%~85.1%,与DuCV-1 ORF V1 的同源性为96.2%~100.0%,与DuCV-2 ORF V1 的同源性为92.8%~99.7%,与DuCV-3 ORF V1 的同源性为89%,与2018年山东鸭源DuCV病毒株SDHZ1223(QIP76518.1)等多株DuCV-1的ORF V1核苷酸序列同源性达100%。Du-AH2367株ORF C1与GenBank数据库中的GoCV ORF C1的同源性为46.9%~51.4%,与GenBank数据库中的DuCV-1 ORF C1的同源性为92.6%~99.6%,与DuCV-2 ORF C1的同源性为86%~94.6%,与DuCV-3 ORF C1的同源性为50.8%,差异较大,与2018年山东鸭源DuCV病毒株SDHZ1223(QIP76519.1)等多株DuCV-1的ORF C1序列同源性高达99.6%。
DuCV Du-AH2367毒株ORF V1长879 nt,编码 一个由292个氨基酸组成的Rep蛋白。该蛋白含有滚环复制相关及dNTP结合域超家族解旋酶基序等多个功能基序,这些基序与已报道的基因1型DuCV毒株高度一致。ORF C1长774 nt,编码一个由257个氨基酸组成的Cap蛋白,与DuCV-1 Cap蛋白 6个B细胞抗原表位(5–32 nt、56–88 nt、101–113 nt、130–139 nt、147–195 nt、229–235 nt)高度保守,与DuCV-2和DuCV-3在这些表位分别存在11个和110个位点差异(图5)。
3. 讨论
水禽圆环病毒可引起禽生长迟缓、腹泻和羽毛脱落等,损伤免疫器官,并引起因淋巴细胞严重减少的免疫抑制性疾病[27−29]。已有的病例报道表明两种WFCV具有各自的易感动物,具有明显的种属特异性,GoCV仅感染不同品种和日龄的鹅,DuCV仅感染不同品种和日龄的鸭[30]。赵光伟等[18]于2009年首次报道了自重庆一例患病鹅中检测到DuCV;2024年本团队在评估建立的鸭圆环病毒新型检测技术特异性时,自早期收集的27份患病鹅样品中检测到3份DuCV-2阳性样品[22];Xu等[20]也报道自2021年和2022年收集的鹅组织样品中分别检测到DuCV-1和DuCV-2。与鹅群中出现了DuCV跨种感染类似,鸭群中同样检测到GoCV的跨种感染。2013年Shehata等[19]从观赏鸭中检测到1例GoCV阳性样品;2024年Xu等[21]开展鸭圆环病毒分子流行病学调查时发现,从河南和安徽的两家养鸭场采集的10份样品中均检测到GoCV,WFCV感染呈现出了极为复杂的流行态势。
本研究对收集的153份鹅样品进行GoCV和DuCV检测发现,鹅群水禽圆环阳性率高达34.6%(53/153),其中GoCV单纯感染的鹅阳性样品占总阳性样品的88.7%(47/53),DuCV单纯感染的鹅阳性样品占总阳性样品9.4%(5/53),另外还发现1例GoCV和DuCV共感染阳性样品。可见,鹅群中WFCV感染呈现病毒生态多样性,以GoCV感染为主,同时还存在DuCV的跨种感染,以及GoCV和DuCV的共感染的情况。经病例回访跟踪调查,该样品来源于安徽一家鸭、鹅混养的农户送检的鹅病例,实验室的病原检测发现该病例还存在小鹅瘟病毒感染。可见该病例存在较为严重的多病原共感染现象,这可能与水禽的饲养方式有关。尽管网床平养、笼养等设施化养殖模式已在我国水禽(尤其是肉鸭)养殖中广泛应用,但东南沿海地区的水禽养殖户或农户仍较多地采用地面旱养或地面加水域圈养等方式,且存在鸡、鸭、鹅混养现象,这些因素为GoCV和DuCV在水禽中的广泛流行及交叉跨种感染提供了有利条件。
本研究在鹅中发现的共感染病例是迄今发现的全球首例GoCV和DuCV共感染病例。对共感染鹅病例样品中的GoCV与DuCV的全基因组分子特征、遗传进化、ORF V1和ORF C1编码的蛋白氨基酸变异分析表明,二者的基因组均未出现基因重组、插入或缺失等显著变异,两个主要的功能蛋白的重要功能基序高度保守。但临床上GoCV与DuCV交叉跨种感染及共感染病例的出现无疑为WFCV的防控提出了更严峻的挑战,尤其是共感染的出现是否导致更复杂、更严重的临床发病还有待进一步研究。GoCV和DuCV相互间的交叉跨种感染已是一个不容忽视的问题,对WFCV病的诊断与防控造成了严峻考验。深入了解水禽中圆环病毒感染情况,对保障水禽健康养殖、制定科学防控策略具有重要意义。
4. 结论
本研究应用PCR技术对鹅群中水禽圆环病毒开展流行检测发现,当前鹅群中的GoCV和DuCV水禽圆环病毒流行较严重,呈现病原生态多样性,除检测到DuCV的跨种感染外,还发现首例GoCV和DuCV共感染病例。基因组序列分析表明该自然共感染病例中的GoCV和DuCV遗传稳定,未发生明显的遗传变异现象。研究结果将为今后深入开展WFCV跨种感染的分子机制研究及鹅WFCV病的防治策略制定提供科学依据。
-
表 1 收集的鹅组织样品信息
Table 1 Information on tissue specimens collected from geese farms
收集时间
Collection time组织样品来源与数量 Sample sources and number/份 合计 Total/份 福建 广西 安徽 广东 江西 浙江 2023年 28 21 25 10 8 7 99 2024年 19 13 5 8 5 4 54 合计 Total 47 34 30 18 13 11 153 -
[1] TODD D,WESTON J H,SOIKE D,et al. Genome sequence determinations and analyses of novel circoviruses from goose and pigeon[J]. Virology,2001,286(2) :354−362. DOI: 10.1006/viro.2001.0985
[2] SOIKE D,KOHLER B,ALBRECHT K. A circovirus-like infection in geese related to a runting syndrome[J]. Avian Pathology,1999,28(2) :199−202. DOI: 10.1080/03079459994939
[3] HATTERMANN K,SCHMITT C,SOIKE D,et al. Cloning and sequencing of duck circovirus (DuCV) [J]. Archives of Virology,2003,148(12) :2471−2480. DOI: 10.1007/s00705-003-0181-y
[4] STENZEL T,FARKAS K,VARSANI A. Genome sequence of a diverse goose circovirus recovered from greylag goose[J]. Genome Announcements,2015,3(4) :e00767−15.
[5] KOZDRUŃ W,WOŹNIAKOWSKI G,SAMOREK-SALAMONOWICZ E,et al. Viral infections in goose flocks in Poland[J]. Polish Journal of Veterinary Sciences,2012,15(3) :525−530. DOI: 10.2478/v10181-012-0080-9
[6] 万春和,施少华,程龙飞,等. 朗德鹅圆环病毒全基因组序列测定和遗传演化分析[J]. 中国动物传染病学报,2010,18(4) :6−12. DOI: 10.3969/j.issn.1674-6422.2010.04.002 WAN C H,SHI S H,CHENG L F,et al. Genomic cloning and sequence analysis of circovirus from Landes geese[J]. Chinese Journal of Animal Infectious Diseases,2010,18(4) :6−12. (in Chinese) DOI: 10.3969/j.issn.1674-6422.2010.04.002
[7] 余旭平,郑新添,何世成,等. 鹅圆环病毒浙江永康株全基因组的克隆及序列分析[J]. 微生物学报,2005,45(6) :860−864. DOI: 10.3321/j.issn:0001-6209.2005.06.009 YU X P,ZHENG X T,HE S C,et al. Cloning and analysis of the complete genome of a Goose circovirus from Yongkang Zhejiang[J]. Acta Microbiologica Sinica,2005,45(6) :860−864. (in Chinese) DOI: 10.3321/j.issn:0001-6209.2005.06.009
[8] SCOTT A N J,BECKETT A,SMYTH J A,et al. Serological diagnosis of goose circovirus infections[J]. Avian Pathology,2006,35(6) :495−499. DOI: 10.1080/03079450601087841
[9] CHEN C L,CHANG P C,LEE M S,et al. Nucleotide sequences of goose circovirus isolated in Taiwan[J]. Avian Pathology,2003,32(2) :165−171. DOI: 10.1080/0307945021000071614
[10] CHEN J D,WANG H,PEI H,et al. The prevalence,coinfection,and evolutionary and molecular characteristics of prevalent goose circovirus in Guangdong,China[J]. Avian Diseases,2021,65(4) :559−571.
[11] BANDA A,GALLOWAY-HASKINS R I,SANDHU T S,et al. Genetic analysis of a duck circovirus detected in commercial Pekin ducks in New York[J]. Avian Diseases,2007,51(1) :90−95. DOI: 10.1637/0005-2086(2007)051[0090:GAOADC]2.0.CO;2
[12] FRINGUELLI E,SCOTT A N J,BECKETT A,et al. Diagnosis of duck circovirus infections by conventional and real-time polymerase chain reaction tests[J]. Avian Pathology,2005,34(6) :495−500.
[13] CHEN C L,WANG P X,LEE M S,et al. Development of a polymerase chain reaction procedure for detection and differentiation of duck and goose circovirus[J]. Avian Diseases,2006,50(1) :92−95. DOI: 10.1637/7435-090705R1.1
[14] LEI X N,WANG A P,ZHU S Y,et al. From obscurity to urgency: A comprehensive analysis of the rising threat of duck circovirus[J]. Veterinary Research,2024,55(1) :12. DOI: 10.1186/s13567-024-01265-2
[15] 陈济铛,陈冈,牛森,等. 广东鹅源圆环病毒分子流行病学调查与毒株遗传进化分析[J]. 中国家禽,2020,42(4) :97−102. CHEN J D,CHEN G,NIU S,et al. Epidemiological investigation and genome analysis of goose circovirus in Guangdong of China[J]. China Poultry,2020,42(4) :97−102. (in Chinese)
[16] LIU H,LI L X,SUN W C,et al. Molecular survey of duck circovirus infection in poultry in southern and southwestern China during 2018 and 2019[J]. BMC Veterinary Research,2020,16(1) :80. DOI: 10.1186/s12917-020-02301-x
[17] TRAN G T H,MAI N T,BUI V N,et al. Duck circovirus in northern Vietnam: Genetic characterization and epidemiological analysis[J]. Archives of Virology,2022,167(9) :1871−1877. DOI: 10.1007/s00705-022-05501-y
[18] 赵光伟,谭雅文,李金凤,等. 重庆地区一例鹅感染鸭圆环病毒病的PCR诊断[J]. 畜禽业,2009,20(6) :66−68. DOI: 10.3969/j.issn.1008-0414.2009.06.042 ZHAO G W,TAN Y W,LI J F,et al. Diagnosis of duck circovirus infected the goose by PCR in Chongqing area[J]. Livestock and Poultry Industry,2009,20(6) :66−68. (in Chinese) DOI: 10.3969/j.issn.1008-0414.2009.06.042
[19] SHEHATA A A,GERRY D M,HEENEMANN K,et al. Goose parvovirus and circovirus coinfections in ornamental ducks[J]. Avian Diseases,2016,60(2) :516−522. DOI: 10.1637/11335-112615-Case
[20] XU S Q,MAN Y Z,XU X,et al. Genetic heterogeneity of duck circovirus first detected in geese from China[J]. Poultry Science,2024,103(12) :104284. DOI: 10.1016/j.psj.2024.104284
[21] XU S Q,MU X H,XU X,et al. First report of goose circovirus identified in ducks from China[J]. Poultry Science,2024,103(8) :103936. DOI: 10.1016/j.psj.2024.103936
[22] FU H R,ZHAO M,CHEN S Y,et al. Simultaneous detection and differentiation of DuCV-1 and DuCV-2 by high-resolution melting analysis[J]. Poultry Science,2024,103(4) :103566. DOI: 10.1016/j.psj.2024.103566
[23] GUO J,TIAN J,TAN X,et al. Pathological observations of an experimental infection of geese with goose circovirus[J]. Avian Pathology,2011,40(1) :55−61. DOI: 10.1080/03079457.2010.538371
[24] 万春和,黄瑜. 鹅圆环病毒研究进展[J]. 中国家禽,2015,37(9) :46−48 WAN C H,HUANG Y. Research progress in goose circovirus[J]. China Poultry,2015,37(9) :46−48. (in Chinese)
[25] 王梦鸽,黄瑜,傅秋玲,等. 鸭圆环病毒和鹅圆环病毒三引物鉴别检测方法的建立[J]. 福建农业学报,2023,38(1) :7−11. WANG M G,HUANG Y,FU Q L,et al. Establishment of a triple-primer multiplex PCR assay for discriminative detection of duck and goose circoviruses[J]. Fujian Journal of Agricultural Sciences,2023,38(1) :7−11. (in Chinese)
[26] CHEN C J,WU Y,LI J W,et al. TBtools-II: A “one for all,all for one” bioinformatics platform for biological big-data mining[J]. Molecular Plant,2023,16(11) :1733−1742. DOI: 10.1016/j.molp.2023.09.010
[27] TODD D. Circoviruses: Immunosuppressive threats to avian species: A review[J]. Avian Pathology,2000,29(5) :373−394. DOI: 10.1080/030794500750047126
[28] SOIKE D,ALBRECHT K,HATTERMANN K,et al. Novel circovirus in mulard ducks with developmental and feathering disorders[J]. Veterinary Record,2004,154(25) :792−793. DOI: 10.1136/vr.154.25.792
[29] 黄瑜,万春和,彭春香,等. 鸭圆环病毒感染的临床症状[J]. 中国家禽,2013,35(5) :47−48. DOI: 10.3969/j.issn.1004-6364.2013.05.013 HUANG Y,WAN C H,PENG C X,et al. Clinical symptoms of duck circovirus infection[J]. China Poultry,2013,35(5) :47−48. (in Chinese) DOI: 10.3969/j.issn.1004-6364.2013.05.013
[30] 傅光华,程龙飞,施少华,等. 鸭圆环病毒全基因组克隆与序列分析[J]. 病毒学报,2008,24(2) :138−143. DOI: 10.3321/j.issn:1000-8721.2008.02.010 FU G H,CHENG L F,SHI S H,et al. Genome cloning and sequence analysis of duck circovirus[J]. Chinese Journal of Virology,2008,24(2) :138−143. (in Chinese) DOI: 10.3321/j.issn:1000-8721.2008.02.010




 下载:
下载: