Identification and Expressions of PLT Family of Polygonatum cyrtonema
-
摘要:目的 PLT(Plethora)属于AP2/ERF型转录因子,在植物生长发育及抗胁迫等过程中发挥重要作用。本文旨在探究多花黄精PLT(Polygonatum cyrtonema Hua PLT,PcPLT)基因在根状茎器官生长发育中及盐胁迫下的调控作用,为深入研究PcPLT基因的功能提供理论依据。方法 基于多花黄精转录组数据库进行PcPLT基因家族鉴定及生物信息学分析,通过qRT-PCR技术检测其在多花黄精不同组织部位及不同浓度NaCl处理下的相对表达量,利用RT-PCR技术构建PcPLT2-2、PcPLT2-7基因融合表达载体,观察其荧光信号验证亚细胞定位。结果 共鉴定获得15个PcPLT家族成员,该家族无内含子结构,蛋白编码长度为159~601个氨基酸,均为亲水蛋白;进化树分析显示PcPLT与百合科石刁柏(Asparagus officinalis L.)亲缘关系最近;亚细胞定位预测及验证结果显示PcPLT2-2定位于细胞质、细胞核中,PcPLT2-7定位于细胞核中。qRT-PCR结果显示PcPLT2-3和PcPLT2-7在根状茎中表达最高,PcPLT1-3、PcPLT1-4、PcPLT2-7响应盐胁迫调控。结论 PcPLT不仅具有组织特异性还能提高抗逆性,提示可能作为器官发育调节因子参与多花黄精根状茎膨大的形态建成,为深入研究PLT调控植物根状茎器官生长发育的生物学功能和多花黄精遗传改良奠定基础。Abstract:Objective Regulatory functions of Polygonatum cyrtonema Hua PLT (PcPLT) in the growth and development of rhizomes of the plant were investigated.Methods Based on the P. cyrtonema transcriptome database, the identity and bioinformatics of PcPLT family were obtained. Relative expressions in tissues of the plant and that under NaCl stress were detected using the qRT-PCR technique. The fusion expression vectors of PcPLT2-2 and PcPLT2-7 were constructed, and their fluorescence signals examined to determine subcellular localization.Results Fifteen PcPLT family members were identified. They were hydrophilic proteins, absent of intron structure, coded 159-601 amino acids, and evolutionarily closely related to Liliaceae Asparagus officinalis L. The predicted subcellular localization of PcPLT2-2 was in cytoplasm and nucleus, while PcPLT2-7 in nucleus only. PcPLT2-3 and PcPLT2-7 mostly in the rhizomes; and PcPLT1-3, PcPLT1-4, and PcPLT2-7 responsive to salt stress.Conclusion PcPLT were tissue-specific and capable of enhancing the stress resistance of P. cyrtonema. They might act as an organ development regulator associated with the morphogenesis of rhizome expansion. If so, the result obtained in this study would be of value for the in-depth understanding of the biological functions of PLT.
-
Keywords:
- Polygonatum cyrtonema Hua /
- PLT /
- gene family /
- expression analysis
-
0. 引言
【研究意义】多花黄精(Polygonatum cyrtonema Hua)为百合科(Liliaceae)黄精属(Polygonatum)多年生草本植物。其根状茎肥厚,富含多糖、甾体皂苷、黄酮类等多种对人体有益物质,是我国传统药食同源的珍稀药材,具补气养阴、健润脾肺、益肾之功效,开发利用前景广阔[1-3]。多花黄精根状茎作为主要药用部位,生长缓慢,极易受到外界不良生长环境的影响,部分适宜生长地区土壤盐渍化日益严重逐年扩散,对多花黄精的生长和品质都有较大的影响,目前相关研究多局限于药理作用[4]、生理指标[5]、组织培养[6]等方面,对分子生物学方面的研究少之又少。因此对多花黄精根状茎生长发育及抗盐胁迫相关基因的深入研究具有非常重要的意义。【前人研究进展】转录因子(Transcription factor,TF)是能与真核基因启动子区域中顺式作用元件发生特异性结合,和下游靶基因顺式作用元件相互作用,进而调控靶基因表达的一类蛋白[7],在植物的生长发育、形态建成、抗逆及次生代谢物等方面具有重要的调控作用。Plethora(PLT)属于AP2/ERF型转录因子,含有两个 AP2 重复结构。PLT基因在胚胎发育、干细胞龛(静止中心,QC)、分生组织维护、器官生长发育等过程中具有核心作用[8];在盐胁迫、干旱胁迫、高温、低温胁迫等非生物胁迫条件下,调节相关功能基因的表达以提高抗逆性[9]。QC是干细胞的组织中心,植物干细胞具有自我更新和持续分裂子细胞的能力,是植物根、茎、叶和花等器官发生的源泉。PLT1 和 PLT2是干细胞维持静止中心所必需的两个基因,静止中心的正常形成要求PLT1、PLT2 、SHORT-ROOT(SHR)和SCARECROW(SCR )共同参与,PLT、SCR、植物特异性转录因子TCP的3个基因互作形成 PLT-TCP-SCR 复合体,与WOX5 启动子中的 PLT 结合位点结合进行诱导表达,在胚胎发生过程中特化干细胞龛,并在胚胎后维持干细胞属性[8,10]。PLT基因与生长素关联密切,参与较多的基因调控网络,转录响应生长素积累并依赖于生长素响应转录因子进行转录,通过生长素的局部合成[11]及PIN蛋白[12]作为介导定向运输转运生长素在QC 中形成了浓度梯度[13-14],作用于下游的PLT基因,形成一个调控网络来维持稳定的生长素浓度和根尖干细胞龛的位置[15],对根分生组织的大小和维持至关重要[16]。PLT3、PLT5 和 PLT7调节侧根原基的启动及建立正确的对称生长轴中基因表达程序[17-18]。Kareem等[19]揭示了PLT 控制不定芽再生的机制:第一步,PLT3、PLT5 和 PLT7 通过 PLT1 和 PLT2激活根干细胞建立多能性;第二步,PLT3、PLT5 和 PLT7通过增强因子 Cupshaped cotyledon2(CUC2)调节来完成芽形成。PLT活性高能维持干细胞活性,PLT活性低有利于干细胞进行有丝分裂,PLT活性更低则促进细胞分化[8]。PLT基因在根干细胞区域和不定芽的生成过程中都表现出特异性表达[19]。PLT基因不仅能响应种子萌发、生根等器官发育,响应激素信号,还能调控非生物胁迫[9,20]。【本研究切入点】PLT基因家族的功能研究大多集中在模式植物拟南芥(Arabidopsis thaliana L.)和水稻(Oryza sativa L.)中,且主要集中在根系中,目前国内有关PLT基因家族的研究较少,在多花黄精中更是未见报道,PLT基因家族是否调控多花黄精生长发育及行使相关功能还未曾揭示。【拟解决的关键问题】以实验室前期获得的多花黄精不同组织部位转录组数据为基础,对多花黄精PLT家族进行生物信息学分析并对关键成员进行克隆,测定其在不同组织部位和调控盐胁迫方面的表达情况,以期为多花黄精PLT家族基因的生物学功能研究、多花黄精遗传改良奠定基础。
1. 材料与方法
1.1 材料
野生植株不同组织部位(果实、叶片、根、茎秆、根状茎)转录组数据材料由采自福建省邵武市大竹镇(北纬27°11′6.80″,东经117°35′4.60″)野生多花黄精测序获得(另文发表),盐胁迫转录组数据由多花黄精组培苗经不同浓度NaCl处理24 h后采样测序获得。
1.2 方法
1.2.1 总RNA的提取及cDNA的合成
参考RNAprep Pure总RNA提取试剂盒(TIANGEN)说明书提取多花黄精样品总RNA,用Thermo超微量核酸检测仪测定RNA浓度,通过1%琼脂糖凝胶电泳检测RNA纯度,然后采用All supeiMix(TransGen Biotech公司)和GeneRacerTM试剂盒(TaKaRa)逆转录成cDNA用于PLT基因定量和克隆,用SYBR Green Master Mix(No Rox)进行qRT-PCR反应。
1.2.2 多花黄精 PLT 基因家族的鉴定
从 Pfam 数据库(http://pfam.xfam.org/)获取 PLETHORA(PF00847)结构域的隐马尔可夫模型文件,使用 TBtools软件对多花黄精转录组数据库进行搜索。使用 CDD(https://www.ncbi.nlm.nih.gov/Structure/bwrpsb/bwrpsb.cgi)和SMART(http://smart.embl-heidelberg.de/)对候选序列进行保守结构域鉴定和筛选。采用在线网站 ExPASy(https://web.expasy.org/protparam/)分析多花黄精PLT成员的氨基酸数量、蛋白分子量、等电点、不稳定指数和亲水性。利用SignalP 4.1 Server(https://services.healthtech.dtu.dk/service.php?SignalP)、TMHMM Server 2.0(https://services.healthtech.dtu.dk/service.php?TMHMM-2.0)、 Cell-PLoc 2.0(http://www.csbio.sjtu.edu.cn/bioinf/Cell-PLoc-2/)、SOPMA(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)在线网站进行 PcPLT 蛋白的信号肽、跨膜结构、亚细胞定位和二级结构的分析与预测。
1.2.3 多花黄精 PLT 家族基因结构、保守基序和保守结构域分析
运用 GSDS(http://gsds.gao-lab.org/)在线网站分析 PcPLT基因结构。使用 MEME 在线软件(https://meme-suite.org/meme/tools/meme)分析该家族成员编码蛋白的保守基序。同时结合结构域分析结果,使用 TBtools 绘制基因结构、保守基序和保守结构域图。
1.2.4 PcPLT 家族系统进化关系分析
模式植物拟南芥(Arabidopsis thaliana)的 PLT 家族蛋白序列下载自 TAIR 数据库(https://www.arabidopsis.org),水稻(Oryza sative L.)的PLT 家族蛋白序列下载自 RiceData 数据库(http://www.ricedata.cn/gene),从NCBI [National Center for Biotechnology Information (nih.gov)]下载石刁柏(Asparagus officinalis L.)、油棕(Elaeis guineensis)、葡萄(Vitis vinifera)蛋白序列。使用 MEGA 11.0 软件中的最大似然法(Maximum likelihood,ML)构建系统发育树,并使用在线软件 iTOL(https://itol.embl.de/upload.cgi)对进化树进行美化。
1.2.5 PcPLT 家族GO功能注释
为了解到PcPLT 全部基因信息,进行GO数据库功能注释搜索,分析注释信息。
1.2.6 多花黄精PLT基因特异表达分析
为进一步了解PLT 基因在多花黄精不同组织器官中的表达模式,通过qRT-PCR检测PLT 基因在不同组织部位以及NaCl不同浓度处理下的表达情况。根据HieffTM qPCR SYBR Green Master Mix(YEASEN)说明书进行qRT-PCR反应。 反应体系为20 μL,包括Mix 10 μL,ddH2O 7.2 μL,cDNA 模版2 μL,上、下引物各0.4 μL。内参基因选用β-tubulin基因,依据2−△△Ct法分析计算PcPLT基因的相对表达量,并使用GraphPad Prism进行图表绘制。分析多花黄精PLT基因在不同组织器官中及不同浓度NaCl处理下的表达情况(引物序列见表1)。
表 1 引物序列Table 1. Sequences of primers used引物名称
Primer name引物序列
Primer sequence (5′-3′)退火温度Tm/℃ 用途 Usage PLT2-2-Q-F
PLT2-2-Q-RCGTCATCTTATCGTGGAGTTAC
CATCTTTCCGCCTTGTTACT55 qRT-PCR引物 PLT2-7-Q-F
PLT2-7-Q-RTCAAGTGTAATGGGAGGGA
AGAGAATAGCGGCAATGTTG55 qRT-PCR引物 β-tubulin-Q-F
β-tubulin-Q-RTTGTCGAAAATGCTGACGAG
CAAGCTTCCGGAGATCAGAG60 内参基因引物 PLT2-2-GFP-F
PLT2-2-GFP-RCATGCCATGGATGAAGAAGTTTGCGGTGCC
GGACTAGTTAACTCAAGTTTGCAGGGTT60 载体构建、亚细胞定位 PLT2-7-GFP-F
PLT2-7-GFP-RCATGCCATGGATGGGGAACTTGACCAAG
GGACTAGTGCTCCTGGGGTAGAAATAGG64 载体构建、亚细胞定位 PLT1-3-Q-F
PLT1-3-Q-RTACCTCCTCCACCACTTCTC
TCTTCAAATCAGAGTCCGC60 qRT-PCR引物 PLT1-4-Q-F
PLT1-4-Q-RATGGTGAGTGCTTGGCTT
TGCCTCCCTTTCCTTGTT50 qRT-PCR引物 1.2.7 过表达载体的构建及亚细胞定位
将PcPLT2-2、PcPLT2-7的CDS序列导入DNAMAN 7.0 软件中,对其进行pCAM-BIA1302的酶切位点预测,本试验选择Nco Ⅰ和Spe I 酶切位点进行后续试验。经扩增、胶回收、连接后将连接产物导入DH5α大肠杆菌感受态细胞,挑取单菌落摇菌后送测,提取重组质粒pCAMBIA1302-目的片段GFP。将带目的片段的重组质粒与pCAMBIA1302植物表达载体分别进行酶切,并回收。采用冻融法转化根癌农杆菌3101,然后采用农杆菌介导法侵染洋葱内表皮PLT进行蛋白亚细胞定位,洋葱37 ℃暗培养3 d后取表皮进行观察(引物序列见表1)。
2. 结果与分析
2.1 多花黄精PLT家族成员鉴定
利用PLT结构域对多花黄精不同组织部位转录组进行搜索筛选,最终鉴定出15个PLT家族成员,依次命名为PcPLT1-1–PcPLT1-4、PcPLT2-1–PcPLT2-11(表2)。PcPLT编码长度159~601 aa、分子量17827.93 Da(PcPLT2-4)~66506.19(PcPLT2-1)、等电点(pI)为6.47~9.64的蛋白质。所有蛋白的不稳定系数都大于 40,表明均为不稳定蛋白;亲水性指数都为负值,表明均为亲水蛋白。信号肽预测结果显示均不含信号肽且15个成员中都不具有跨膜结构。亚细胞定位显示, PcPLT1-4、PcPLT2-1、PcPLT2-2、PcPLT2-5、PcPLT2-8、PcPLT2-9、PcPLT2-10定位在细胞质、细胞核中,PcPLT1-1、PcPLT1-2、PcPLT1-3、PcPLT2-3、PcPLT2-4、PcPLT2-6、PcPLT2-7、PcPLT2-11定位在细胞核中。通过在线软件 SOPMA 分析预测多花黄精 PLT 蛋白序列的二级结构,发现多花黄精 PLT 家族蛋白中大部分成员无规则卷曲结构所占比例高,β-转角结构所占比例最低,所有 α-螺旋结构所占比例都高于延伸链结构。
表 2 多花黄精PLT 家族成员信息Table 2. Information on PcPLT family members基因名称
Gene name基因ID
Gene ID注释到
拟南芥成员
Annotated
Arabidopsis氨基酸数量
Amino
acids/aa分子量
Molecular
weight/Da等电点
pI脂肪系数
Aliphatic
index二级结构
Secondary structure/%亚细胞定位
Subcellular
localizationα-螺旋
Alpha
helixβ-转角
Beta
turn延伸链
Extended
strand无规卷曲
Random
coilPcPLT1-1 >Cluster-11683.110576.p1 AtPLT1 317 35195.85 8.96 54.64 32.18 5.36 10.73 51.74 细胞核 PcPLT1-2 >Cluster-11683.110575.p1 AtPLT1 469 51308.34 6.47 53.41 25.59 5.33 12.15 56.93 细胞核 PcPLT1-3 >Cluster-11683.173509.p1 AtPLT1 468 51236.27 6.47 52.88 25.21 5.13 12.61 57.05 细胞核 PcPLT1-4 >Cluster-11683.233671.p1 AtPLT1 421 47120.43 7.71 58.24 24.47 7.84 13.78 53.92 细胞质、细胞核 PcPLT2-1 >Cluster-11683.222146.p1 AtPLT2 601 66506.19 6.51 59.63 18.47 3.99 12.48 65.06 细胞质、细胞核 PcPLT2-2 >Cluster-11683.100274.p1 AtPLT2 277 30714.02 7.25 61.66 34.30 7.22 15.52 42.96 细胞质、细胞核 PcPLT2-3 >Cluster-11683.77849.p1 AtPLT2 180 20995.90 9.64 70.94 33.89 7.78 26.11 32.22 细胞核 PcPLT2-4 >Cluster-11683.163200.p1 AtPLT2 159 17827.93 6.48 67.61 42.77 5.66 16.35 35.22 细胞核 PcPLT2-5 >Cluster-11683.105355.p1 AtPLT2 312 34191.20 6.94 73.91 26.92 6.73 13.14 53.21 细胞质、细胞核 PcPLT2-6 >Cluster-11683.151415.p2 AtPLT2 199 22672.77 9.11 74.02 39.70 7.54 22.61 30.15 细胞核 PcPLT2-7 >Cluster-11683.204809.p1 AtPLT2 332 36898.44 9.11 61.84 25.00 5.12 12.65 57.23 细胞核 PcPLT2-8 >Cluster-11683.147730.p1 AtPLT2 395 44135.24 9.07 63.01 27.59 6.84 15.95 49.62 细胞质、细胞核 PcPLT2-9 >Cluster-11683.151416.p1 AtPLT2 391 43773.69 8.89 59.64 24.55 7.42 13.81 54.22 细胞质、细胞核 PcPLT2-10 >Cluster-11683.133871.p1 AtPLT2 380 42322.23 9.27 61.89 24.47 7.37 13.68 54.47 细胞质、细胞核 PcPLT2-11 >Cluster-11683.182808.p2 AtPLT2 184 20988.80 9.26 69.46 39.67 7.07 18.48 34.78 细胞核 2.2 多花黄精PLT基因结构、保守结构域及保守基序分析
为进一步揭示 PcPLT的结构和功能特性,采用在线网站 MEME 对多花黄精 PLT 蛋白保守基序进行分析,共鉴定到 10 个保守基序,分别命名为 motif1–motif10(图1),每个蛋白序列上分布的基序种类和顺序有一定差异性,揭示了功能的多样性,其中除PcPLT2-11外,其他成员均含有motif1、motif2、motif3、motif5,具有高度保守性;所有成员均含有2个AP2结构域且无内含子。
2.3 多花黄精 PLT 家族系统进化分析
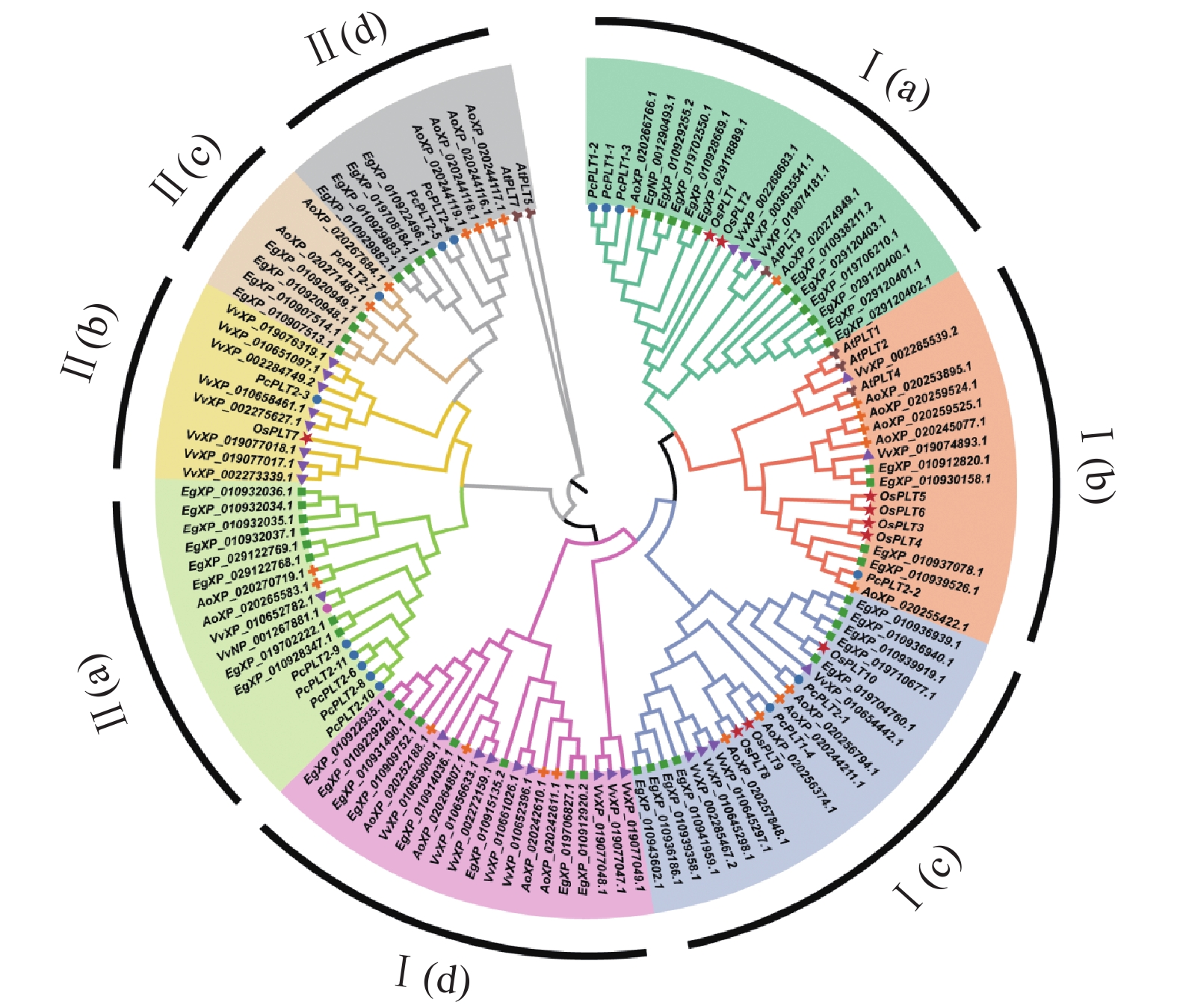
为分析多花黄精PLT家族的进化关系,参考水稻、拟南芥、石刁柏、油棕和葡萄PLT成员氨基酸序列构建系统进化树(图2)。参考水稻PLT家族的进化分类,将多花黄精PLT家族分为2个大类(Ⅰ和Ⅱ)8个亚家族[Ⅰ(a)、Ⅰ(b)、Ⅰ(c)、Ⅰ(d)、Ⅱ(a)、Ⅱ(b)、Ⅱ(c)、Ⅱ(d)],两个大类各包含6和9个多花黄精成员,进化树显示多花黄精与石刁柏亲缘关系较近,油棕其次,推测是因为多花黄精与石刁柏都属于百合科植物,具有一定相同的序列,而与拟南芥亲缘关系最远,可能是拟南芥是双子叶植物,多花黄精是单子叶植物,因此同源性较其他单子叶植物更远,这有利于进一步探索PcPLT的生物学功能。
2.4 多花黄精PLT基因家族的不同组织部位表达分析
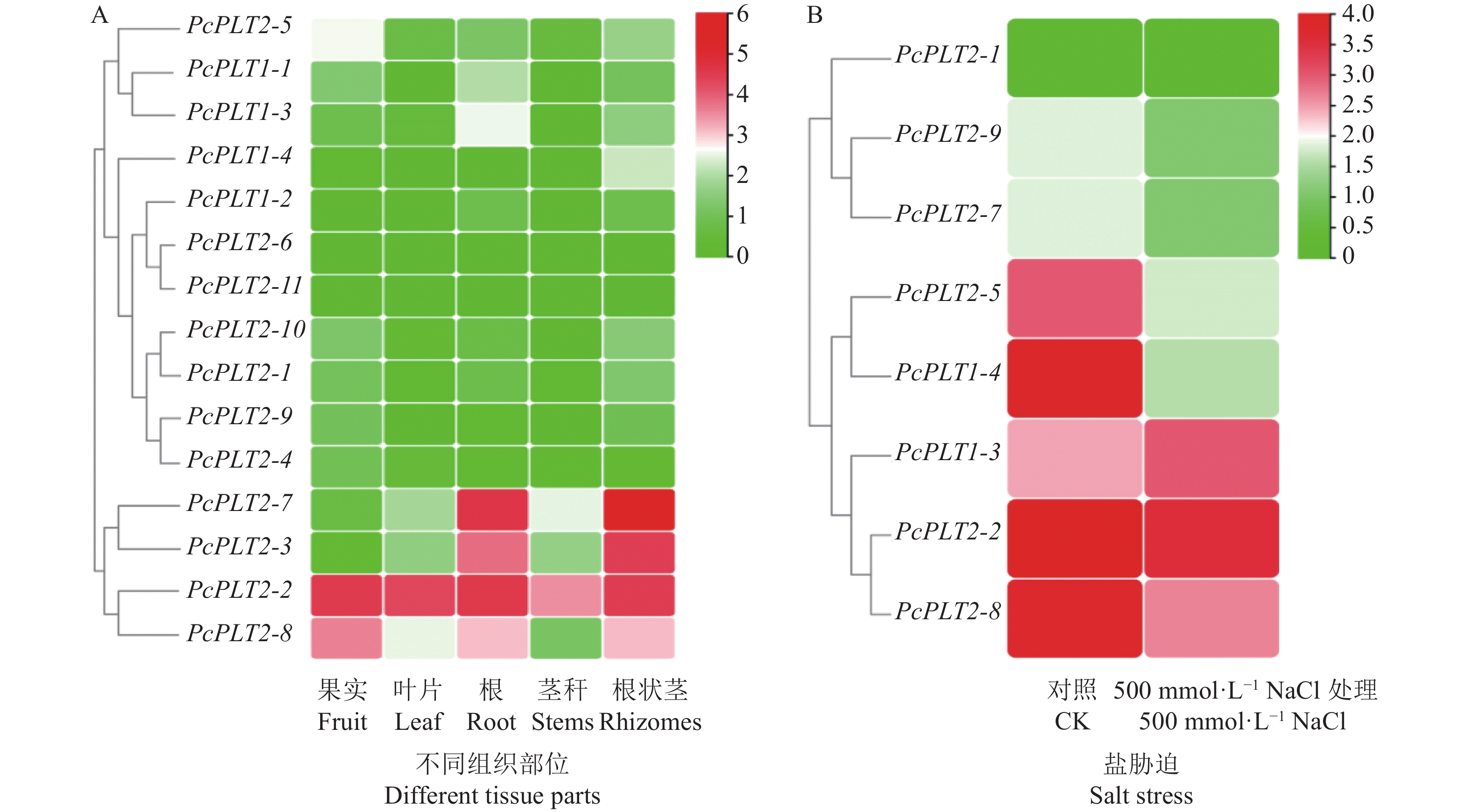
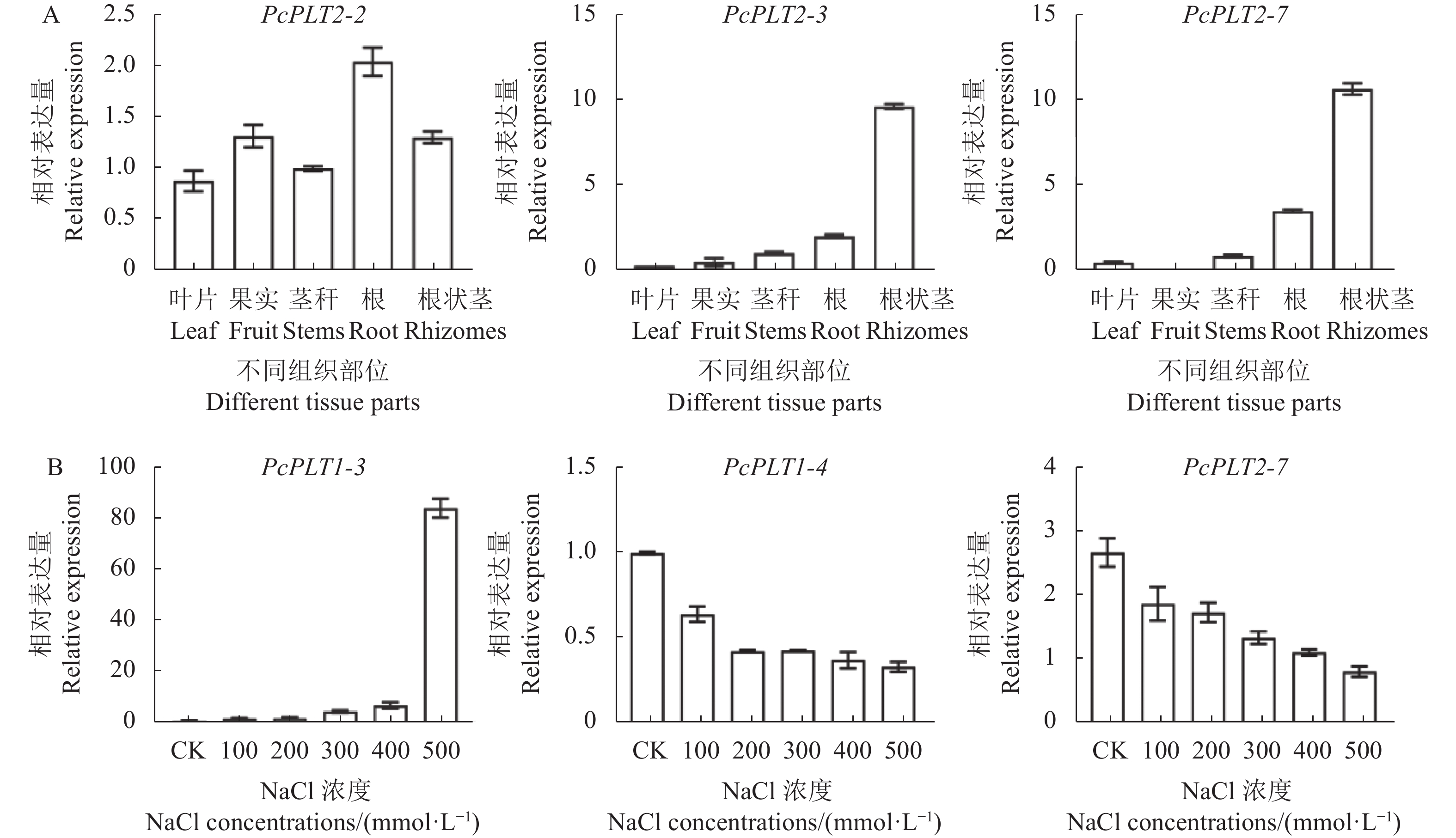
为了探究PLT家族基因不同组织部位的表达差异,使用多花黄精不同组织部位和盐胁迫转录组中PLT家族基因的表达量(FPKM)绘制热图(图3)。不同组织部位转录组结果(图3A)显示,15个PcPLT基因在不同组织部位中均有不同程度的表达,PcPLT2-7、PcPLT2-3、PcPLT2-2、PcPLT2-8在根状茎和根中有较高的表达量,PcPLT2-2和PcPLT2-8在所有部位表达量都较高,PcPLT2-3和PcPLT2-7在根状茎里的表达量最高。根据不同部位转录组筛选到的PcPLT成员在盐胁迫转录组中进行比对,共比对到8个成员(图3B),在500 mmol·L−1 NaCl处理下PcPLT1-3有明显上调表达,PcPLT2-2、PcPLT2-8、PcPLT1-4、PcPLT2-5和PcPLT2-9相比较于CK都有不同程度的下调表达。选取PcPLT2-2、PcPLT2-3、PcPLT2-7进行不同组织部位的表达量分析,选取PcPLT1-3、PcPLT1-4、PcPLT2-7进行不同浓度盐胁迫的qRT-PCR表达分析。结果表明(图4):PcPLT2-2、PcPLT2-3、PcPLT2-7在多花黄精各个组织部位均有表达,以茎秆为CK进行表达量计算,其中PcPLT2-2在根的表达量最高,在叶的表达量最低;PcPLT2-3在根状茎的表达量最高,在叶的表达量最低,根状茎的表达量是叶的45倍;PcPLT2-7在根状茎的表达量最高,在果实的表达量最低,根状茎的表达量是果实的135倍,推测PcPLT基因在根状茎和根中行使重要功能。PcPLT1-3在不同浓度NaCl处理下呈现出上调表达,在500 mmol·L−1 NaCl时表达量最高;PcPLT1-4和PcPLT2-7随着NaCl浓度的提高相对表达量表现出下降的趋势,在500 mmol·L−1 NaCl表达量最低,表明PcPLT1-3正调控盐胁迫,PcPLT1-4、PcPLT2-7负调控盐胁迫。
2.5 多花黄精PcPLT2-2、PcPLT2-7的载体构建及亚细胞定位
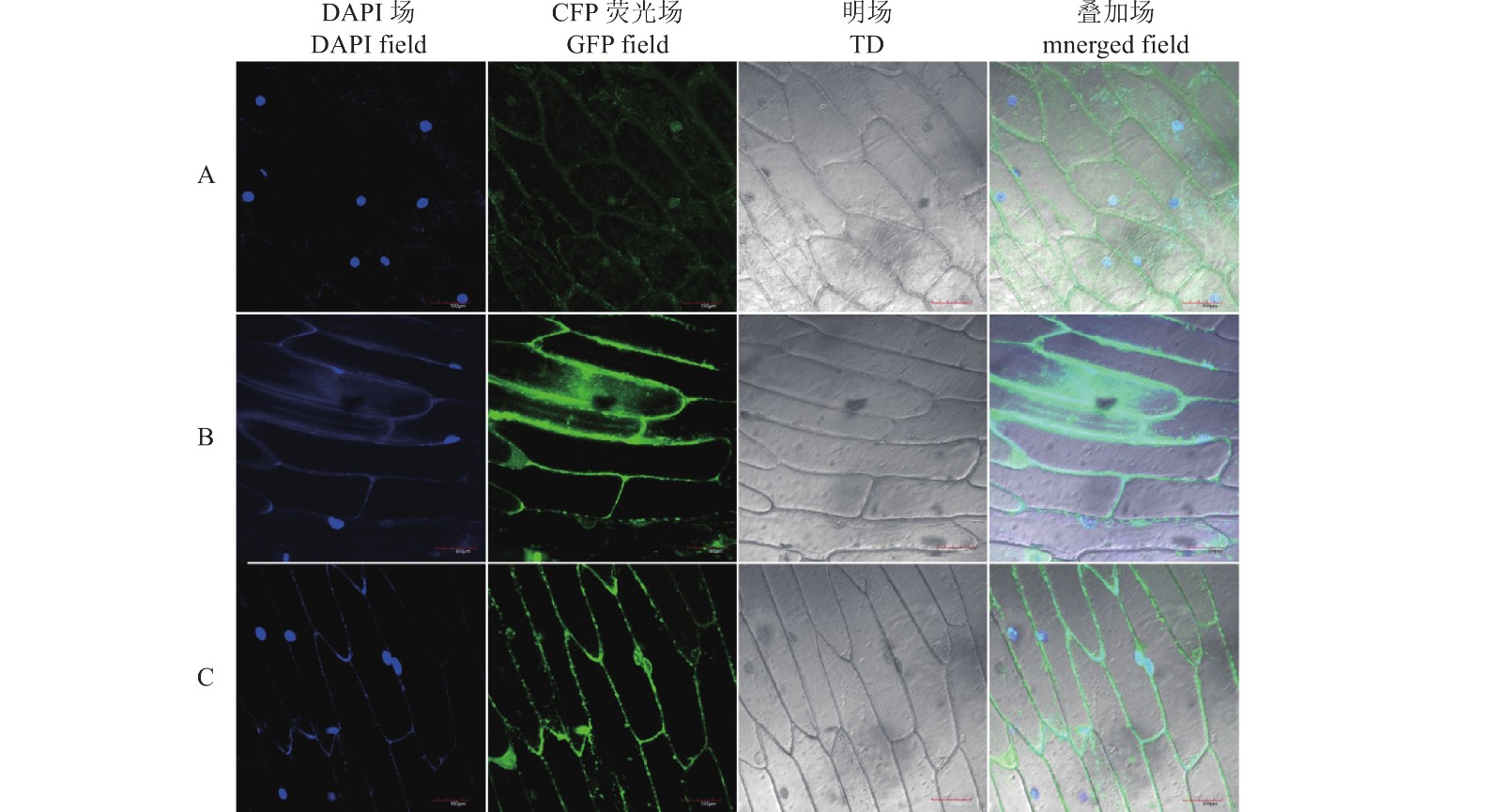
为进一步研究PcPLT基因家族亚细胞定位,将构建好的pCAMBIA1302-PcPLT-GFP载体进行洋葱注射,洋葱亚细胞定位结果显示(图5):1302空载GFP充斥在洋葱整个细胞中,1302-PcPLT2-2-GFP定位在细胞质、细胞核,1302-PcPLT2-7-GFP定位在细胞核,均与亚细胞定位预测结果一致。
![]() 图 5 PcPLT蛋白在洋葱表皮中的亚细胞定位A:pCAMBIA1302-GFP荧光信号;B:pCAMBIA1302-PcPLT2-2-GFP荧光信号;C:pCAMBIA1302-PcPLT2-7-GFP荧光信号。Figure 5. Subcellular localization of PcPLT in onion epidermal cellsA: pCAMBIA1302-GFP fluorescence signal; B: pCAMBIA1302-PcPLT2-2-GFP fluorescence signal; C: pCAMBIA1302-PcPLT2-7-GFP fluorescence signal.
图 5 PcPLT蛋白在洋葱表皮中的亚细胞定位A:pCAMBIA1302-GFP荧光信号;B:pCAMBIA1302-PcPLT2-2-GFP荧光信号;C:pCAMBIA1302-PcPLT2-7-GFP荧光信号。Figure 5. Subcellular localization of PcPLT in onion epidermal cellsA: pCAMBIA1302-GFP fluorescence signal; B: pCAMBIA1302-PcPLT2-2-GFP fluorescence signal; C: pCAMBIA1302-PcPLT2-7-GFP fluorescence signal.3. 讨论与结论
3.1
多花黄精PcPLT家族成员鉴定与进化特性 PLT 作为AP2/ERF型转录因子,在植物体各个组织中广泛表达,尤其在幼嫩的组织中表达量高。已被证明具有促进根部分生组织生长、芽从头再生、激活根部干细胞建立全能性、茎尖分生组织定位、启动花序分生组织原基等功能,通过与其他转录因子及生长素进行互作影响植物器官的发育与形态建成,对多花黄精遗传改良和增产具有重要意义。
本研究通过生物信息学方法从多花黄精不同组织部位转录组信息鉴定到15个PLT成员,该家族成员均为不稳定亲水蛋白,不含信号肽且不具有跨膜结构,无内含子。系统进化关系表明,PcPLT与石刁柏和油棕的亲缘关系最近,通过在线软件 SOPMA 分析预测多花黄精 PLT 蛋白序列的二级结构,发现多花黄精 PLT 家族蛋白中大部分成员都是无α-螺旋结构和延伸链结构占比较多。蛋白功能的发挥与所在位置关联密切,只有被运输到合适位置才能参与生命活动行使功能,蛋白的亚细胞定位与其蛋白的功能密不可分,因此研究亚细胞定位是研究蛋白功能的一种手段[21]。细胞核是细胞遗传和代谢的调控中心,控制着细胞的分裂和死亡,在细胞的代谢、生长、分化中起着重要作用,PcPLT2-2、PcPLT2-7亚细胞定位结果表明,PcPLT2-2定位在细胞质、细胞核中,PcPLT2-7定位在细胞核中,这与预测结果一致,由此推测 PLT基因在多花黄精生长发育中有重要作用。
3.2 PcPLT可能促进多花黄精的生长发育及参与生长素的调控
Nole-Wilson等[22]研究表明,PLT基因在新生幼嫩组织中表达量更高,而多花黄精不同组织部位qRT-PCR表明,PcPLT2-3、PcPLT2-7基因在根状茎中表达量最高,因此推测PLT基因可能在根状茎新生组织中高表达,更有可能促进多花黄精根状茎的生长发育。生长素是一类重要的植物激素,参与调控植物生长发育等众多生物学过程,尤其在调控根系生长、侧根发育等过程中发挥着不可替代的作用,参与根干细胞龛的形态建成。在拟南芥中,生长素合成基因YUC3和YUC8是PLT2激活靶基因[23],芽中的PLT5-GR在DEX存在的情况下能调控YUC5 表达,PLT与生长素之间相互激活,PLT蛋白通过激活YUC促进生长素积累[24]。不同PLT基因成员与生长素早期反应的共表达模式不同,其中与PLT2和PLT5共表达的生长素早期反应基因明显多于PLT1和PLT7,而与PLT4/BBM共表达的生长素早期反应基因仅为少数。PLT在调节器官发育的细胞分裂和分化阶段中发挥作用的分子机制可能涉及根和地上部的不同靶基因,这可能是由于这些器官系统的不同结构和生长策略。需要对拟南芥和其他物种的根和芽中的AIL/PLT基因调控网络进行更详细的分析,以阐明生长调节剂是如何在不同的发育环境中进化定位。
3.3 PcPLT可能参与多花黄精非生物胁迫的调控
植物在受到非生物胁迫时,会通过升高体内脱落酸(ABA )水平进行响应,这一途径已被充分报道[25]。宋婷婷等[9]通过对水曲柳进行低温(4 ℃)、NaCl、NaHCO3 和 PEG6000 处理,利用qRT-PCR检测PLT基因的表达情况,结果表明均有明显响应,证明PLT基因积极参与植物非生物胁迫耐受性的调控;本研究通过不同浓度NaCl处理表明, PcPLT1-3 的表达量随着NaCl浓度的提高出现上升的趋势, PcPLT1-4 和 PcPLT2-7 则表现出下调,初步说明 PcPLT 调控盐胁迫,因此推测PLT基因在多花黄精中同样存在以上功能,但如何响应或参与非生物胁迫体调控的相关机理尚未明晰。
-
图 5 PcPLT蛋白在洋葱表皮中的亚细胞定位
A:pCAMBIA1302-GFP荧光信号;B:pCAMBIA1302-PcPLT2-2-GFP荧光信号;C:pCAMBIA1302-PcPLT2-7-GFP荧光信号。
Figure 5. Subcellular localization of PcPLT in onion epidermal cells
A: pCAMBIA1302-GFP fluorescence signal; B: pCAMBIA1302-PcPLT2-2-GFP fluorescence signal; C: pCAMBIA1302-PcPLT2-7-GFP fluorescence signal.
表 1 引物序列
Table 1 Sequences of primers used
引物名称
Primer name引物序列
Primer sequence (5′-3′)退火温度Tm/℃ 用途 Usage PLT2-2-Q-F
PLT2-2-Q-RCGTCATCTTATCGTGGAGTTAC
CATCTTTCCGCCTTGTTACT55 qRT-PCR引物 PLT2-7-Q-F
PLT2-7-Q-RTCAAGTGTAATGGGAGGGA
AGAGAATAGCGGCAATGTTG55 qRT-PCR引物 β-tubulin-Q-F
β-tubulin-Q-RTTGTCGAAAATGCTGACGAG
CAAGCTTCCGGAGATCAGAG60 内参基因引物 PLT2-2-GFP-F
PLT2-2-GFP-RCATGCCATGGATGAAGAAGTTTGCGGTGCC
GGACTAGTTAACTCAAGTTTGCAGGGTT60 载体构建、亚细胞定位 PLT2-7-GFP-F
PLT2-7-GFP-RCATGCCATGGATGGGGAACTTGACCAAG
GGACTAGTGCTCCTGGGGTAGAAATAGG64 载体构建、亚细胞定位 PLT1-3-Q-F
PLT1-3-Q-RTACCTCCTCCACCACTTCTC
TCTTCAAATCAGAGTCCGC60 qRT-PCR引物 PLT1-4-Q-F
PLT1-4-Q-RATGGTGAGTGCTTGGCTT
TGCCTCCCTTTCCTTGTT50 qRT-PCR引物 表 2 多花黄精PLT 家族成员信息
Table 2 Information on PcPLT family members
基因名称
Gene name基因ID
Gene ID注释到
拟南芥成员
Annotated
Arabidopsis氨基酸数量
Amino
acids/aa分子量
Molecular
weight/Da等电点
pI脂肪系数
Aliphatic
index二级结构
Secondary structure/%亚细胞定位
Subcellular
localizationα-螺旋
Alpha
helixβ-转角
Beta
turn延伸链
Extended
strand无规卷曲
Random
coilPcPLT1-1 >Cluster-11683.110576.p1 AtPLT1 317 35195.85 8.96 54.64 32.18 5.36 10.73 51.74 细胞核 PcPLT1-2 >Cluster-11683.110575.p1 AtPLT1 469 51308.34 6.47 53.41 25.59 5.33 12.15 56.93 细胞核 PcPLT1-3 >Cluster-11683.173509.p1 AtPLT1 468 51236.27 6.47 52.88 25.21 5.13 12.61 57.05 细胞核 PcPLT1-4 >Cluster-11683.233671.p1 AtPLT1 421 47120.43 7.71 58.24 24.47 7.84 13.78 53.92 细胞质、细胞核 PcPLT2-1 >Cluster-11683.222146.p1 AtPLT2 601 66506.19 6.51 59.63 18.47 3.99 12.48 65.06 细胞质、细胞核 PcPLT2-2 >Cluster-11683.100274.p1 AtPLT2 277 30714.02 7.25 61.66 34.30 7.22 15.52 42.96 细胞质、细胞核 PcPLT2-3 >Cluster-11683.77849.p1 AtPLT2 180 20995.90 9.64 70.94 33.89 7.78 26.11 32.22 细胞核 PcPLT2-4 >Cluster-11683.163200.p1 AtPLT2 159 17827.93 6.48 67.61 42.77 5.66 16.35 35.22 细胞核 PcPLT2-5 >Cluster-11683.105355.p1 AtPLT2 312 34191.20 6.94 73.91 26.92 6.73 13.14 53.21 细胞质、细胞核 PcPLT2-6 >Cluster-11683.151415.p2 AtPLT2 199 22672.77 9.11 74.02 39.70 7.54 22.61 30.15 细胞核 PcPLT2-7 >Cluster-11683.204809.p1 AtPLT2 332 36898.44 9.11 61.84 25.00 5.12 12.65 57.23 细胞核 PcPLT2-8 >Cluster-11683.147730.p1 AtPLT2 395 44135.24 9.07 63.01 27.59 6.84 15.95 49.62 细胞质、细胞核 PcPLT2-9 >Cluster-11683.151416.p1 AtPLT2 391 43773.69 8.89 59.64 24.55 7.42 13.81 54.22 细胞质、细胞核 PcPLT2-10 >Cluster-11683.133871.p1 AtPLT2 380 42322.23 9.27 61.89 24.47 7.37 13.68 54.47 细胞质、细胞核 PcPLT2-11 >Cluster-11683.182808.p2 AtPLT2 184 20988.80 9.26 69.46 39.67 7.07 18.48 34.78 细胞核 -
[1] 国家药典委员会. 中华人民共和国药典: 二部 (2020年版)[M]. 北京: 中国医药科技出版社, 2020. [2] 张娇, 王元忠, 杨维泽, 等. 黄精属植物化学成分及药理活性研究进展 [J]. 中国中药杂志, 2019, 44(10):1989−2008. ZHANG J, WANG Y Z, YANG W Z, et al. Research progress in chemical constituents in plants of Polygonatum a their pharmacological effects [J]. China Journal of Chinese Materia Medica, 2019, 44(10): 1989−2008.(in Chinese)
[3] TANG C, YU Y M, QI Q L, et al. Steroidal saponins from the rhizome of Polygonatum sibiricum [J]. Journal of Asian Natural Products Research, 2019, 21(3): 197−206. DOI: 10.1080/10286020.2018.1478-815
[4] 单春苗, 王晨凯, 施圆圆, 等. 多花黄精甾体皂苷生物合成途径分析及关键酶基因研究 [J]. 中国中药杂志, 2020, 45(12):2847−2857. SHAN C M, WANG C K, SHI Y Y, et al. Identification of key enzyme genes involved in biosynthesis of steroidal saponins and analysis of biosynthesis pathway in Polygonatum cyrtonema [J]. China Journal of Chinese Materia Medica, 2020, 45(12): 2847−2857.(in Chinese)
[5] 童龙, 张磊, 高勇军, 等. 不同遮阴处理对多花黄精生理生长的影响 [J]. 西南林业大学学报(自然科学), 2020, 40(3):68−75. TONG L, ZHANG L, GAO Y J, et al. Growth and physiological characteristics of Polygonatum cyrtonema under different shading treatments [J]. Journal of Southwest Forestry University (Natural Sciences), 2020, 40(3): 68−75.(in Chinese)
[6] 刘剑东, 幸菲菲, 彭思静, 等. 多花黄精丛生芽的诱导与增殖条件 [J]. 植物生理学报, 2020, 56(6):1277−1285. LIU J D, XING F F, PENG S J, et al. The conditions for induction and proliferation on cluster buds of Polygonatum cyrtonema [J]. Plant Physiology Journal, 2020, 56(6): 1277−1285.(in Chinese)
[7] MAO H D, YU L J, HAN R, et al. ZmNAC55, a maize stress-responsive NAC transcription factor, confers drought resistance in transgenic Arabidopsis [J]. Plant Physiology and Biochemistry, 2016, 105: 55−66. DOI: 10.1016/j.plaphy.2016.04.018
[8] AIDA M, BEIS D, HEIDSTRA R, et al. The PLETHORA genes mediate patterning of the Arabidopsis root stem cell niche [J]. Cell, 2004, 119(1): 109−120. DOI: 10.1016/j.cell.2004.09.018
[9] 宋婷婷, 梁楠松, 吕义品, 等. 水曲柳FmPLT基因家族的鉴定及表达分析 [J]. 北京林业大学学报, 2022, 44(2):11−21. SONG T T, LIANG N S, LÜ Y P, et al. Identification and expression analysis of FmPLT gene family of Fraxinus mandschurica [J]. Journal of Beijing Forestry University, 2022, 44(2): 11−21.(in Chinese)
[10] GALINHA C, HOFHUIS H, LUIJTEN M, et al. PLETHORA proteins as dose-dependent master regulators of Arabidopsis root development [J]. Nature, 2007, 449(7165): 1053−1057. DOI: 10.1038/nature06206
[11] LJUNG K, HULL A K, CELENZA J, et al. Sites and regulation of auxin biosynthesis in Arabidopsis roots [J]. The Plant Cell, 2005, 17(4): 1090−1104. DOI: 10.1105/tpc.104.029272
[12] BLILOU I, XU J, WILDWATER M, et al. The PIN auxin efflux facilitator network controls growth and patterning in Arabidopsis roots [J]. Nature, 2005, 433(7021): 39−44. DOI: 10.1038/nature03184
[13] BENKOVÁ E, MICHNIEWICZ M, SAUER M, et al. Local, efflux-dependent auxin gradients as a common module for plant organ formation [J]. Cell, 2003, 115(5): 591−602. DOI: 10.1016/S0092-8674(03)00924-3
[14] FRIML J, WIŚNIEWSKA J, BENKOVÁ E, et al. Lateral relocation of auxin efflux regulator PIN3 mediates tropism in Arabidopsis [J]. Nature, 2002, 415(6873): 806−809. DOI: 10.1038/415806a
[15] 张倩倩, 郑童, 予茜, 等. 生长素与植物根尖干细胞巢的维持 [J]. 植物学报, 2018, 53(1):126−138. ZHANG Q Q, ZHENG T, YU A Y, et al. Auxin and the maintenance of root stem cell niches in plants [J]. Chinese Bulletin of Botany, 2018, 53(1): 126−138.(in Chinese)
[16] GRIENEISEN V A, XU J, MARÉE A F M, et al. Auxin transport is sufficient to generate a maximum and gradient guiding root growth [J]. Nature, 2007, 449(7165): 1008−1013. DOI: 10.1038/nature06215
[17] MATSUZAKI Y, OGAWA-OHNISHI M, MORI A, et al. Secreted peptide signals required for maintenance of root stem cell niche in Arabidopsis [J]. Science, 2010, 329(5995): 1065−1067. DOI: 10.1126/science.1191132
[18] DU Y J, SCHERES B. PLETHORA transcription factors orchestrate de novo organ patterning during Arabidopsis lateral root outgrowth [J]. Proceedings of the National Academy of Sciences of the United States of America, 2017, 114(44): 11709−11714. DOI: 10.1073/pnas.1714410114
[19] KAREEM A, DURGAPRASAD K, SUGIMOTO K, et al. PLETHORA genes control regeneration by a two-step mechanism [J]. Current Biology, 2015, 25(8): 1017−1030. DOI: 10.1016/j.cub.2015.02.022
[20] 丁一巍, 詹亚光, 张佳薇, 等. 水曲柳2个PLT转录因子基因的克隆及表达分析 [J]. 植物研究, 2019, 39(1):139−147. DING Y W, ZHAN Y G, ZHANG J W, et al. Cloning and expression analysis of two PLT transcription factors genes in Fraxinus mandshurica [J]. Bulletin of Botanical Research, 2019, 39(1): 139−147.(in Chinese)
[21] 张松, 黄波, 夏学峰, 等. 蛋白质亚细胞定位的生物信息学研究 [J]. 生物化学与生物物理进展, 2007, 34(6):573−579. ZHANG S, HUANG B, XIA X F, et al. Bioinformatics research in subcellular localization of protein [J]. Progress in Biochemistry and Biophysics, 2007, 34(6): 573−579.(in Chinese)
[22] NOLE-WILSON S, TRANBY T L, KRIZEK B A. AINTEGUMENTA-like (AIL) genes are expressed in young tissues and may specify meristematic or division-competent states [J]. Plant Molecular Biology, 2005, 57(5): 613−628. DOI: 10.1007/s11103-005-0955-6
[23] SANTUARI L, SANCHEZ-PEREZ G F, LUIJTEN M, et al. The PLETHORA gene regulatory network guides growth and cell differentiation in Arabidopsis roots [J]. The Plant Cell, 2016, 28(12): 2937−2951. DOI: 10.1105/tpc.16.00656
[24] PINON V, PRASAD K, GRIGG S P, et al. Local auxin biosynthesis regulation by PLETHORA transcription factors controls phyllotaxis in Arabidopsis [J]. Proceedings of the National Academy of Sciences of the United States of America, 2013, 110(3): 1107−1112. DOI: 10.1073/pnas.1213497110
[25] MITTLER R, BLUMWALD E. The roles of ROS and ABA in systemic acquired acclimation [J]. The Plant Cell, 2015, 27(1): 64−70. DOI: 10.1105/tpc.114.133090




 下载:
下载: