Research Progress on CFEM Proteins in Phytopathogenic Fungi
-
摘要: 病原真菌在侵染植物过程中,往往分泌效应因子增加致病性或被寄主抗病基因识别激发寄主强烈抗性,因此效应因子在植物-病原物的互作中发挥重要作用。CFEM(Common in Fungal Extracellular Membrane)蛋白是真菌中特有的一类位于细胞外膜的蛋白,往往起效应因子的作用。本文综述了植物病原真菌CFEM蛋白家族的结构特点,在不同物种中的表达和定位,CFEM蛋白对真菌胞内铁吸收和生长发育方面的调控作用,抑制寄主免疫反应和促进真菌寄生的作用机制以及CFEM蛋白起源和进化过程等方面的研究进展,同时对目前CFEM蛋白研究有待阐明的问题进行了讨论并展望了未来的研究方向。本文将有助于人们进一步了解植物-病原真菌互作分子机理以及CFEM蛋白的致病机理,为培育作物抗病品种和制定植物真菌病害防控策略提供参考。Abstract: In the process of infecting plants, pathogenic fungi may often secrete effectors to increase pathogenicity, or conversely, be recognized by the host to trigger resistance. Hence, such effectors are crucial for the well-being of a plant at time of a pathogenic intrusion. As an effector, CFEM (common in fungal extracellular membrane) protein is found only in the outer membrane of a fungal pathogen. This article reviews the origin, evolution, growth, development, structural characteristics, expressions and localization in different species, and intracellular iron absorption regulations of the CFEM protein family as well as the host immunology and infection-promoting factors associated with the proteins in the phytopathogens. Some of the yet-to-be-clarified issues on the molecular mechanisms and plant-pathogen interactions relating to CFEM proteins are discussed to facilitate the cultivation of disease-resistant plants and the development of ecologically friendly disease control strategies on crops.
-
Keywords:
- Phytopathogenic fungi /
- CFEM protein /
- plant immunity /
- iron absorption
-
植物病原菌在侵染植物的过程中会分泌一些效应因子(又称效应蛋白,effector)到寄主细胞,随后效应因子靶标到不同寄主蛋白,破坏寄主抗病信号途径进而促进致病[1]。因此,效应因子是病原物毒性水平和寄主范围的关键决定因子[2]。病原物效应因子可抑制病原物相关分子模式(pathogen-associated molecular patter,PAMP)触发的寄主免疫反应(PAMP-triggered immunity,PTI)或效应因子触发的免疫反应(effector-triggered immunity,ETI)[3]。PTI和ETI的反应强度不同,ETI会导致强烈的过敏反应(Hypersensitive Response,HR),这种反应发生在病原物侵染部位并阻止病原物进一步扩散[4-5]。
CFEM(Common in Fungal Extracellular Membrane)是真菌中特有的一类位于细胞外膜的蛋白,许多CFEM结构蛋白被证明在病原真菌与寄主植物互作中起效应因子的作用[6-7]。目前有关真菌CFEM蛋白生物学功能的研究相对匮乏,本文主要综述真菌CFEM蛋白的结构、表达模式与定位、起源和进化过程以及调控真菌铁吸收、生长发育与致病性的功能与机制,对阐明植物-病原真菌互作分子机理、揭示真菌CFEM蛋白的功能奠定基础。
1. CFEM蛋白家族
1.1 CFEM蛋白的结构
CFEM是真菌中特异的常见于细胞外膜的一种蛋白,其结构域约有60个氨基酸,共有8个间隔排列的高度保守的半胱氨酸(图1),其特点是将其与已知的富含半胱氨酸的结构域区分开来。与其他富含半胱氨酸的蛋白不同,该蛋白结构域仅存在于真菌中,是常见的真菌胞外膜蛋白,并因此而得名[6]。CFEM类效应因子可以附着在真菌细胞壁上,并可以转移到糖基磷脂酰肌醇(Glycosylphosphatidylinositol,GPI)锚定位点,在真菌致病过程中发挥重要作用[8]。
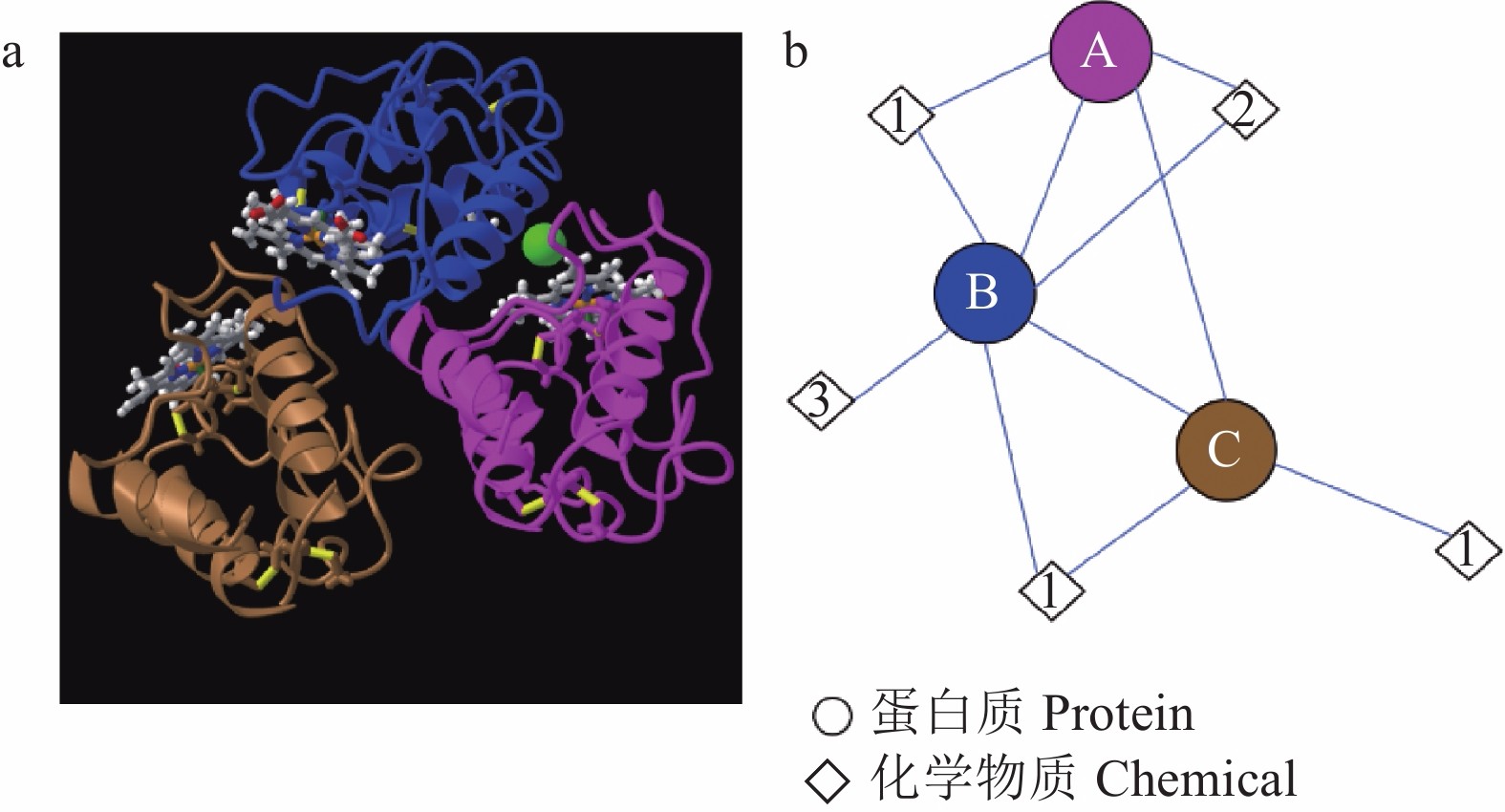
自首次从粗球孢子菌(Coccidioides immitis)中鉴定出CFEM类蛋白以来,已经从多种真菌中鉴定出CFEM蛋白,包括白色念珠菌(Candida albicans)、近平滑假丝酵母(Candida parapsilosis)、稻瘟菌(Magnaporthe oryzae)、少孢节丛孢菌(Arthrobotrys oligospora)、禾谷炭疽菌(Colletotrichum graminicola)和烟曲霉(Aspergillus fumigatus)等[6,8-10]。例如,稻瘟菌中发现了19个CFEM蛋白[11],白色念珠菌中发现了6个,少孢节丛孢菌中发现了13个[7],禾谷炭疽菌中发现了24个[12]。这些已经发现的CFEM蛋白中,白色念珠菌中的Csa2是唯一鉴定了三维(3D)晶体结构的 (图2):该蛋白的CFEM结构域由6个α-螺旋和1个β-链组成,可形成以拉长的N-末端环为柄的螺旋篮状结构[13]。Gong等[12]在禾谷炭疽菌中发现的24个CFEM蛋白中也包含类似于Csa2的螺旋篮状结构,其中6个CFEM蛋白(CgCFEM3、7、8、9、15和20)包含4个α-螺旋,3个CFEM蛋白(CgCFEM4、6和14)包含3个α-螺旋,而CgCFEM1仅包含2个α-螺旋。
![]() 图 2 白色念珠菌Csa2蛋白的CFEM结构a:Csa2蛋白3个CFEM结构域的3D晶体结构为三聚体(引自NCBI); b:Csa2蛋白的分子组分及它们之间相互作用示意图。A、B、C分别代表3个不同的CFEM结构域,1,2,3分别代表血红素B/C、氯离子和1,2-乙二醇。Figure 2. Structure of CFEM protein domain in Csa2 of Candida albicansa: “3D crystal structure of 3 CFEM protein domains in Csa2 is a trimer” as quoted from NCBI; b: Schematic molecular components and interactions of Csa2. A, B, and C represent 3 CFEM protein domains; 1, 2, and 3 represent heme B/C, chloride ion, and 1,2-ethanediol, respectively.
图 2 白色念珠菌Csa2蛋白的CFEM结构a:Csa2蛋白3个CFEM结构域的3D晶体结构为三聚体(引自NCBI); b:Csa2蛋白的分子组分及它们之间相互作用示意图。A、B、C分别代表3个不同的CFEM结构域,1,2,3分别代表血红素B/C、氯离子和1,2-乙二醇。Figure 2. Structure of CFEM protein domain in Csa2 of Candida albicansa: “3D crystal structure of 3 CFEM protein domains in Csa2 is a trimer” as quoted from NCBI; b: Schematic molecular components and interactions of Csa2. A, B, and C represent 3 CFEM protein domains; 1, 2, and 3 represent heme B/C, chloride ion, and 1,2-ethanediol, respectively.1.2 CFEM蛋白的表达和定位
不同种类真菌中CFEM蛋白的表达部位不同,同一种CFEM蛋白在同一种真菌不同时期的表达量也不同。灰霉病菌(Botrytis cinerea)中的BcCFEM1是一种具有假定GPI锚定位点的CFEM蛋白,大豆接种灰霉病菌后,BcCFEM1的表达水平从0 hpi (hour post-infection)到36 hpi呈上升趋势,之后逐渐下降,但是仍高于0 hpi时的转录水平[14]。在番茄叶片感染灰霉病菌后发现,非G蛋白偶联受体(non-G-protein-coupled receptor,non-GPCR)膜结合CFEM蛋白Bcin07g03260在该菌分生孢子萌发阶段(16 hpi)的表达水平比0 hpi增加了3倍,在坏死生长阶段(48 hpi)表达水平又随之降低[15]。Ling等[16]发现尖孢镰刀菌(Fusarium oxysporum)中属于CFEM家族的基因CFEM_DR1、2、3、4、5和6在侵染的不同时期的表达有所不同。F. oxysporum f. sp. cubense1(Foc1)侵染甘蓝后,CFEM_DR基因的表达分析中可以检测到CFEM_DR2、4、5、6这4个基因的转录水平,但不能检测到CFEM_DR1和CFEM_DR3,可能是因为Foc侵染期间这些基因的表达水平较低。总体而言,这4个CFEM_DR基因的表达在3 dpi(day post-infection)时显著增加。
近平滑假丝酵母中已发现7个CFEM蛋白,其中CFEM2、CFEM3和CFEM6的表达依赖于生物膜和细胞壁调节因子Bcr1,并且这些基因都在低铁条件下被诱导[17]。与这种表达模式相似的还有白色念珠菌中的CFEM蛋白Csa1、Pga7、Pga10和Rbt5[18-19]。Sosinska等研究发现,与pH=4.0的生长条件相比,白色念珠菌在pH=7.0的条件下,Rbt5蛋白的表达水平提高了10倍[20-21]。除此之外,部分CFEM蛋白的表达还与温度有关,例如在可可毛色二孢菌(Lasiodiplodia theobromae)中,与25 ℃和35 ℃时的培养条件相比,LtCFEM1、LtCFEM2、LtCFEM4、LtCFEM5和LtCFEM7在30 ℃时显示出更高的转录水平;与25 ℃相比,在30 ℃和35 ℃下LtCFEM6和LtCFEM8的表达水平均被上调,然而LtCFEM3的表达却被下调[22]。以上结果表明,不同真菌中CFEM蛋白在不同部位、不同时期以及不同生长条件下的表达量不同,预示CFEM蛋白在病原真菌侵染植物时发挥的功能可能是多种多样的。
蛋白质作用的位置与其功能密切相关。为了解CFEM蛋白在细胞中的定位,Gong等[12]选取了禾谷炭疽菌中的10个CFEM效应蛋白在烟草中进行异源表达分析,结果发现大多数CFEM的全长(full length,FL)和去掉信号肽(without signal peptide,NS)部分在细胞中的分布相似。其中CgCFEM1、4、7、9、14、15和20这7种蛋白在细胞膜上被检测到,CgCFEM1 (FL和NS)、CgCFEM4 (NS)和CgCFEM9(FL)在胞质中积累;CgCFEM6和CgCFEM8在细胞核中表达。但也有部分CFEM效应蛋白全长序列与去信号肽序列定位信息不一致,如CgCFEM15 (FL)和CgCFEM20 (FL)定位于细胞膜上,而去掉信号肽之后则在包括细胞膜、细胞核和胞质的整个细胞中表达[12,23]。此外,通过对稻瘟菌中CFEM家族的ACI1蛋白信号肽序列和跨膜区段的分析以及GPI锚定位点的预测,表明它可能是一种膜定位蛋白[24]。该菌中同属于CFEM家族的Pth11和WISH也具有这种特征[25-26]。绿僵菌(Metarhizium anisopliae)中的MaCFEM81、85、87、89和90也属于膜定位蛋白,然而该菌的MaCFEM88在细胞膜和细胞核上均能表达[27]。Wang等[28]发现黄萎病菌(Verticillium dahliae)中的2个CFEM蛋白VdSCP76和VdSCP77,无论是融合红色荧光蛋白还是绿色荧光蛋白,都位于宿主细胞外围,尤其是在质膜上。这表明CFEM蛋白可能在植物体内不同部位发挥作用,明确其在寄主细胞内的定位可以为进一步研究其功能提供可靠的依据。
2. CFEM蛋白的演化
2.1 CFEM蛋白的起源
CFEM结构域至今仅发现存在于真菌蛋白中,在动物、植物和原核生物蛋白中尚未发现该结构[29]。为研究CFEM结构的起源,有研究者对100种真菌的基因组进行CFEM结构搜索,结果显示有64种真菌含有359个CFEM结构的蛋白,主要分布于子囊菌门和担子菌门中,说明CFEM蛋白在真菌中分布较广泛[30-31]。
为了更清楚地了解CFEM结构的分布模式与起源,张真娜[30]构建了担子菌门14个、子囊菌亚门89个和酵母菌亚门10个CFEM蛋白结构域的系统发育关系。结果表明CFEM结构在整个系统发育树中呈分散状态,它们不在同一分类门中聚集出现,这表明CFEM没有独立的起源。也就是说,CFEM结构域的单一起源可以追溯到子囊菌门和担子菌门的最近共同祖先。
2.2 CFEM蛋白的进化
CFEM结构域的形成机制有多种,结构域重复和基因重复是两种最常见的CFEM结构扩增机制,此外还有结构域滑移、基因转换等机制。随着结构域的扩展,其功能也出现了相应的分化[30]。例如,非致病性的酵母菌(Saccharomyces cerevisiae)中只有1个含有CFEM的蛋白CCW14,其参与细胞壁内层的形成[32-33]。致病性真菌尽管往往有多个含CFEM结构的蛋白,但都有一个与CCW14同源的蛋白参与细胞壁的形成,剩下的其他CFEM蛋白则具有不同的功能[7]。这说明CFEM结构最初始的功能只是构成细胞壁或细胞膜的结构组成部分,致病性等功能是在后期的进化过程中产生的。
通过比较不同物种CFEM结构域的长度发现,不同真菌中CFEM的数量存在明显差异[34-35],致病性真菌的CFEM结构蛋白比非致病性真菌的更多。物种自身结构的复杂性以及功能的变化,这些可能是导致CFEM蛋白数目增加的原因。也就是说,越高等的真菌物种含有的CFEM结构域更多,而有些物种所处的环境变化多端,如营寄生生活的真菌或致病性真菌,所含的CFEM结构域数目往往也相应增加,有可能与该结构域维持物种基本的生存能力有关系[7]。
3. CFEM蛋白的功能及作用机制
3.1 维持真菌胞内所需铁含量
几乎所有植物、细菌和真菌生长发育都需要铁营养元素[36]。在病原微生物侵染寄主植物时,病原微生物及其寄主都需要铁离子来保持各自的生理功能、生长和增殖。因此,铁离子信号影响植物与病原微生物之间的互作关系。目前已在多个真菌体内发现了铁吸收系统,例如,铁载体调控的Fe3+吸收、还原铁同化作用、亚铁血红素吸收或者Fe2+直接吸收[37]。
一些研究表明,CFEM蛋白在维持真菌细胞内铁含量方面发挥重要的作用。例如,白色念珠菌中的高度同源的两个蛋白Rbt5和Rbt51是CFEM家族的一种细胞外GPI锚定血红素结合蛋白,可促进血红素铁的摄取。Rbt5是白色念珠菌利用血红素和血红蛋白作为铁源的关键因子,缺失会引起严重的缺铁表型[17]。Pga7的缺失会引起比Rbt5缺失更严重的缺铁表型,表明这两个蛋白质都能有效地帮助白色念珠菌从血红蛋白中获取铁元素[38]。其中Rbt5对白色念珠菌的致病性并没有明显作用,而Pga7是该菌致病所必需的[38-39]。酵母菌中虽然缺乏血红蛋白受体Rbt5的直系同源物,但是在其体内异源表达白色念珠菌中的Rbt51时,同样也能使其将血红蛋白作为铁源[10]。除此之外,Csa2是白色念珠菌中Rbt5蛋白家族和CFEM蛋白家族的成员,有研究证明Csa2在白色念珠菌菌丝生长过程中参与利用血红蛋白中的铁元素[40]。Csa2蛋白的第80位天冬氨酸残基能帮助C. albicans获取血红素中的Fe3+,如果该突变位点为组氨酸残基,则该菌获取的是血红素中的Fe2+[13]。Srivastava等[41]首次证明了光滑假丝酵母(Candida glabrata)中属于CFEM家族的细胞壁结构蛋白CgCcw14和假定溶血素CgMam3,对维持细胞内铁含量至关重要。
3.2 调控真菌的生长发育
Ling等[16]研究发现尖孢镰刀菌的CFEM_DR蛋白在菌丝体中的表达比在分生孢子中的表达要高,并且一些CFEM_DR基因在菌丝体生长晚期的表达比早期更高,表明CFEM_DR基因在尖孢镰刀菌菌丝体生长中具有重要作用。而且尖孢镰刀菌能穿透寄主根的皮层组织进入维管组织,并通过菌丝体的生长与液流一起运输。灰霉病菌中的BcCFEM1敲除后,虽然∆Bccfem1突变体分生孢子的萌发没有受到影响,但其分生孢子产量明显减少[14]。CFEM蛋白Bcin07g03260的缺失,导致∆CFEM-Bcin07g03260突变体分生孢子萌发率明显降低,同时芽管长度显著缩短[15]。
稻瘟菌中被命名为WISH的蛋白同时具有CFEM和GPCR结构。该基因调控稻瘟菌的菌丝生长和分生孢子分化与形成,此外,还在维持细胞壁完整性、对宿主的表面感应和真菌表面疏水性方面发挥调控作用。敲除WISH后,∆wish突变体分生孢子产量明显增加并且无法形成附着胞进而导致无法侵染寄主,这表明WISH蛋白负调控稻瘟菌分生孢子的产生且是该菌附着胞形成所必需的[42]。
希金斯炭疽菌(Colletotrichum higginsianum Sacc.)中含有CFEM结构域的效应因子ChEP113被敲除后,∆ChEP113突变体菌株气生菌丝增多,菌丝生长变慢且形态明显改变:末端笔直、分叉减少,孢子产量和菌丝重量明显增加;孢子萌发时大多只在一侧产生芽管,且只有少数分生孢子可以正常萌发并产生黑化附着胞,这表明ChEP113蛋白参与调控希金斯炭疽菌的生长发育,在该菌菌落形态、菌丝生长、产孢和孢子萌发方面发挥重要作用[43]。
3.3 影响真菌致病力
已有研究表明,真菌在侵染阶段表达的分泌蛋白更有可能影响病原体的致病性[44]。Wang等通过研究表明在侵染阶段玉米大斑病菌(Setosphaeria turcica)中包含StCFEM3、4、5,10、12和13这6个效应蛋白在内的13个CFEM蛋白都有表达,这表明玉米大斑病菌中的StCFEMs可能起着致病因子的作用[45]。尖孢镰刀菌中CFEM_DR基因在侵染寄主后第3天在寄主根中大量表达,而该蛋白已经被证实了在宿主的定植和侵染中发挥重要的致病作用[16]。小新壳梭孢菌(Neofusicoccum parvum)中含有CFEM结构域的蛋白NpCysRP5也是在早期侵染阶段高度表达[46]。此外,还有灰霉病菌中的BcCFEM1[14]、尖孢镰刀菌中的CFEM_DR基因[16]、C. graminicola中22个含有CFEM结构域的效应因子[12]以及可可毛色二孢菌中除LtCFEM2和LtCFEM6外的LtCFEMs等[22],并且在烟草中异源表达BcCFEM1、LtCFEM1会导致叶片明显萎黄[14,22]。Chen等[47]通过研究发现禾谷镰刀菌(Fusarium graminearum)中的FgCFEM11和FgCFEM23有可能在侵染植物期间作为效应因子参与致病过程。小麦叶锈菌(Puccinia triticina)中的CFEM效应因子PTTG-08198加速了由BAX触发的植物细胞死亡并且诱导了烟草细胞中活性氧(Reactive Oxygen Species,ROS)的积累[48]。MoCDIP2和MoCDIP11是稻瘟菌中诱导寄主细胞死亡和引发水稻防御反应的CFEM效应因子[49-50],而禾谷炭疽菌中具有CFEM结构域的GLRG_01006.1却是能抑制细胞死亡的效应因子[23]。
为进一步研究CFEM蛋白对真菌毒力的影响,Zhu等对BcCFEM1进行了敲除,结果表明突变体菌株表现出对寄主的致病性降低以及对过氧化氢的敏感性增强。推测突变体的致病性降低可能是其对植物防御相关活性氧高度敏感性的结果[14]。核盘菌(Sclerotinia sclerotiorum)中的SsCFEM1被敲除后,突变体菌株毒力也明显下降[51]。Peng等[52]通过局部感染和注射蜡蛾幼虫来评估白僵菌(Beauveria bassiana)的毒力,结果显示野生型菌株杀死了所有幼虫,而∆Bbcfem7和∆Bbcfem8突变体菌株对蜡蛾幼虫的致死率不到50%,表明BbCFEM7和BbCFEM8显著促进了该菌的毒力。以上结果都说明了CFEM蛋白对真菌病原物的重要性,同时也表明CFEM蛋白可能作为毒性致病因子参与调控真菌致病性相关的重要生理过程。但不是所有的CFEM蛋白都和真菌致病性有关,例如CfmA-C可能与烟曲霉细胞壁的稳定性有关,但其缺失不影响该菌的致病力[8],还有CCW14是在非致病性真菌中发现的[32-33]。
3.4 促进真菌寄生
在白色念珠菌中,菌丝体的生长和生物膜的形成能力被认为是重要的毒力特征,Pérez等的研究中表明,Csa1、Pga7和Rbt5在菌丝中上调表达,并且编码这类蛋白的基因被敲除后,突变体在生物膜形成过程中存在缺陷[53]。这表明这些蛋白可能作为信号分子和毒性致病因子在病原菌侵染过程中发挥作用。除此之外,在稻瘟菌中,WISH被敲除后,附着胞发育明显被抑制,无法识别寄主表面和穿透水稻叶片,导致其致病性几乎丧失,这表明WISH蛋白参与宿主表面识别和真菌致病性[43]。ACI1为胞外受体信号分子,通过与腺苷酸环化酶(MAC1)互作,参与环磷酸腺苷(cyclic adenosine monophosphate,cAMP)信号传递,直接影响稻瘟菌附着胞的形成[54-55]。同样地,Pth11作为稻瘟菌附着胞及芽管胞内信号分子,被敲除后,突变体表现出附着胞形成延迟以及致病性减弱[11,56]。CFEM作为一种膜蛋白,可以充当细胞表面受体或信号转导因子,或者在宿主-病原体互作中充当粘附分子[6],因此,推测CFEM结构域蛋白可能通过这些途径发挥作用从而促进真菌寄生。
4. 问题与展望
CFEM作为真菌特有的蛋白结构,其在不同真菌中的分布数有较大差异,说明CFEM蛋白生物学功能存在较大进化与差异。CFEM蛋白除了最初的功能参与细胞壁形成有关外,目前报道的CFEM蛋白功能主要体现在维持真菌胞内所需铁含量,参与真菌生长发育过程,促进病原真菌致病及抑制寄主免疫反应等。然而,CFEM的研究存在以下几方面问题有待进一步解决:尽管CFEM蛋白在真菌中分布广泛,然而关于CFEM蛋白功能的研究目前大多数只限于稻瘟菌、禾谷炭疽菌、白色念珠菌等少数真菌,在其他真菌中的研究非常的少;其次,CFEM蛋白的功能逐渐被人们揭示,然而关于其作用机理、作用靶标的研究还存在较大空白,需进一步增加有关CFEM蛋白功能研究的深度,解析CFEM蛋白的功能分子机制;此外,虽然CFEM结构域的长度与氨基酸序列具有高度的保守性,但不同真菌物种CFEM结构域的数量存在显著差异,其功能的多样性是否与真菌体内CFEM蛋白数量以及结构域进化有关。这些问题都需要进一步的深入研究来阐明。通过揭示CFEM蛋白的功能,其作用靶标与上下游信号网络及其起源和分化,将有助于阐明CFEM蛋白的生理功能,进一步丰富植物-病原物相互作用机理,为作物病害防控策略的制定提供参考。
-
图 2 白色念珠菌Csa2蛋白的CFEM结构
a:Csa2蛋白3个CFEM结构域的3D晶体结构为三聚体(引自NCBI); b:Csa2蛋白的分子组分及它们之间相互作用示意图。A、B、C分别代表3个不同的CFEM结构域,1,2,3分别代表血红素B/C、氯离子和1,2-乙二醇。
Figure 2. Structure of CFEM protein domain in Csa2 of Candida albicans
a: “3D crystal structure of 3 CFEM protein domains in Csa2 is a trimer” as quoted from NCBI; b: Schematic molecular components and interactions of Csa2. A, B, and C represent 3 CFEM protein domains; 1, 2, and 3 represent heme B/C, chloride ion, and 1,2-ethanediol, respectively.
-
[1] SNELDERS N C, ROVENICH H, PETTI G C, et al. A plant pathogen utilizes effector proteins for microbiome manipulation [J]. Nature Plants, 2020(1): 926725.
[2] LIU W D, LIU J L, TRIPLETT L, et al. Novel insights into rice innate immunity against bacterial and fungal pathogens [J]. Annual Review of Phytopathology, 2014, 52: 213−241. DOI: 10.1146/annurev-phyto-102313-045926
[3] TORUÑO T Y, STERGIOPOULOS I, COAKER G. Plant-pathogen effectors: Cellular probes interfering with plant defenses in spatial and temporal manners [J]. Annual Review of Phytopathology, 2016, 54: 419−441. DOI: 10.1146/annurev-phyto-080615-100204
[4] RAFIQI M, ELLIS J G, LUDOWICI V A, et al. Challenges and progress towards understanding the role of effectors in plant-fungal interactions [J]. Current Opinion in Plant Biology, 2012, 15(4): 477−482. DOI: 10.1016/j.pbi.2012.05.003
[5] XU Q, TANG C, WANG X, et al. An effector protein of the wheat stripe rust fungus targets chloroplasts and suppresses chloroplast function [J]. Nature Communications, 2019, 10(1): 5571. DOI: 10.1038/s41467-019-13487-6
[6] KULKARNI R D, KELKAR H S, DEAN R A. An eight-cysteine-containing CFEM domain unique to a group of fungal membrane proteins [J]. Trends in Biochemical Sciences, 2003, 28(3): 118−121. DOI: 10.1016/S0968-0004(03)00025-2
[7] ZHANG Z N, WU Q Y, ZHANG G Z, et al. Systematic analyses reveal uniqueness and origin of the CFEM domain in fungi [J]. Scientific Reports, 2015, 5: 13032. DOI: 10.1038/srep13032
[8] YAKIR V, YANA S, EMMA L, et al. The three Aspergillus fumigatus CFEM-domain GPI-anchored proteins (CfmA-C) affect cell-wall stability but do not play a role in fungal virulence [J]. Fungal Genetics and Biology, 2014, 63: 55−64. DOI: 10.1016/j.fgb.2013.12.005
[9] RAMANUJAM R, CALVERT M E, SELVARAJ P, et al. The late endosomal hops complex anchors active g-protein signaling essential for pathogenesis in Magnaporthe oryzae [J]. PLoS Pathogens, 2013, 9(8): e1003527. DOI: 10.1371/journal.ppat.1003527
[10] WEISSMAN Z, KORNITZER D. A family of Candida cell surface haem-binding proteins involved in haemin and haemoglobin-iron utilization [J]. Molecular Microbiology, 2004, 53(4): 1209−1220. DOI: 10.1111/j.1365-2958.2004.04199.x
[11] KOU Y, TAN Y H, RAMANUJAM R, et al. Structure-function analyses of the Pth11 receptor reveal an important role for CFEM motif and redox regulation in rice blast [J]. The New Phytologist, 2017, 214(1): 330−342. DOI: 10.1111/nph.14347
[12] GONG A D, JING Z Y, ZHANG K, et al. Bioinformatic analysis and functional characterization of the CFEM proteins in maize anthracnose fungus Colletotrichum graminicola [J]. Journal of Integrative Agriculture, 2020, 19(2): 541−550. DOI: 10.1016/S2095-3119(19)62675-4
[13] NASSER L, WEISSMAN Z, PINSKY M, et al. Structural basis of haem-iron acquisition by fungal pathogens [J]. Nat Microbiol, 2016, 1(11): 16156. DOI: 10.1038/nmicrobiol.2016.156
[14] ZHU W, WEI W, WU Y, et al. BcCFEM1, a CFEM domain-containing protein with putative GPI-anchored site, is involved in pathogenicity, conidial production, and stress tolerance in Botrytis cinerea [J]. Frontiers in Microbiology, 2017, 8: 1807. DOI: 10.3389/fmicb.2017.01807
[15] ARYA G C, SRIVASTAVA D A, PANDARANAYAKA E P J, et al. Characterization of the role of a non-GPCR membrane-bound CFEM protein in the pathogenicity and germination of Botrytis cinerea [J]. Microorganisms, 2020, 8(7): E1043. DOI: 10.3390/microorganisms8071043
[16] LING J, ZENG F, CAO Y X, et al. Identification of a class of CFEM proteins containing a new conserved motif in Fusarium oxysporum [J]. Physiological and Molecular Plant Pathology, 2015, 89: 41−48. DOI: 10.1016/j.pmpp.2014.12.001
[17] DING C, VIDANES G M, MAGUIRE S L, et al. Conserved and divergent roles of Bcr1 and CFEM proteins in Candida parapsilosis and Candida albicans [J]. PLoS One, 2011, 6(12): e28151. DOI: 10.1371/journal.pone.0028151
[18] CHEN C, PANDE K, FRENCH S D, et al. An iron homeostasis regulatory circuit with reciprocal roles in Candida albicans commensalism and pathogenesis [J]. Cell Host & Microbe, 2011, 10(2): 118−135.
[19] SORGO A G, BRUL S, DE KOSTER C G, et al. Iron restriction-induced adaptations in the wall proteome of Candida albicans[J]. Microbiology (Reading, England), 2013, 159(pt 8): 1673-1682.
[20] SOSINSKA G J, DE KONING L J, DE GROOT P W J, et al. Mass spectrometric quantification of the adaptations in the wall proteome of Candida albicans in response to ambient pH[J]. Microbiology (Reading), 2011, 157(pt 1): 136-146.
[21] PÉREZ A, RAMAGE G, BLANES R, et al. Some biological features of Candida albicans mutants for genes coding fungal proteins containing the CFEM domain [J]. FEMS Yeast Research, 2011, 11(3): 273−284. DOI: 10.1111/j.1567-1364.2010.00714.x
[22] PENG J, WU L, ZHANG W, et al. Systemic identification and functional characterization of common in fungal extracellular membrane proteins in Lasiodiplodia theobromae [J]. Front Plant Sci, 2021, 12: 804696. DOI: 10.3389/fpls.2021.804696
[23] 井忠英. 玉米炭疽病菌CFEM效应子的系统鉴定与功能分析[D]. 北京: 中国农业科学院, 2015. JING Z Y. Systematic identification and functional analysis of CFEM effectors in Colletotrichum graminicola[D]. Beijing: Chinese Academy of Agricultural Sciences Dissertation, 2015. (in Chinese)
[24] DENG J, DEAN R A. Characterization of adenylate cyclase interacting protein ACI1 in the rice blast fungus, Magnaporthe oryzae [J]. Open Mycology Journal, 2008, 2(1): 74−81. DOI: 10.2174/1874437000802010074
[25] DEZWAAN T M, CARROLL A M, VALENT B, et al. Magnaporthe grisea pth11p is a novel plasma membrane protein that mediates appressorium differentiation in response to inductive substrate cues [J]. Plastic and Reconstructive Surgery, 1999, 11(10): 2013−2030.
[26] XU X, LI G, LI L, et al. Genome-wide comparative analysis of putative Pth11-related G protein-coupled receptors in fungi belonging to Pezizomycotina [J]. BMC Microbiology, 2017, 17(1): 166. DOI: 10.1186/s12866-017-1076-5
[27] CAI N, LIU R, YAN D, et al. Bioinformatics analysis and functional characterization of the CFEM proteins of Metarhizium anisopliae [J]. J Fungi (Basel), 2022, 8(7): 661. DOI: 10.3390/jof8070661
[28] WANG D, ZHANG D D, SONG J, et al. Verticillium dahliae CFEM proteins manipulate host immunity and differentially contribute to virulence [J]. BMC Biol, 2022, 20(1): 55. DOI: 10.1186/s12915-022-01254-x
[29] KULKARNI R D, THON M R, PAN H, et al. Novel G-protein-coupled receptor-like proteins in the plant pathogenic fungus Magnaporthe grisea [J]. Genome Biol, 2005, 6(3): R24. DOI: 10.1186/gb-2005-6-3-r24
[30] 张真娜. CFEM结构域在真菌中的进化研究[D]. 昆明: 云南大学, 2012. ZHANG Z N. Evolutionary pattems of CFEM domain in fungi[D]. Kunming: Yunnan university, 2012. (in Chinese)
[31] JAMES T Y, KAUFF F, SCHOCH C L, et al. Reconstructing the early evolution of Fungi using a six-gene phylogeny [J]. Nature, 2006, 443(7113): 818−822. DOI: 10.1038/nature05110
[32] MOUKADIRI I, ARMERO J, ABAD A, et al. Identification of a mannoprotein present in the inner layer of the cell wall of Saccharomyces cerevisiae [J]. RSC Advances, 1997, 179(7): 2154−2162.
[33] INOKUMA K, KITADA Y, BAMBA T, et al. Improving the functionality of surface-engineered yeast cells by altering the cell wall morphology of the host strain [J]. Applied Microbiology and Biotechnology, 2021, 105(14/15): 5895−5904.
[34] PENDLETON A L, SMITH K E, FEAU N, et al. Duplications and losses in gene families of rust pathogens highlight putative effectors [J]. Frontiers in Plant Science, 2014, 5: 299.
[35] VELA-CORCÍA D, BAUTISTA R, DE VICENTE A, et al. De novo analysis of the epiphytic transcriptome of the cucurbit powdery mildew fungus Podosphaera xanthii and identification of candidate secreted effector proteins [J]. PLoS One, 2016, 11(10): e0163379. DOI: 10.1371/journal.pone.0163379
[36] XING Y, XU N, BHANDARI D D, et al. Bacterial effector targeting of a plant iron sensor facilitates iron acquisition and pathogen colonization [J]. The Plant Cell, 2021, 33(6): 2015−2031. DOI: 10.1093/plcell/koab075
[37] ALBAROUKI E, DEISING H B. Infection structure-specific reductive iron assimilation is required for cell wall integrity and full virulence of the maize pathogen Colletotrichum Graminicola [J]. Molecular Plant-Microbe Interactions, 2013, 26(6): 695−708. DOI: 10.1094/MPMI-01-13-0003-R
[38] KUZNETS G, VIGONSKY E, WEISSMAN Z, et al. A relay network of extracellular heme-binding proteins drives C. albicans iron acquisition from hemoglobin [J]. PLoS Pathogens, 2014, 10(10): e1004407. DOI: 10.1371/journal.ppat.1004407
[39] YE F, ALBAROUKI E, LINGAM B, et al. An adequate Fe nutritional status of maize suppresses infection and biotrophic growth of Colletotrichum Graminicola [J]. Physiologia Plantarum, 2014, 151(3): 280−292. DOI: 10.1111/ppl.12166
[40] OKAMOTO-SHIBAYAMA K, KIKUCHI Y, KOKUBU E, et al. Csa2, a member of the Rbt5 protein family, is involved in the utilization of iron from human hemoglobin during Candida albicans hyphal growth [J]. FEMS Yeast Research, 2014, 14(4): 674−677. DOI: 10.1111/1567-1364.12160
[41] SRIVASTAVA V K, SUNEETHA K J, KAUR R. A systematic analysis reveals an essential role for high-affinity iron uptake system, haemolysin and CFEM domain-containing protein in iron homoeostasis and virulence in Candida glabrata [J]. The Biochemical Journal, 2014, 463(1): 103−114. DOI: 10.1042/BJ20140598
[42] SABNAM N, ROY BARMAN S. WISH, a novel CFEM GPCR is indispensable for surface sensing, asexual and pathogenic differentiation in rice blast fungus [J]. Fungal Genetics and Biology, 2017, 105: 37−51. DOI: 10.1016/j.fgb.2017.05.006
[43] 皮磊. 希金斯炭疽菌效应分子ChEP011和ChEP113的功能分析[D]. 广州: 华南农业大学, 2019. PI L. Functional analysis of effectors ChEP011 and ChEP113 of Colletotrichum higginsianum[D]. Guangzhou: South China Agricultural University, 2019. (in Chinese)
[44] ELLIS J G, RAFIQI M, GAN P, et al. Recent progress in discovery and functional analysis of effector proteins of fungal and oomycete plant pathogens [J]. Current Opinion in Plant Biology, 2009, 12(4): 399−405. DOI: 10.1016/j.pbi.2009.05.004
[45] GONG A D, JING Z Y, ZHANG K, et al. Bioinformatic analysis and functional characterization of the cfem proteins in maize anthracnose fungus Colletotrichum graminicola [J]. Journal of Integrative Agriculture, 2020(2): 541−550.
[46] VÁZQUEZ-AVENDAÑO R, RODRÍGUEZ-HAAS J B, VELÁZQUEZ-DELGADO H, et al. Insights of the Neofusicoccum parvum-Liquidambar styraciflua interaction and identification of new cysteine-rich proteins in both species [J]. Journal of Fungi (Basel, Switzerland), 2021, 7(12): 1027.
[47] CHEN L, WANG H, YANG J, et al. Bioinformatics and transcriptome analysis of CFEM proteins in Fusarium graminearum [J]. J Fungi (Basel), 2021, 7(10): 871. DOI: 10.3390/jof7100871
[48] ZHAO S, SHANG X, BI W, et al. Genome-wide identification of effector candidates with conserved motifs from the wheat leaf rust fungus Puccinia triticina [J]. Front Microbiol, 2020, 11: 1188. DOI: 10.3389/fmicb.2020.01188
[49] CHEN S, SONGKUMARN P, VENU R C, et al. Identification and characterization of in planta-expressed secreted effector proteins from Magnaporthe oryzae that induce cell death in rice [J]. Mol Plant Microbe Interact, 2013, 26(2): 191−202. DOI: 10.1094/MPMI-05-12-0117-R
[50] GUO X, ZHONG D, XIE W, et al. Functional identification of novel cell death-inducing effector proteins from Magnaporthe oryzae [J]. Rice (New York, N Y ), 2019, 12(1): 59.
[51] 纪旭. 核盘菌分泌蛋白SsCFEM1的功能研究[D]. 长春: 吉林大学, 2020. JI X. Functional analysis of the secretory protein SsCFEM1 in Sclerotinia sclerotiorum[D]. Changchun: Jilin University, 2020. (in Chinese)
[52] PENG Y J, HOU J, ZHANG H, et al. Systematic contributions of CFEM domain-containing proteins to iron acquisition are essential for inter species interaction of the filamentous pathogenic fungus Beauveria bassiana [J]. Environmental Microbiology, 2022, 24(8): 3693−3704. DOI: 10.1111/1462-2920.16032
[53] PÉREZ A, PEDRÓS B, MURGUI A, et al. Biofilm formation by Candida albicans mutants for genes coding fungal proteins exhibiting the eight-cysteine-containing CFEM domain [J]. FEMS Yeast Research, 2006, 6(7): 1074−1084. DOI: 10.1111/j.1567-1364.2006.00131.x
[54] CHOI W, DEAN R A. The adenylate cyclase gene MAC1 of Magnaporthe grisea controls appressorium formation and other aspects of growth and development [J]. PLoS One, 1997, 9(11): 1973−1983.
[55] SALOMON D, KINCH L N, TRUDGIAN D C, et al. Marker for type VI secretion system effectors [J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(25): 9271−9276. DOI: 10.1073/pnas.1406110111
[56] JIN Q C, DONG H T, PENG Y L, et al. Application of cDNA array for studying the gene expression profile of mature appressoria of Magnaporthe grisea [J]. J Zhejiang Univ Sci B, 2007, 8(2): 88−97. DOI: 10.1631/jzus.2007.B0088
-
期刊类型引用(1)
1. 王艳玲,郭勇,高洁. 病原真菌CFEM蛋白功能及分子机制研究进展. 华中农业大学学报. 2025(01): 156-167 .  百度学术
百度学术
其他类型引用(2)





 下载:
下载: