Cloning and Expression of SBEII in Eleocharis tuberosa
-
摘要:目的 克隆荸荠(Eleocharis tuberosa)淀粉分支酶CwSBEII基因,分析该基因的蛋白功能特性及其在荸荠不同组织以及不同膨大时期的表达模式,为进一步深入研究CwSBEII基因的功能提供理论参考。方法 采用RT-PCR的方法克隆得到CwSBEII基因的cDNA序列,通过生物信息学分析其蛋白功能特性,利用荧光定量PCR法检测该基因在荸荠球茎中的时空表达特征。结果 成功克隆得到CwSBEII基因,该基因的开放阅读框(ORF)长度为2547 bp,编码848个氨基酸。CwSBEII蛋白分子量为96100.52 Da;理论等电点(pI)值为5.24,不稳定指数为41.07,GRAVY为−0.473,属于不稳定亲水蛋白。系统进化分析表明,荸荠与禾本科的小麦(Triticum aestivum)、水稻(Oryza sativa)、玉米(Zea mays)的SBEII蛋白聚为一支,亲缘关系最近。荧光定量PCR结果表明,CwSBEII基因在根、叶片、荸荠皮以及荸荠肉中均有表达,但在叶片中的表达量最高。在荸荠球茎膨大初期CwSBEII基因表达出现明显上调。结论 CwSBEII基因在荸荠中特异性表达,在叶片中的表达量最高,膨大初期表达明显上调,推测该基因在荸荠淀粉合成中发挥着重要作用。Abstract:Objective CwSBEII in Eleocharis tuberosa was cloned to understand the functions and expressions of the gene in various tissues and corms at different expansion stages.Methods The cDNA sequence of CwSBEII of Chinese water chestnut was cloned by RT-PCR, functions deciphered by bioinformatics, and spatiotemporal expressions in tissues and corms determined by fluorescence quantitative PCR.Results CwSBEII had an ORF length of 2 547 bp encoding 848 amino acids. The molecular weight of this unstable hydrophilic protein was 96 100.52 Da with a theoretical pI of 5.24, an instability index of 41.07, and a GRAVY reading at −0.473. It was closely related to and clustered with the SBEIIs of wheat (Triticum aestivum), rice (Oryza sativa), and maize (Zea Mays) of the grass family. Although found in the roots, leaves, peels as well as corm, CwSBEII was most highly expressed in the leaves and significantly upregulated in the early stage of corm expansion of Chinese water chestnut.Conclusion CwSBEII was specifically expressed in various tissues of Chinese water chestnut and most highly in the leaves. It was significantly upregulated during the early stage of corm expansion and might play an important role in the starch synthesis of E. tuberosa.
-
Keywords:
- Eleocharis tuberosa /
- CwSBEII /
- cloning /
- gene expression
-
0. 引言
【研究意义】荸荠(Eleocharis tuberosa)是中国特色农产品,在福建、浙江和广西等南方大部分地区均有种植。荸荠俗称马蹄,是莎草科(Cyperaceae Juss)荸荠属多年生浅水草本植物,球茎清脆多汁,富含碳水化合物、蛋白质等多种营养物质[1, 2]。荸荠碳水化合物是荸荠球茎的主要营养物质,并以淀粉的形式储存于其中,淀粉含量在不同品种以及不同膨大时期均存在明显差异。有研究发现广西平乐荸荠的淀粉含量比广西灵川荸荠等品种淀粉含量高[3];朱世东[4]发现荸荠球茎膨大阶段大致分3个时期:膨大前期(1~20 d)、膨大中期(21~40 d)、膨大后期(41~70 d)。细胞的分裂和膨大是荸荠球茎宏观上体积增大的原因,而在膨大的过程中淀粉颗粒首先从球茎的基部中间位置的薄壁细胞开始积累,慢慢扩散至球茎顶部和周围的细胞,最后充满整个球茎的薄壁细胞[5]。有研究指出,在荸荠球茎开始膨大后约50 d时,球茎干物质含量增加主要来自薄壁细胞中淀粉的积累[6]。但目前有关荸荠淀粉合成相关酶及其编码基因的研究较少,深入了解该基因的功能对促进荸荠淀粉积累、提升荸荠品质具有重要意义。【前人研究进展】植物淀粉生物合成过程较为复杂,其中至少涉及ADP-葡萄糖焦磷酸化酶(ADP-glucose pyrophosphorylase,AGPase)、淀粉合成酶(Starch synthase,SS)、淀粉分支酶(Starch branching enzyme,SBE)以及脱分支酶(Debranching enzyme,DBE)等4种关键酶,其中淀粉合成酶(SBE)对支链淀粉的合成起着重要作用[7]。SBE及其多种同工酶已经从拟南芥(Arabidopsis thalian)[8]、小麦(Triticum aestivum)[9]、玉米(Zea mays)[10]以及水稻(Oryza sativa)[11]等作物中得到克隆鉴定。系统发育分析表明,高等植物中SBE根据氨基酸序列相似性可以分为SBEI和SBEII两个同工型[12]。SBEI主要负责直链淀粉长链淀粉的合成,SBEII则产生短链淀粉,主要作用于支链淀粉[13]。以往研究已经表明,高等植物中SBE突变或者缺失会导致直链淀粉和支链淀粉结构的改变。例如,通过RNA干扰技术抑制水稻中SBEI基因的表达,可使直链淀粉含量提高2倍 [14]。李苗等[15]研究发现,使用CRISPR/Cas9技术敲除小麦SBEIIa基因,可促进直链淀粉合成从而增加小麦直链淀粉含量,可见SBEIIa基因在小麦支链淀粉合成过程中发挥重要作用。Schwall等[16]也在研究中发现马铃薯SBEII基因缺失的突变体植株中,含有大量的支链淀粉而且淀粉成分也发生很大的变化。由此可见,支链淀粉的合成与SBE相关基因的表达有直接关系。同时,研究发现SBEI和SBEII在甘薯(Sweet Potato)[17]、芋头(Colocasia esculenta)[18]以及木薯(Manihot esculenta Crantz)[19]中的表达具有组织和时空特异性,表明不同SBE同工酶在不同物种的淀粉合成中发挥着不同的作用。目前已在荸荠球茎中克隆得到GBSS基因、EdAGPLI以及AGPase等3个参与淀粉合成的基因[20-22]。【本研究切入点】目前仅有少量关于荸荠球茎中淀粉生物合成的研究,而关于荸荠SBEII基因的研究至今尚未见报道。【拟解决的关键问题】本研究以桂林荸荠为对象,基于已获得的荸荠转录组测序结果,利用同源克隆的方法对筛选得到的SBEII基因进行克隆与生物信息学分析,并利用荧光定量PCR技术对荸荠不同组织以及膨大过程中球茎的SBEII基因进行表达特性分析,以期为荸荠CwSBEII的基因功能深入探究以及该基因参与淀粉合成的机制提供理论参考。
1. 材料与方法
1.1 试验材料
供试材料为地方品种桂林荸荠,采自广西壮族自治区贺州市八步区荸荠种植田,选择无虫害、无机械损伤的荸荠植株,在球茎膨大后期收集根、叶片、荸荠皮和荸荠肉以及球茎不同膨大时期(S1~S4时期,球茎最宽处直径分别约为10 、20 、35和50 mm)的样本,洗净去皮。取样时每个时期选取3个块茎大小相近的荸荠混合取样后,分装液氮,速冻于−80 ℃保存备用。
1.2 试验方法
1.2.1 总RNA的提取和各样品的cDNA合成
参照华越洋生物科技(北京) 有限公司的植物RNA提取试剂盒提取各样品的总RNA。RNA的浓度通过超微量分光光度计检测分析。选择浓度合适的RNA根据PrimeScriptTM RT reagent Kit with gDNA Eraser反转录试剂盒(TaKaRa,大连)进行单链cDNA的合成,具体操作步骤参照说明书,将合成的cDNA模板保存于−20 ℃冰箱中,用于后续试验。
1.2.2 基因克隆
根据之前从荸荠转录组数据中得到的与其他植物SBEII基因同源性较高的序列片段(转录组中编号为isoform_22367),利用primer 5.0软件设计用于扩增该基因CDS序列的引物,其引物序列见表1。以不同膨大时期的荸荠cDNA为模板进行PCR扩增,反应体系为50 μL,包含cDNA模板1 μL,上下游引物各1 μL,酶25 μL,ddH2O 22 μL。反应扩增程序:94 ℃预变性3 min,95 ℃变性15 s,55 ℃ 退火30 s,72 ℃延伸 3 min,35个循环后72 ℃终延伸10 min,产物于4 ℃保存。
表 1 荸荠SBEII基因克隆和表达所用引物Table 1. Primer used for cloning and expression analysis of SBEII in Chinese water chestnut引物名称
Primer name引物序列(5′-3′)
Sequence(5′-3′)CwSBEII (F) ATGACGTTCGCTCTATCGGGATCGG CwSBEII (R) TTACTCCTCACAGAGAGCATAGACA Q-PCR CwSBEII (F) CCTCCTGAAGAAGAAAAGTACGTC Q-PCR CwSBEII (R) AGCTAGCATAGTAAGAGTGCTCCTG 18S RNA (F) ATGATTAAGAGGGACAGTCGGGGGC 18S RNA (R) CTAGGACGGTATCTGATCGTCTTCG PCR扩增产物经过琼脂糖凝胶电泳检测后切胶回收纯化,回收方法参照DNA凝胶回收试剂盒说明书。回收纯化后的产物与PMD 18-T克隆载体连接,并通过热激法转化至DH5α感受态细胞中,涂布在含有抗性的LB固态培养基上,37 ℃培养12~16 h后挑取单克隆进行摇菌,经菌液PCR检测后,将阳性克隆菌样送至上海生工生物工程有限公司完成测序,测序正确序列用于后续生物信息学分析。
1.3 SBEII基因生物信息学分析
通过ORF finder(http://www.detaibio.com/sms2/orf_find.html)网站查找CwSBEII基因的开放阅读框(ORF);分别使用ClustaW 1.83和DNAMAN软件进行多重序列比对和同源性比对;在ExPASy(https://web.expasy.org/protparam/)网站分析蛋白分子量及理化性质;利用NPS@:SOPMA secondary structure prediction(https://npsa-prabi.ibcp.fr/cgi-bin/npsa_automat.pl?page=npsa_sopma.html)和SWISS-MODEL(https://swissmodel.expasy.org/)网站分别预测蛋白质二级结构、三级结构;利用Mega 5软件构建系统进化树[21-22]。
1.4 SBEII基因的表达分析
通过荧光定量PCR法对目的基因相对表达量进行检测,以18S rRNA(登录号:MG742686)为内参。根据获得的SBEII基因cDNA序列设计荧光定量PCR特异引物(表1)。以荸荠根、叶片、荸荠皮、荸荠肉等组织以及不同膨大时期荸荠肉的cDNA为模板,采用PrimeScriptTM RT reagent Kit剂盒进行荧光定量PCR扩增,反应体系为25 μL,包含上下游引物各1 μL,cDNA模板2 μL,酶12.5 μL,ddH2O 8.5 μL,具体步骤参考试剂盒说明书。利用2−△△Ct方法计算荸荠SBEII基因在球茎不同膨大时期和不同组织中的相对表达量。每个样品的表达量均为3次重复的平均值。
2. 结果与分析
2.1 荸荠SBEII基因克隆
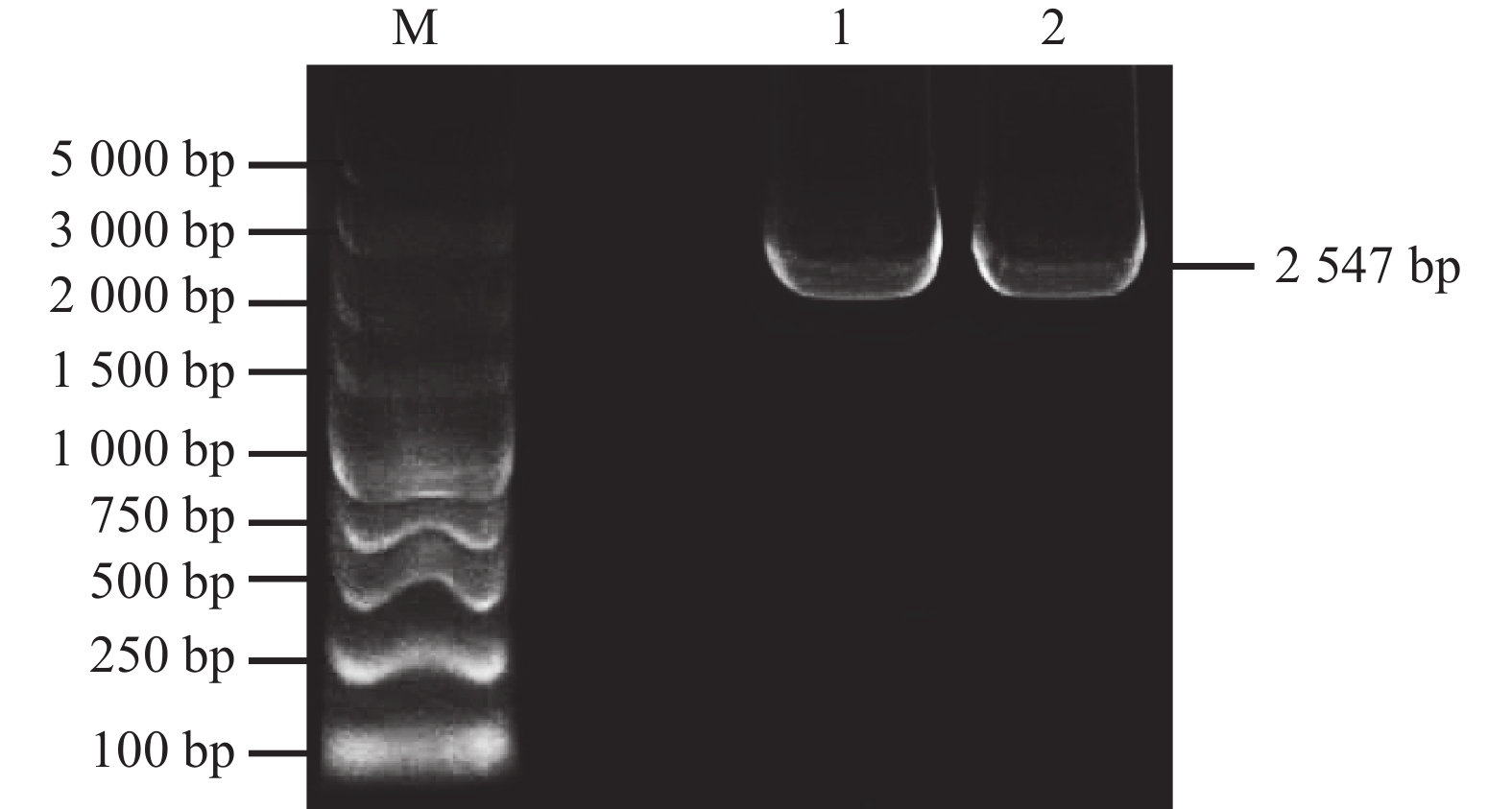
在荸荠球茎全长转录组数据中筛选出转录本isoform_22367,该转录本在各大数据库均注释为SBE,在NCBI官网上进行BLAST比对确认。该序列作为参照设计PCR特异性引物,以荸荠组织cDNA为模板进行PCR扩增,经琼脂糖凝胶电泳检测发现获得产物长度约为2500 bp(图1)。将该产物回收后与载体连接并测序,得到该基因序列,并与转录本序列进行比对,序列完全一致,最终获得该基因的开放阅读框(ORF)长为2547 bp。将该基因编码区全长序列在NCBI上进行比对,发现该序列与其他多种植物SBEII基因的同源性大于77 %。由此确定该基因为SBEII同源基因,并命名为CwSBEII。
2.2 CwSBEII基因的生物信息学分析
通过在线软件Expasy-Prot Param Tool分析CwSBEII基因的氨基酸序列,结果表明,该序列含有848个氨基酸残基,分子表达式为C4294H6447N1151O1297S36,分子量为96100.52,理论等电点pI值为5.24。不稳定指数为41.07,为不稳定蛋白。CwSBEII氨基酸组成中丝氨酸(Ser)最多,为8.7 %,其次是甘氨酸(Gly),占8.5 %,并且此蛋白质中不含吡咯赖氨酸(Pyl)和硒半胱氨酸(Sec)。其中带正电荷总数为81个,带负电荷酸性氨基酸为117个,即整个蛋白质带负电。CwSBEII蛋白的脂肪族氨基酸指数为67.37。
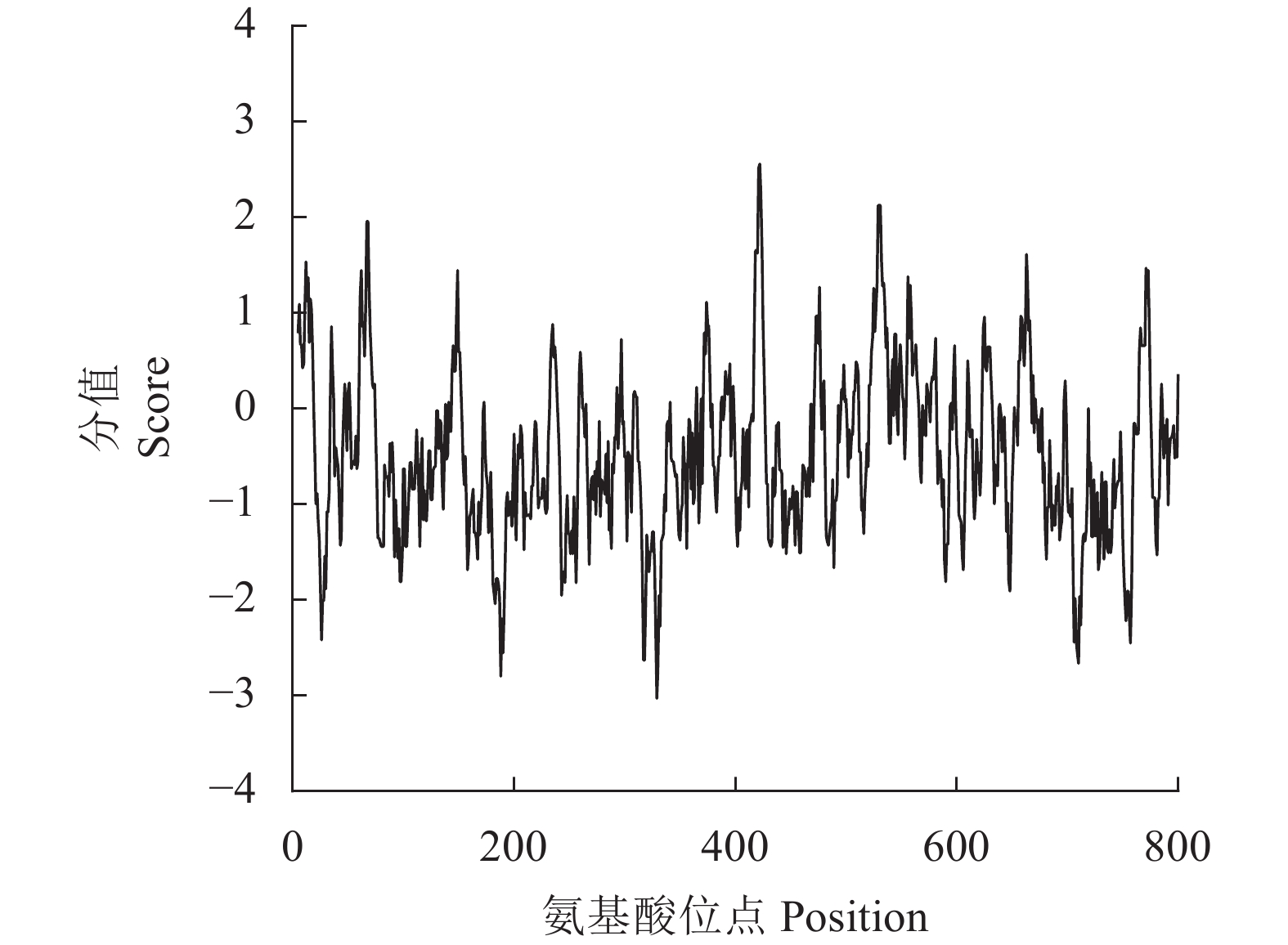
通过在线软件ProtScale对CwSBEII基因编码的蛋白质进行亲疏水性预测分析(图2),发现超过一半图像位于负坐标,通过计算亲水性总平均值(GRAVY)为−0.473,表明该CwSBEII蛋白属于亲水性蛋白。
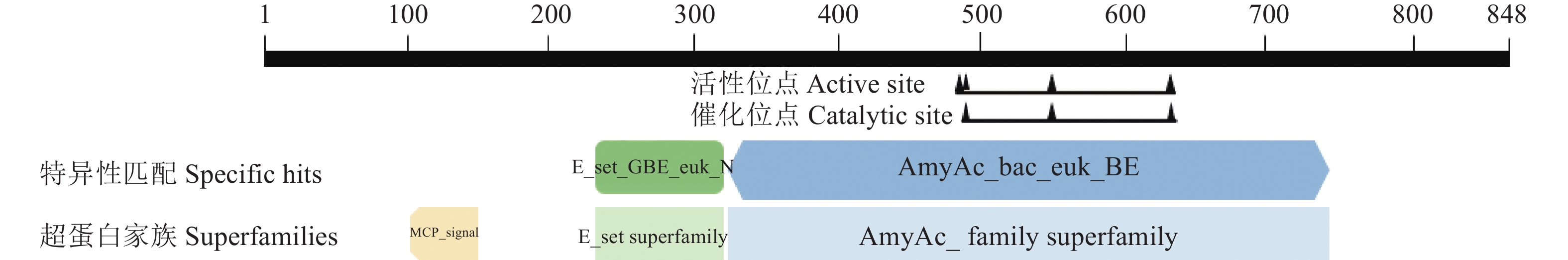
在 NCBI 的 Conserved domains 数据库预测 CwSBEII基因的蛋白保守结构域,发现CwSBEII蛋白含有AmyAc_bac_euk_BE和E_set_GBE_euk_N两个保守结构域,分别属于AmyAc_family 超家族、MCP_signal 超家族(图3)。
利用ExPaSy-SOPMA对CwSBEII蛋白进行二级结构预测(图4),结果显示该蛋白含有33.14%的α-螺旋、6.01%的β-折叠、20.40%的延伸链以及40.45%的无规则卷曲。利用Swiss-model以系统自动匹配水稻为模板对CwSBEII蛋白进行同源建模(图5),CwSBEII的三级结构中β-折叠是最明显的结构组件。另外利用Plant-mPLoc server在线软件预测得知荸荠叶绿体中存在CwSBEII基因编码的蛋白质。
2.3 SBEII氨基酸多重序列比对和系统发育进化树的构建
为进一步了解CwSBEII基因的功能和特点,使用ClustaW 1.83和Mega 5生物软件,对荸荠和其他11个物种的SBEII氨基酸序列进行系统发育进化树的构建(图6)。不同SBE主要分为两簇,其中绿豆(Vigna radiata)和菜豆(Phaseolus vulgaris)等6个物种聚为一簇,拟南芥和荸荠等5个物种聚为一簇。说明荸荠、拟南芥、小麦、水稻、玉米等5个物种的SBE蛋白之间亲缘关系较近,与绿豆、菜豆等亲缘关系较远。
将荸荠SBEII基因所编码的蛋白序列与其他5种植物的SBEII蛋白进行同源比对(图7),结果显示CwSBEII编码的氨基酸序列与拟南芥、小麦、水稻和玉米SBEII蛋白的同源性依次为68.09%、75.65%、76.84%、77.16%。表明SBEII在不同物种中具有较高的保守性,特别是7个已知的蛋白活性位点(图7)。
2.4 CwSBEII组织特异性表达分析
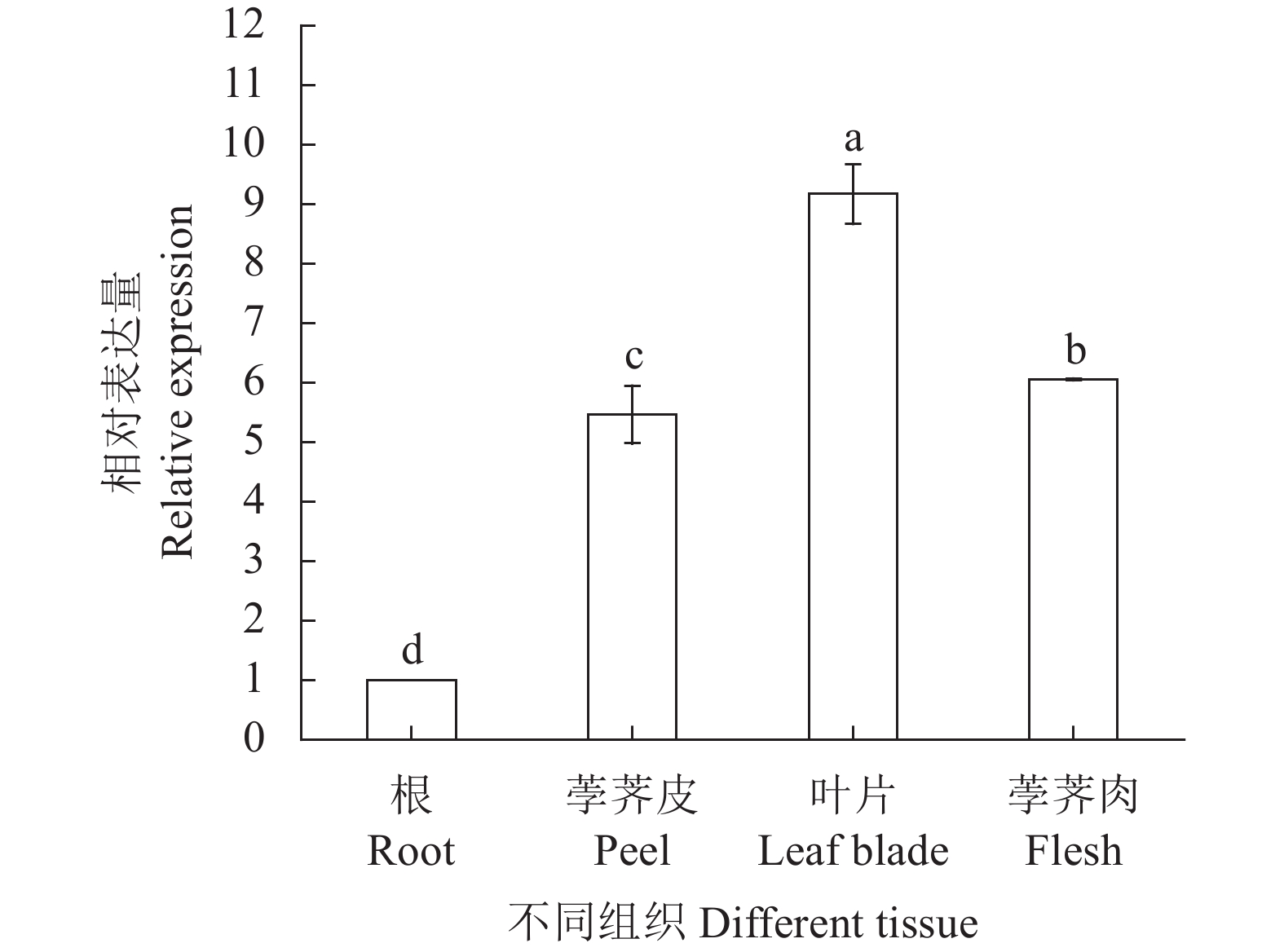
为了研究CwSBEII在荸荠不同组织器官中的作用,利用荧光定量PCR方法分析其在叶片、根、荸荠皮和荸荠肉中的表达模式。结果表明CwSBEII基因在荸荠各组织中均有表达,其中CwSBEII在叶片中表达量最高,在荸荠皮和肉中也均有较高的表达水平,而在根中的表达量最低,不同组织之间的相对表达量均差异显著(P<0.05)(图8)。
2.5 CwSBEII在球茎膨大过程中的表达水平分析
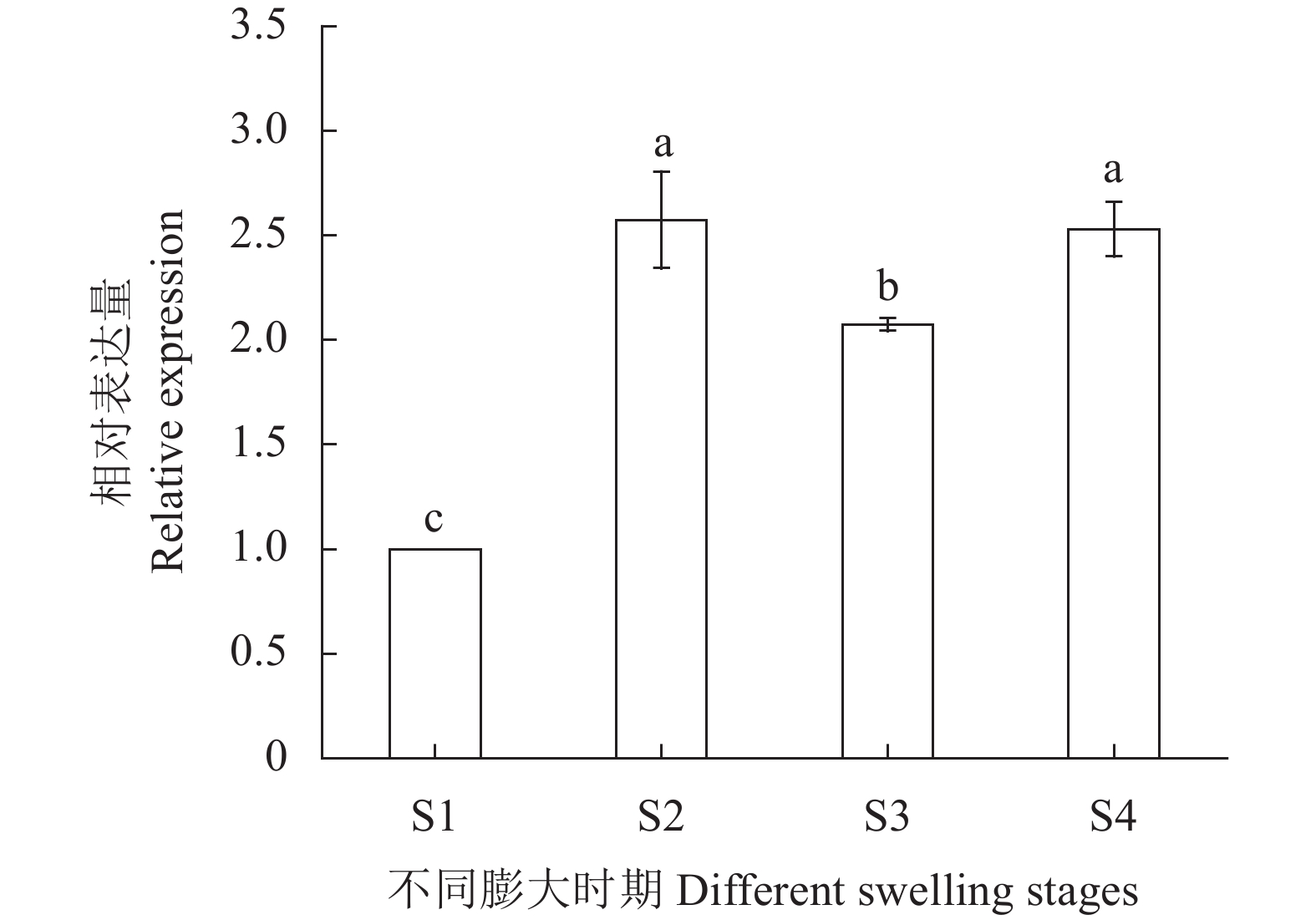
为了揭示CwSBEII在荸荠球茎膨大过程中的作用,利用荧光定量PCR方法分析其在不同膨大时期的表达模式。从图9可以看出,CwSBEII在荸荠球茎膨大的S2和S4时期相对表达量较高,并且显著高于其他时期(P<0.05),球茎膨大前期(S1时期)表达量显著低于其他3个时期(P<0.05)。
3. 讨论与结论
园艺作物的球茎、块茎和块根是重要的经济器官,积累大量的营养物质。山药块茎膨大过程中,块茎质量、还原糖和淀粉含量随着块茎的膨大呈现先逐渐增加后快速增大至趋于稳定或者缓慢下降的趋势[23]。不同葛根品种块根的淀粉含量在膨大前期和后期呈现快速增长的趋势,且在膨大后期达到最大值后趋于稳定的变化趋势[24]。慈姑球茎膨大过程中,球茎淀粉含量表现出“缓慢-快速-缓慢”增长的变化趋势,表明淀粉是慈姑球茎的主要组成物质[25]。荸荠作为典型的球茎植物,其球茎含有丰富的淀粉类碳水化合物。荸荠球茎中富含淀粉,一些“粉蹄”品种的淀粉含量更高,但荸荠球茎膨大过程中淀粉的积累规律和研究机制尚不清晰。目前,已经在莲藕[26]、马铃薯[27]、山药[23]以及芭蕉芋[28]等块茎植物和水仙[29]等球茎植物中克隆出许多编码淀粉合成的基因,如GBSS、SSS、SBE。但是,关于SBEII参与荸荠球茎淀粉合成的报道尚未报道。
本研究发现,荸荠CwSBEII的开放阅读框包含2547 bp,编码848个氨基酸,CwSBEII蛋白的长度与板栗[30]接近但短于慈姑[25]的SBEII蛋白。经预测发现,荸荠CwSBEII蛋白属于亲水性不稳定蛋白,主要分布在叶绿体中。多种植物的SBEII蛋白具有AmyAc_bac_euk_BE和E_set_GBE_euk_N两个保守结构域,其中AmyAc_bac_euk_BE是α-淀粉酶催化活性中心,可以水解淀粉中的α-1,4糖苷键[30-32]。荸荠CwSBEII蛋白也属于α-淀粉酶超家族,并具有相似的两个保守结构域,表明该蛋白具有淀粉酶催化活性。前人研究发现SBEII与SBEI相比有一个较长的N端,SBEII对支链淀粉的活性大于对直链淀粉的活性,SBEI刚好相反[33],这些均表明CwSBEII是一种典型的淀粉分支酶[25]。序列比对结果显示,荸荠SBEII编码的氨基酸序列与禾本科的其他物种SBEII有较高的序列一致性,尤其是关键酶活性位点Asp312、His317、Arg381、Asp383、Glu438、His505、Asp506的氨基酸残基完全相同[34]。经系统进化树分析发现,荸荠与水稻OsSBEII、玉米ZmSBEII等禾本科聚为一支,与其亲缘关系较近,说明该基因确实为桂林荸荠中的SBEII,并且在进化过程中具有较高的保守性。
木薯SBEII在叶片和块根中发挥功能,并具有较高的表达水平[19]。然而马铃薯SBEI专一表达于块茎中,而SBEII则相对专一表达于叶片中[35]。这表明SBEI和SBEII的表达模式具有明显的组织特异性。本研究通过荧光定量PCR技术分析了CwSBEII在荸荠各组织器官以及球茎不同膨大时期的表达模式,结果显示,CwSBEII在荸荠中表现为组织特异性表达,在荸荠叶片组织中的表达量最高,在球茎皮和球茎肉中也有较高的表达水平,推测该基因可能参与荸荠叶片和荸荠球茎发育的调控。除了组织特异性表达,SBE基因在多种植物中也呈现明显的时空特异性表达。本研究发现,CwSBEII在球茎S1~S4膨大时期表达量呈现出“上升-下降-上升”的变化趋势,特别是在S1~S2的膨大初期CwSBEII表达出现显著上调,推测该基因可能参与荸荠球茎淀粉的合成。
-
表 1 荸荠SBEII基因克隆和表达所用引物
Table 1 Primer used for cloning and expression analysis of SBEII in Chinese water chestnut
引物名称
Primer name引物序列(5′-3′)
Sequence(5′-3′)CwSBEII (F) ATGACGTTCGCTCTATCGGGATCGG CwSBEII (R) TTACTCCTCACAGAGAGCATAGACA Q-PCR CwSBEII (F) CCTCCTGAAGAAGAAAAGTACGTC Q-PCR CwSBEII (R) AGCTAGCATAGTAAGAGTGCTCCTG 18S RNA (F) ATGATTAAGAGGGACAGTCGGGGGC 18S RNA (R) CTAGGACGGTATCTGATCGTCTTCG -
[1] 孔进喜, 韩文芳, 吕广英, 等. 荸荠食品加工研究进展 [J]. 保鲜与加工, 2011, 11(1):43−46. DOI: 10.3969/j.issn.1009-6221.2011.01.013 KONG J X, HAN W F, LV G Y, et al. Research progress of Chinese water chestnut processing [J]. Storage & Process, 2011, 11(1): 43−46.(in Chinese) DOI: 10.3969/j.issn.1009-6221.2011.01.013
[2] 赵广河, 陈振林. 鲜切荸荠研究进展 [J]. 食品研究与开发, 2012, 33(1):197−200. DOI: 10.3969/j.issn.1005-6521.2012.01.055 ZHAO G H, CHEN Z L. Study on fresh-cut Chinese water chestnut [J]. Food Research and Development, 2012, 33(1): 197−200.(in Chinese) DOI: 10.3969/j.issn.1005-6521.2012.01.055
[3] 江文, 李杨瑞, 杨丽涛, 等. 荸荠球茎主要性状观察及营养品质分析 [J]. 中国蔬菜, 2009(2):51−54. JIANG W, LI Y R, YANG L T, et al. Study on agronomic characters and nutrition of Chinese water-chest nut(Eleocharis tuberosa) [J]. China Vegetables, 2009(2): 51−54.(in Chinese)
[4] 朱世东. 荸荠球茎膨大生理的研究 [J]. 安徽农业科学, 1996, 24(1):51−54. ZHU S D. Physiological studies on corm expansion of Chinese water- chestnut (Eleochatis tuberosa Schult) [J]. Journal of Anhui Agricultural Sciences, 1996, 24(1): 51−54.(in Chinese)
[5] 施国新, 常福辰, 徐祥生. 荸荠匍匐茎的发育和球茎的膨大研究 [J]. 南京师大学报(自然科学版), 1989, 12(4):71−76. SHI G X, CHANG F C, XU X S. Studies on stolon development and corm enlargement of Eleocharis tuberosa [J]. Journal of Nanjing Normal University (Natural Science Edition), 1989, 12(4): 71−76.(in Chinese)
[6] 徐国鑫, 李效尊, 杨百战, 等. 荸荠球茎膨大及淀粉积累规律研究 [J]. 长江蔬菜, 2015(22):81−83. DOI: 10.3865/j.issn.1001-3547.2015.22.029 XU G X, LI X Z, YANG B Z, et al. Study on expansion and amyloids accumulation of water chestnut corm [J]. Journal of Changjiang Vegetables, 2015(22): 81−83.(in Chinese) DOI: 10.3865/j.issn.1001-3547.2015.22.029
[7] 马岚, 李育先, 刘颖, 等. 谷子淀粉合成酶家族分析 [J]. 分子植物育种, 2020, 18(16):5213−5220. DOI: 10.13271/j.mpb.018.005213 MA L, LI Y X, LIU Y, et al. Analysis of starch synthase family in millet [J]. Molecular Plant Breeding, 2020, 18(16): 5213−5220.(in Chinese) DOI: 10.13271/j.mpb.018.005213
[8] FISHER D K, GAO M, KIM K N, et al. Two closely related cDNAs encoding starch branching enzyme from Arabidopsis thaliana [J]. Plant Molecular Biology, 1996, 30(1): 97−108. DOI: 10.1007/BF00017805
[9] 赵法茂, 毕建杰, 李天骄, 等. 小麦籽粒淀粉分支酶同工酶基因型与酶活性关系研究 [J]. 作物学报, 2007, 33(11):1850−1855. DOI: 10.3321/j.issn:0496-3490.2007.11.018 ZHAO F M, BI J J, LI T J, et al. Relationship between isozyme genotypes and activities of starch branching enzyme in wheat grain [J]. Acta Agronomica Sinica, 2007, 33(11): 1850−1855.(in Chinese) DOI: 10.3321/j.issn:0496-3490.2007.11.018
[10] 徐亚维, 王丕武, 柴晓杰. 玉米淀粉分支酶sbeIIb基因启动子的克隆与特异表达 [J]. 安徽农业科学, 2010, 38(36):20538−20541. DOI: 10.3969/j.issn.0517-6611.2010.36.013 XU Y W, WANG P W, CHAI X J. Cloning and expression of the promoter of maize starch-branching enzyme sbeIIb gene [J]. Journal of Anhui Agricultural Sciences, 2010, 38(36): 20538−20541.(in Chinese) DOI: 10.3969/j.issn.0517-6611.2010.36.013
[11] 陈秀花, 刘巧泉, 吴信淦, 等. 水稻分支酶基因Sbe1和Sbe3 cDNA的克隆及全序列分析 [J]. 中国水稻科学, 2003, 17(2):109−112. DOI: 10.3321/j.issn:1001-7216.2003.02.004 CHEN X H, LIU Q Q, WU X G, et al. cDNA cloning and sequence analysis of rice Sbe1 and Sbe3 genes [J]. Chinese Journal of Rice Science, 2003, 17(2): 109−112.(in Chinese) DOI: 10.3321/j.issn:1001-7216.2003.02.004
[12] 高振宇, 黄大年. 植物支链淀粉合成的关键酶: 淀粉分支酶 [J]. 生物工程进展, 1998, 18(6):29−31. GAO Z Y, HUANG D N. Starch branching enzyme: The key enzyme of amylopectin biosynthesis in plants [J]. Progress in Biotechnology, 1998, 18(6): 29−31.(in Chinese)
[13] BÅGA M, NAIR R B, REPELLIN A, et al. Isolation of a cDNA encoding a granule-bound 152-kilodalton starch-branching enzyme in wheat [J]. Plant Physiology, 2000, 124(1): 253−264. DOI: 10.1104/pp.124.1.253
[14] 张鹏. 抑制淀粉分支酶类基因表达对稻米品质影响的研究[D]. 扬州: 扬州大学, 2008. ZHANG P. Transgenic-mediated knockdown of the expression of genes encoding starch branching enzymes and its effects on grain quality in rice (Oryza sativa L. )[D]. Yangzhou: Yangzhou University, 2008. (in Chinese)
[15] 李苗, 陈晓军, 马斯霜, 等. 小麦淀粉分支酶CRISPR/Cas9基因敲除系统gRNA表达载体构建 [J]. 分子植物育种, 2019, 17(20):6668−6672. DOI: 10.13271/j.mpb.017.006668 LI M, CHEN X J, MA S S, et al. Expression vector construction of starch branching enzyme gRNA with CRISPR/Cas9 gene knockout system of Triticum aestivum L [J]. Molecular Plant Breeding, 2019, 17(20): 6668−6672.(in Chinese) DOI: 10.13271/j.mpb.017.006668
[16] SCHWALL G P, SAFFORD R, WESTCOTT R J, et al. Production of very-high-amylose potato starch by inhibition of SBE A and B [J]. Nature Biotechnology, 2000, 18(5): 551−554. DOI: 10.1038/75427
[17] QIN H, ZHOU S, ZHANG Y Z. Characterization and expression analysis of starch branching enzymes in sweet potato [J]. Journal of Integrative Agriculture, 2013, 12(9): 1530−1539. DOI: 10.1016/S2095-3119(13)60369-X
[18] 顾绘, 陈赛男, 李良俊, 等. 芋淀粉分支酶SBE基因的克隆与表达分析 [J]. 园艺学报, 2016, 43(10):2049−2058. GU H, CHEN S N, LI L J, et al. Cloning and expression analysis of starch branching enzyme gene in taro(Colocasia esculenta) [J]. Acta Horticulturae Sinica, 2016, 43(10): 2049−2058.(in Chinese)
[19] 裴金利, 陈新, 夏志强, 等. 木薯淀粉分支酶基因MeSBE2.2的克隆和表达分析 [J]. 中国农业大学学报, 2015, 20(4):36−41. DOI: 10.11841/j.issn.1007-4333.2015.04.05 PEI J L, CHEN X, XIA Z Q, et al. Cloning and expression analysis of starch branching enzyme gene(MeSBE2.2)in cassava [J]. Journal of China Agricultural University, 2015, 20(4): 36−41.(in Chinese) DOI: 10.11841/j.issn.1007-4333.2015.04.05
[20] 俞梅珍. 荸荠颗粒结合型淀粉合成酶基因的克隆与表达分析[D]. 扬州: 扬州大学, 2017. YU M Z. Cloning and expression analysis of granule-bound starch synthase gene in Chinese water chestnut (Eleocharis tuberosa)[D]. Yangzhou: Yangzhou University, 2017. (in Chinese)
[21] 何芳练, 邱祖杨, 董伟清, 等. 荸荠EdAGPL1基因的克隆及表达分析 [J]. 分子植物育种, 2021, 19(17):5654−5661. HE F L, QIU Z Y, DONG W Q, et al. Cloning and expression analysis of EdAGPL1 gene in Eleocharis dulcis [J]. Molecular Plant Breeding, 2021, 19(17): 5654−5661.(in Chinese)
[22] 董伟清, 江文, 何芳练, 等. 荸荠淀粉合成酶AGPase的基因克隆及表达分析 [J]. 西南农业学报, 2021, 34(5):956−963. DONG W Q, JIANG W, HE F L, et al. Gene cloning and expression analysis of ADP-glucose pyrophosphorylase in Eleocharis dulcis [J]. Southwest China Journal of Agricultural Sciences, 2021, 34(5): 956−963.(in Chinese)
[23] 罗海玲, 龚明霞, 周芸伊, 等. 山药块茎发育过程中淀粉积累及差异蛋白分析 [J]. 华南农业大学学报, 2018, 39(6):61−69. DOI: 10.7671/j.issn.1001-411X.2018.06.010 LUO H L, GONG M X, ZHOU Y Y, et al. Analysis of starch accumulation and differentially expressed proteins during the development of Chinese yam tuber [J]. Journal of South China Agricultural University, 2018, 39(6): 61−69.(in Chinese) DOI: 10.7671/j.issn.1001-411X.2018.06.010
[24] 郭丽君, 羽健宾, 肖冬, 等. 葛根淀粉合成关键酶活性动态及其与块根产量和淀粉积累的相关性研究 [J]. 广西植物, 2022, 42(4):639−647. DOI: 10.11931/guihaia.gxzw202006013 GUO L J, YU J B, XIAO D, et al. Dynamics of key enzyme activity in starch synthesis and its correlation with yield and starch accumulation of root tubers in Kudzu [J]. Guihaia, 2022, 42(4): 639−647.(in Chinese) DOI: 10.11931/guihaia.gxzw202006013
[25] 陈赛男. 慈姑淀粉分支酶SBE基因的克隆与表达分析[D]. 扬州: 扬州大学, 2016. CHEN S N. Cloning and expression analysis of starch branching enzyme gene in arrowhead (Sagittaria sagittifolia L. )[D]. Yangzhou: Yangzhou University, 2016. (in Chinese)
[26] 陆叶, 李良俊, 梁国华, 等. 莲藕GBSS基因cDNA全长的克隆与序列分析 [J]. 长江蔬菜, 2010(14):19−23. DOI: 10.3865/j.issn.1001-3547.2010.14.006 LU Y, LI L J, LIANG G H, et al. Analysis on clone and sequence of GBSS gene's cDNA in lotus root (Nelumbo nucifera gaertn) [J]. Journal of Changjiang Vegetables, 2010(14): 19−23.(in Chinese) DOI: 10.3865/j.issn.1001-3547.2010.14.006
[27] 杨涛. 马铃薯淀粉合成酶Ⅲ基因cDNA的克隆及RNA干扰载体的构建[D]. 兰州: 甘肃农业大学, 2009. YANG T. Cloning of starch synthase Ⅲ cDNA from potato and construction of RNA interference vector[D]. Lanzhou: Gansu Agricultural University, 2009. (in Chinese)
[28] 杨龙, 付瑜华, 罗春芳, 等. 芭蕉芋块茎颗粒结合淀粉合成酶I基因的克隆与序列分析 [J]. 分子植物育种, 2020, 18(22):7376−7384. YANG L, FU Y H, LUO C F, et al. Cloning and sequence analysis of the granule-bound starch synthase I gene of cannas delis Ker [J]. Molecular Plant Breeding, 2020, 18(22): 7376−7384.(in Chinese)
[29] 董建涛, 张璐, 姚馨婷, 等. 中国水仙可溶性淀粉合成酶基因NtSSS的克隆和表达分析 [J]. 分子植物育种, 2018, 16(21):6915−6920. DONG J T, ZHANG L, YAO X T, et al. Cloning and expression analysis of soluble starch synthase gene NtSSS in Narcissus tazetta var. chinensis [J]. Molecular Plant Breeding, 2018, 16(21): 6915−6920.(in Chinese)
[30] 陈良珂. 板栗坚果淀粉积累规律及淀粉分支酶的基因克隆与分析[D]. 北京: 北京农学院, 2017. CHEN L K. Identification of starch branching enzymes involved in starch synthesis and starch accumulation during the development of chestnut(Castanea mollissima blume)cotyledons[D]. Beijing: Beijing University of Agriculture, 2017. (in Chinese)
[31] MACGREGOR E A, JANECEK S, SVENSSON B. Relationship of sequence and structure to specificity in the alpha-amylase family of enzymes [J]. Biochimica et Biophysica Acta, 2001, 1546(1): 1−20. DOI: 10.1016/S0167-4838(00)00302-2
[32] HIZUKURI S. Relationship between the distribution of the chain length of amylopectin and the crystalline structure of starch granules [J]. Carbohydrate Research, 1985, 141(2): 295−306. DOI: 10.1016/S0008-6215(00)90461-0
[33] MUTISYA J, SATHISH P, SUN C X, et al. Starch branching enzymes in Sorghum (Sorghum bicolor) and barley (Hordeum vulgare): Comparative analyses of enzyme structure and gene expression [J]. Journal of Plant Physiology, 2003, 160(8): 921−930. DOI: 10.1078/0176-1617-00960
[34] LARSSON C T, KHOSHNOODI J, EK B, et al. Molecular cloning and characterization of starch-branching enzyme II from potato [J]. Plant Molecular Biology, 1998, 37(3): 505−511. DOI: 10.1023/A:1005908305456
[35] JOBLING S A, SCHWALL G P, WESTCOTT R J. A minor form of starch branching enzyme in potato (Solanum tuberosum L. ) tubers has a major effect on starch structure: Cloning and characterisation of multiple forms of SBEA [J]. The Plant Journal:for Cell and Molecular Biology, 1999, 18(2): 163−171. DOI: 10.1046/j.1365-313X.1999.00441.x
-
期刊类型引用(1)
1. 宋慕波,孙翊然,刘英健,农锦仙,刘云芬,何妹英,殷菲胧,陈振林. 米糠提取物处理对鲜切荸荠黄化的影响. 广东农业科学. 2024(01): 1-9 .  百度学术
百度学术
其他类型引用(2)




 下载:
下载: