Codon Usage Bias of Chloroplast Genome in Liparis bootanensis
-
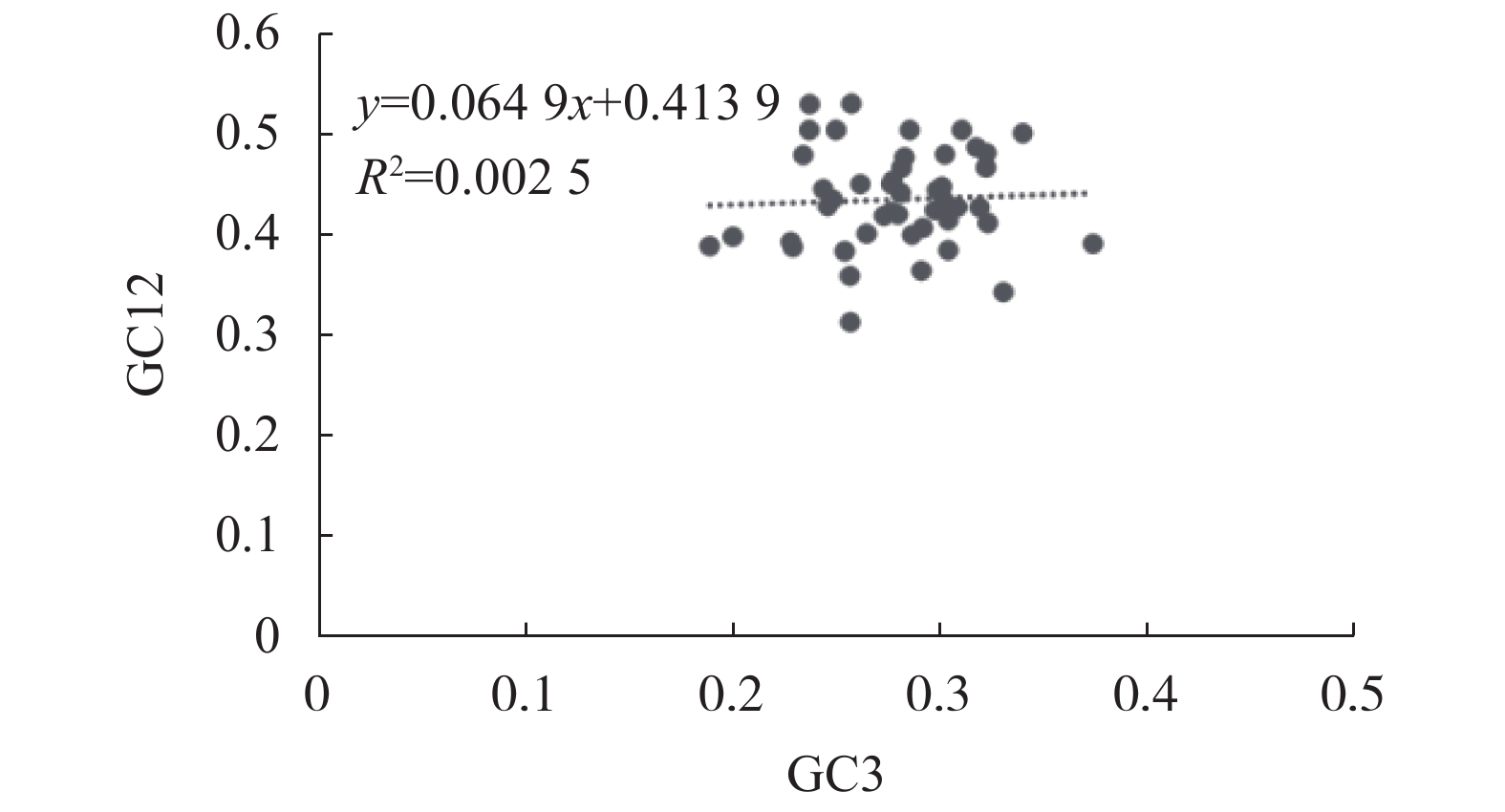
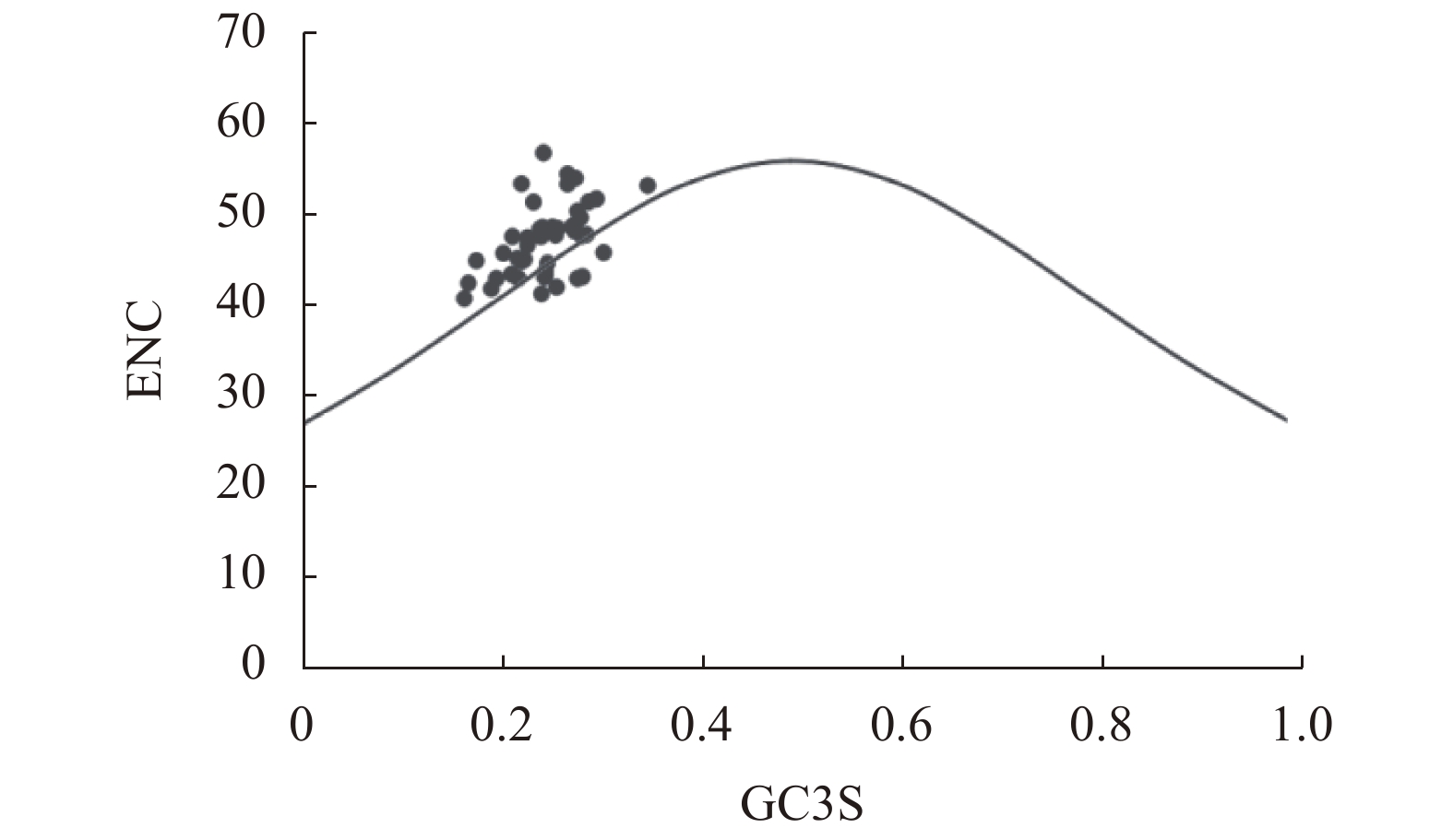
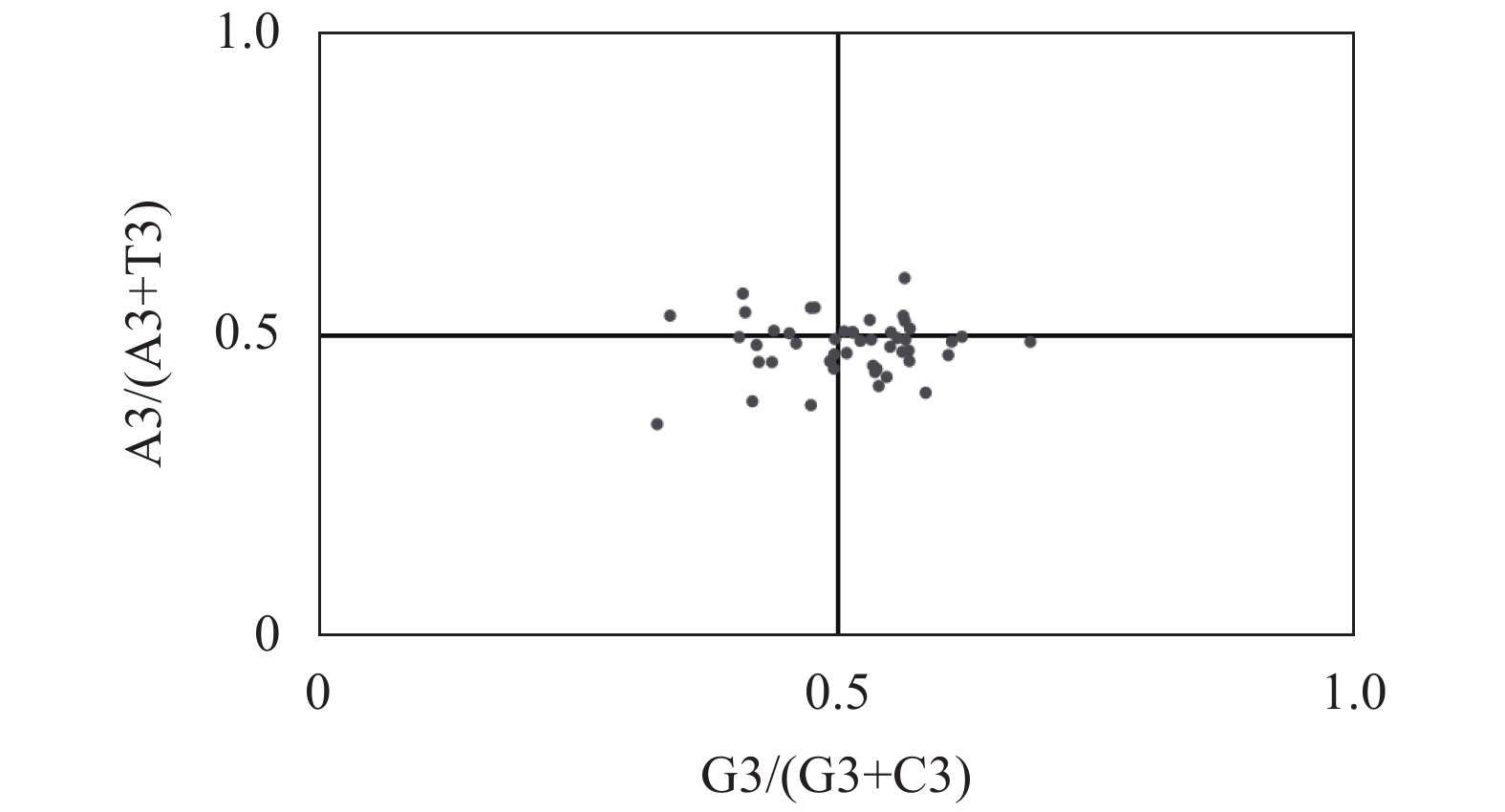
摘要:目的 分析镰翅羊耳蒜叶绿体基因组密码子偏好性,并探讨影响密码子偏好性形成的主要因素。方法 从镰翅羊耳蒜叶绿体基因组中筛选出47条编码序列,通过CodonW和CUSP软件计算不同基因的GC含量,分析密码子使用模式。结果 GC3含量为27.99%,低于GC1(47.00%)和GC2(39.41%);ENC取值范围为40.85~56.80,平均值为47.27;ENC与GC1、GC2之间均无显著相关,与GC3呈极显著正相关;GC12与GC3的相关性不显著,相关系数为0.05;47%基因的ENC比值频数分布在−0.05~0.05区间,53%基因的ENC比值频数分布在−0.05~0.05区间之外;碱基的使用频率为A>T、G>C;17个密码子被确定为最优密码子。结论 镰翅羊耳蒜叶绿体基因组密码子对A或T碱基结尾有偏好性;密码子偏好性较弱;密码子偏好性主要受选择和突变作用共同影响,同时还受其他因素影响,是多因素综合影响的结果。Abstract:Objective The coden usage bias and its infulence factor in the chloroplast genome of Liparis bootanensis were analyzed in this study.Method CodonW and CUSP software were used to analyze the codon usage bias based on 47 codon DNA sequences of the chloroplast genome of L. bootanensis.Result The GC content in the 3rd position of the codons was 27.99%, which was lower than those in the 1st and the 2nd. The effective number of codons ranged from 40.85 to 56.80, averaging 47.27. The ENC showed no significant correlation to GC1 and GC2 but a significant positive correlation to GC3. The correlation between GC12 and GC3 was not significant with a coefficient of 0.05. The frequency of ENC ratio of 47% genes was distributed between −0.05 and 0.05, while that of 53% genes outside -0.05 and 0.05 range. A PR2-plot analysis indicated the base usage frequencies to be A>T and G>C. There were 17 codons selected after screening.Conclusion The 3rd position of codons was rich in A or T. The codon usage bias was weak, complex, and affected by mutation and selection besides other factors.
-
Keywords:
- Liparis bootanensis /
- chloroplast genome /
- codon usage bias
-
0. 引言
【研究意义】土壤是人类赖以生存的自然资源和人类与生物共栖的基地,土壤团聚体的形成及稳定性是土壤结构研究的核心内容,在很大程度上反映了土壤结构的机械稳定性和抗蚀性[1-3]。耕作措施[4]、秸秆还田[5]、施肥制度[6]等土壤管理措施均能够影响土壤团聚体的组成及其稳定性。因此,研究不同耕作措施对土壤团聚体组成及其稳定性的影响对于评价土壤生态效益及可持续利用具有重要意义。【前人研究进展】土壤团聚体通常被划分为大团聚体(>0.25 mm)和微团聚体(<0.25 mm)[7],不同粒级团聚体形成的稳定机制及其在土壤结构改善和有机碳固定中的作用不同[8]。一般认为,>0.25 mm 的团聚体(即土壤团粒结构体)是土壤中最好的结构体,可反映土壤结构的优劣,其含量高低与土壤稳定性呈正相关[9]。在农业生态系统中,耕作通常通过直接破坏土壤大团聚体结构和间接改变生物和化学因素来影响土壤团聚体使土壤团聚体稳定性降低[10-11],与常规耕作相比,免耕土壤中>0.25 mm大团聚体的团聚性较高,稳定性较高,对土壤有机碳有很强的保护作用[12],少耕能够提高土壤大团聚体稳定性从而达到促使有机碳稳定的目的,合理的耕作条件下土壤有机碳积累的主要原因是大团聚体的物理保护作用得到了保障,团聚体稳定性得以提高[13-14]。但也有研究结果表明在土壤有机质含量相对较高的水平下,耕作能促进较大粒径团聚体形成,减少较小粒径团聚体的含量[15]。【本研究切入点】目前关于耕作措施对土壤团聚体分布及其与有机碳稳定性关系的研究结果还不尽一致。【拟解决的关键问题】为此,本研究以优化土壤耕作措施为突破口,在民勤绿洲长期定位试验点开展土壤团聚体组成及其与有机碳稳定性研究,以期为当地合理选择耕作措施,增强民勤绿洲区农田土壤抗蚀性、促进绿洲区农田土壤资源的可持续利用提供参考。
1. 材料与方法
1.1 试验区概况
试验区建于2015年,位于民勤绿洲区的苏武乡(103°07′00.16″E,38°37′10″N)泉水村,占地面积13340 m2。试验区属于典型的温带干旱荒漠气候区,平均海拔1400 m,冬冷夏热,昼夜温差大,年均温度7.8 ℃,最大昼夜温差25.2 ℃;光热资源丰富,年均无霜期162 d,年均日照时数2799.40 h;该区降水稀少,年均降水量113.2 mm,蒸发量高达2644 mm,降水主要集中在7~9月。耕作土壤为本地区开垦种植早、熟化程度高的灌淤土,厚度可达30~60 cm,耕层质地轻,土性热,保肥、保水性差。
1.2 试验设计
试验于2020年4月开展,该试验区自2015年开始设置为免耕(Tn)、少耕(Tm)、深松(Ts)和秋翻(Tf)4 种耕作措施连续定位观测点,以周边自然撂荒地为对照(CK),每处理设置3个重复。4种耕作措施分别为:(1)免耕:收获后不留茬,对土地不做耕翻,直至试验结束才更换地膜;(2)少耕:收获后不留茬,到二茬播种前不搅动土壤,播种前1周左右进行旋耕整地7.5~10 cm,覆膜播种;(3)深松:秋收后利用深松铲对耕地耕松30 cm,对土壤只松不翻,春季旋耕整地7.5~10 cm,覆膜播种;(4)秋翻:秋收后利用铧式犁耕翻土地,翻动土层深度大约为 20 cm,春季旋耕整地7.5~10 cm,覆膜播种。撂荒地(CK):试验全程不对土壤进行扰动,不进行灌水施肥,为自然风蚀土壤。土壤类型为灌淤土,土壤肥力较低,0~20 cm土层基本理化性质见表1。
表 1 试验土壤基本理化性质Table 1. Basic physiochemical properties of sampled soils耕作措施
Tillage measurespH EC/ms·cm−1 有机质
Organic
matter/(g·kg−1)全氮
Total
nitrogen/(g·kg−1)有效磷
Available
phosphorus/(mg·kg−1)速效钾
Rapidly available
potassium/(mg·kg−1)免耕 No-tillage 7.99 0.37 10.66 0.44 1.19 169.50 少耕 Less-tillage 8.20 0.34 10.02 0.36 1.12 168.30 深松 Deep loosening 8.21 0.31 8.99 0.34 1.08 163.20 秋翻 Autumn turning 8.64 0.28 7.64 0.33 1.03 162.80 撂荒地(CK) Abandoned land 8.30 0.77 6.73 0.31 0.89 146.60 试验种植作物为玉米,于每年4月20日左右播种,株行距为20 cm×30 cm,种植密度为7.2×104 株·hm−2,除耕作措施不同外,其他施肥、灌水等田间管理均相同。试验地底肥种类为尿素、磷酸二氢铵、过磷酸钙、硫酸钾,施用量各处理统一为每年施用N 180 kg·hm−2、P2O5 95 kg·hm−2、K2O 45 kg·hm−2,尿素20%作为底肥施用,剩余用作追肥,分别在拔节期至抽穗期追施20%,抽穗期至开花期追施10%,灌浆期追施50%。
1.3 测定项目与方法
1.3.1 土壤样品采集
土壤样品于2020年6月5日采集,在各小区采用五点取样法分别采集0~20 cm和20~40 cm两个深度的土样,将每个小区随机选取的5个采样点土样充分混匀成一个混合样,轻轻用手揉碎大土块,田间现场过5 mm孔径土壤筛,过筛后剔除土壤中肉眼可见的动、植物残体和砂砾。采集的土壤样品一分为二,一部分迅速冷冻处理后带回实验室置于4 ℃冰箱中保存,用于土壤团聚体分级和有机碳矿化量的测定,另一部分直接带回室内进行自然风干,用于团聚体指标的测定。
1.3.2 团聚体分级及培养
团聚体分级参考Bach和Hofmockel(2014)[16]的干筛法,并稍做修改。具体操作步骤如下:将过5 mm筛的土壤样品(<5 mm)置于4 ℃冰箱中进行冷干处理,当土壤水分达到60~70 g·kg−1时,进行筛分处理。筛分装置由三种规格(筛孔直径分别为0.25 mm、1 mm和2 mm)的土壤筛垂直套装组成,顶部筛为2 mm,中部筛为1 mm,底部筛为0.25 mm。筛分前固定好套筛,垂直上下振荡5 cm高度,震荡频率为1 次·s−1,震荡时长为1 min,筛分出>2 mm、1~2 mm、0.25~1 mm和<0.25 mm四级团聚体。
分别称取>2 mm、1~2 mm、0.25~1 mm和<0.25 mm四种不同粒径团聚体土壤样品各5 g装入50 ml硬质塑料杯中,采用称重法调整土壤含水量至60%,置于1000 ml塑料培养盒中。同时放入盛有10 ml 0.5 mol·L−1 NaOH溶液的硬质塑料杯和盛有10 ml蒸馏水的塑料杯,所有小塑料杯底部用双面胶粘贴固定,1000 ml塑料培养盒顶盖边缘使用封口膜密封。将培养盒置于25 ℃恒温培养箱中避光培养14 d收集CO2。每个粒径团聚体设置3次重复,并设空白对照。
1.3.3 测定指标及计算方法
采用干筛法[16]测定>2 mm、1~2 mm、0.25~1 mm和<0.25 mm四种不同粒径土壤团聚体含量。利用各粒径团聚体含量数据,计算土壤各粒径团聚体质量百分数(Vi)[17]、>0.25 mm团聚体含量(R0.25)[8]、土壤团聚体平均重量直径(MWD)[18]、几何平均直径(GWD)[19]、团聚体分形维数(D)。具体计算公式为:
Vi=wiwt×100% (1) MWD=n∑i=1(¯Riwi)n∑i=1wi (2) GMD=exp(n∑i=1wiln¯Ri/n∑i=1wi) (3) D=3−log(¯xixmax (4) 公式(1-3)中,
\overline {{R_i}} 是某级别团聚体平均直径(mm);{w_i} 是该级别团聚体干质量(g);{w_t} 为各粒径团聚体干质量总和(g)。团聚体分形维数(D)的计算采用杨培岭[20]和赵鹏等[17]的计算公式推导即可求得。公式(4)中\overline {{x_i}} 为某级团聚体平均直径(mm);M(r < \overline {{x_i}} ) 为粒径小于{x_i} 的团聚体的质量(g);{M_T} 为团聚体总质量(g);{x_{\max }} 为团聚体的最大粒径(mm)。土壤有机碳采用重铬酸钾法[21]测定,并计算土壤团聚体有机碳在土壤中的贡献率[22],团聚体有机碳贡献率(%)=[(该级团聚体有机碳含量×该级团聚体含量)/各层土壤有机碳含量]×100%。
土壤有机碳累积矿化量采用0.5 mol·L−1NaOH碱液吸收法培养、盐酸滴定法[23-24]测定。14 d培养结束后逐个打开培养盒取出碱液杯,用去离子水反复少量冲洗碱液到三角瓶中,依次加入过量的1.0 mol·L−1BaCl2溶液、2滴0.5%的酚酞指示剂,再用浓度为0.1 mol·L−1的HCl溶液滴定,溶液由红色变为白色即为滴定终点,记下所消耗的盐酸量,通过HCl消耗量计算CO2释放量(以每千克干土释放CO2-C的克数计)。
1.4 数据处理与分析
试验数据采用SPSS 19.0数据处理系统进行差 异显著性分析(Duncan新复极差法p<0.05),利用MicrosoftOffice Excel 2007和Origin 8.0版软件进行数据记录和作图。
2. 结果与分析
2.1 耕作措施对土壤团聚体分布及其稳定性的影响
2.1.1 耕作措施对土壤大团聚体(>0.25 mm)分布的影响
由表2可知,各耕作措施均显著提高了0~20 cm与20~40 cm土层R>0.25 mm团聚体的含量。0~20 cm土层,Tm处理R>0.25 mm团聚体含量达到最大值,且显著高于其他各处理,依次表现为Tm>Tf>Tn>Ts>CK,分别较Tn、Ts、Tf处理和CK显著增加了9.39%、11.38%、6.09%和35.41%,Tn、Ts、Tf处理较CK显著增加了23.79%、21.57%、27.64%;20~40 cm土层,Tm较Tn处理和CK显著增加了13.74%、38.65%,Tn、Ts、Tf处理较CK显著增加了21.90%、35.34%和38.57%,依次表现为Tm>Tf>Ts>Tn>CK,Tm、Ts、Tf各处理间差异不显著。
2.1.2 耕作措施对土壤团聚体稳定性的影响
除Tn处理外,其余各耕作措施均显著提高了0~20 cm与20~40 cm土层土壤团聚体平均重量直径(MWD)(图1-A)。0~20 cm土层,Tm处理的MWD值达到最大值,分别较Tn处理和CK显著增加了15.34%、30.97%,Ts、Tf处理较CK显著增加了20.65%、18.71%,依次表现为Tm>Ts>Tf>Tn>CK,Tm、Ts和Tf处理间差异不显著,Tn与CK间差异不显著;20~40 cm土层,Ts处理的MWD值达到最大值,显著高于Tn、Tf处理和CK 19.90%、6.64%和22.96%,Tm、Tf处理显著高于Tn和CK 15.92%、12.44%和18.88%、15.31%,依次表现为Ts>Tm>Tf>Tn>CK,Tm和Ts处理间差异不显著,Tn处理和CK间差异不显著,Tm和Tf处理间差异不显著。
各耕作措施均显著提高了0~20 cm与20~40 cm土层土壤团聚体几何平均直径(GMD)(图1-B)。0~20 cm土层,Tm处理的GMD值达到最大值,分别较Tn、Tf处理和CK显著增加了22.55%、15.74%、54.32%,但与Ts处理间无显著差异,Tn、Ts、Tf处理较CK显著增加了25.93%、35.80%、33.33%,依次表现为Tm>Ts>Tf>Tn>CK,Tn、Ts、Tf处理间差异不显著;20~40 cm土层,Ts处理的GMD值达到最大值,显著高于Tn处理和CK 28.81%、53.54%,Tm、Ts、Tf处理显著高于Tn处理和CK 27.12%、22.03%和51.52%、45.45%,Tn处理较CK显著增加了19.19%,依次表现为Ts>Tm>Tf>Tn>CK,Tm、Ts、Tf处理间差异不显著。不同土层深度下,各耕作措施对土壤团聚体的分形维数(D值)影响无差异(图1-C)。
2.2 耕作措施对土壤团聚体有机碳稳定性的影响
2.2.1 土壤团聚体有机碳分布
不同土层深度下各粒级团聚体的有机碳含量均表现为Tn、Tm处理显著高于CK(图2)。0~20 cm土层,除Tf处理外,其余各耕作措施土壤有机碳含量均显著高于CK(p<0.05),Tn处理显著高于Ts、Tf处理和CK 18.58%、39.53%和58.40%,Tm处理显著高于Tf和CK 31.15%和48.89%,Ts处理显著高于CK 33.58%,依次表现为Tn>Tm>Ts>Tf>CK,Tn和Tm处理间差异不显著,Tm和Ts处理间差异不显著;20~40 cm土层,除Ts处理外,其余各耕作措施土壤有机碳含量均显著高于CK,Tn处理显著高于Ts、Tf处理和CK 40.08%、22.84%和60.93%,Tm处理显著高于Ts和CK 31.58%和51.16%,Tf处理显著高于CK 31.01%,依次表现为Tn>Tm>Tf>Ts>CK,Tn和Tm处理间差异不显著,Ts和Tf处理间差异不显著。各耕作措施均可提高农田0~20 cm和20~40 cm土层各粒级团聚体中的有机碳含量,以Tn和Tm处理的土壤团聚体有机碳含量较高。在各土层中,有机碳含量在各粒级团聚体中大小依次为(>2 mm)>(1~2 mm)>(0.25~1 mm)>(<0.25 mm),0~20 cm土层各粒级团聚体有机碳含量高于20~40 cm土层。
2.2.2 土壤团聚体有机碳累积矿化量
各耕作措施均显著提高了农田0~20 cm和20~40 cm土层土壤的有机碳累积矿化量,同时增加了各粒级团聚体中的有机碳累积矿化量(图3)。0~20 cm土层,Ts处理的土壤有机碳累积矿化量达到最大值,较Tf、Tm、Tn处理和CK显著增加了37.50%、77.25%、79.81%和138.22%,Tf处理较Tm、Tn处理和CK显著增加了28.91%、30.77%和73.25%,依次表现为Ts>Tf>Tm>Tn>CK,Tn与Tm处理间差异不显著;20~40 cm土层,Tf处理的土壤有机碳累积矿化量达到最大值,较Ts、Tm、Tn处理和CK显著增加了9.40%、19.26%、29.33%和91.45%,Ts处理较Tm、Tn处理和CK显著增加了9.02%、18.22%和75.00%,依次表现为Tf>Ts>Tm>Tn>CK,Tn与Tm处理间差异不显著;各粒级团聚体中Ts和Tf处理有机碳累积矿化量显著高于其他处理,在各粒级团聚体中大小依次为(<0.25 mm)>(0.25~1 mm)>(1~2 mm)>(>2 mm),分别在>2 mm、1~2 mm、0.25~1 mm、<0.25 级团聚体条件下,Ts处理较CK显著提高了133.33%、152.52%、178.57%、109.72%,Tf处理较CK显著提高了144.12%、82.01%、80.71%、33.60%;20~40 cm土层,各粒级团聚体中有机碳累积矿化量以Ts和Tf处理较大,且显著高于CK(除Ts处理下的<0.25 mm粒级团聚体),分别在>2 mm、1~2 mm、0.25~1 mm、<0.25 mm各粒级团聚体条件下,Tf处理较CK显著提高了122.22%、171.67%、133.33%、23.26%,除<0.25 mm粒级团聚体外,Tn、Tm和Ts处理均显著高于CK。
2.2.3 土壤有机碳贡献率
各耕作措施显著降低了微团聚体(<0.25 mm)的有机碳贡献率(图4)。0~20 cm土层,Tn、Tm、Ts和Tf处理较CK显著降低了微团聚体有机碳贡献率32.89%、49.37%、26.61%、38.88%,Tm处理>2 mm和0.25~1 mm粒级的有机碳贡献率显著高于CK 49.27%和46.03%,<0.25 mm粒级显著低于Ts处理和CK 31.01%、49.37%,1~2 mm粒级的有机碳贡献率无显著差异;20~40 cm土层,各耕作措施0.25~1 mm粒级的有机碳贡献率显著高于CK,Ts处理>2 mm粒级的有机碳贡献率显著高于Tn处理27.57%,1~2 mm各处理间无显著差异。
3. 讨论
耕作可以通过对土壤的扰动影响土壤结构和微生态环境,从而调控土壤的养分利用率,最终对作物产量构成影响,合理的耕作措施是保证作物获得高产的基本保证[25]。前人研究认为,大于 0.25 mm的团聚体(R>0.25 mm)是土壤中最好的团粒结构,被广泛认为是影响土壤团聚体稳定性的主要因素[26];>0.25 mm团聚体含量与土壤团聚体稳定性呈正相关[9]。本试验结果表明,试验区土壤主要以0.25~1 mm粒级团聚体为主,4种耕作措施均显著提高了土壤大团聚体(>0.25 mm)的含量,促进了土壤团聚体的形成,增加了土壤的稳定性,其中Tm处理的土壤大团聚体含量最高(表2),这与范如芹等[8]在东北黑土地上的研究结果一致。这说明一方面试验区处在干旱荒漠区,植被稀少,地表裸露,地上部植物有机碳源输入困难,致使自然撂荒地土壤有机碳含量减少,胶结作用下降,不利于土壤大团聚体形成[27];另一方面传统耕作频繁的翻耕处理致使土壤有机质快速矿化,减少了稳定性胶结剂的产生,不利于大团聚体的形成,而适度的翻耕处理则有利于大团聚体的形成[8]。平均重量直径(MWD)、几何平均直径(GMD)作为土壤团聚体稳定性指标,其值越大,团聚体稳定性越好[28]。高建华等[4]认为免耕可提高水稳性团聚体平均重量直径,且平均重量直径随土层的增加而增加,以免耕结合秸秆覆盖提升水稳性团聚体平均重量直径的效果最好;与高建华等[4]不同的是,本试验中Tm、Ts和Tf处理显著提升了各土层土壤团聚体的MWD和GMD,但Tn处理仅显著提高了各土层团聚体的GMD,而对各土层团聚体的MWD无显著影响。其中可能的原因是5~8年是短期耕作和长期耕作的转折点,民勤绿洲区农田土壤特征对5年的短期耕作措施响应存在滞后效应,由于短期的耕作措施处理,使得土壤的某些特征特性未显现。
表 2 不同耕作措施下农田土壤团聚体分布Table 2. Effects of tillage on distribution of soil aggregates土层/cm
Soil layer处理
Treatment土壤团聚体粒径 Soil aggregate particle size/% R0.25 >2 mm 1~2 mm 0.25~1 mm <0.25 mm 0~20 Tn 21.25±1.67 c 13.20±1.33 a 35.33±2.35 a 30.22±1.55 b 69.78±1.64 bc Tm 26.58±0.75 a 14.55±0.84 a 35.20±1.53 a 23.67±2.42 c 76.33±1.82 a Ts 24.30±1.24 ab 13.05±0.61 a 31.19±1.24 b 31.47±2.06 b 68.53±1.09 c Tf 22.80±2.07 bc 13.88±0.87 a 35.27±1.71 a 28.05±2.16 b 71.95±0.65 b CK 18.60±0.92 d 13.62±0.79 a 24.15±1.96 c 43.63±1.58 a 56.37±0.38 d 20~40 Tn 26.56±1.22 c 15.19±0.98 a 30.73±1.65 b 27.53±3.00 b 72.47±1.41 b Tm 32.66±0.06 b 15.81±1.12 a 33.97±1.53 a 17.57±1.67 c 82.43±1.30 a Ts 34.85±0.77 a 16.36±1.25 a 29.25±2.55 b 19.54±2.77 c 80.46±1.25 a Tf 30.78±0.23 b 15.62±0.55 a 35.98±1.28 a 17.62±1.64 c 82.38±0.56 a CK 28.32±1.94 c 13.25±1.08 b 17.88±0.85 c 40.55±2.14 a 59.45±0.62 c 注:表中小写字母表示不同处理在p<0.05(Duncan新复极差法)水平下差异显著。
Note: Data with different letters indicate significant differences among treatments at p<0.05.土壤团聚体与土壤有机碳的固定和分解关系密切。土壤有机碳作为土壤团聚体的主要胶结剂[29],影响土壤团聚体的数量和大小分布;耕作方式通过改变土壤有机碳含量、分布和微生物活动生境,从而影响土壤有机质的分解转化和团聚体的变化[30];大多数研究发现免少耕、秸秆还田等保护性耕作模式土壤有机碳含量显著高于传统耕作,且主要集中分布在 0~10 cm 土层[31-33]。殷文等[34]对河西武威绿洲农田土壤的研究发现,土壤各级别团聚体中的有机碳随着团聚体直径的减少和土层深度的加深而减少;郭菊花等[35]对红壤性水稻土的研究指出,土壤有机碳主要分配在250~2000 μm的大团聚体上。本试验也发现耕作提高了农田0~20 cm和20~40 cm土层各粒级团聚体中的有机碳含量,且有机碳主要分布在大团聚体上,尤以Tn和Tm处理的土壤团聚体有机碳含量较高,各粒级团聚体有机碳含量随着土层深度加深而减少,这说明有机碳含量高的组分主要依靠有机胶结物质,这不仅与耕作有关,还与大团聚体的物理保护机制相关。长期的耕作和连续多年的作物种植,作物枯枝败叶和根系残留在土壤表层集聚,有机碳富集在土壤表层,有机碳作为最主要的胶结物质,促使微团聚体更好地胶结形成大团聚体;而作为对照的自然撂荒地,由于试验区处在干旱荒漠区,受水资源和气候条件限制,植物群落稀疏,种群单一,植物覆盖度低,不能有效地积累凋落物来增加植物残体的输入,加上地表大面积裸露加速了有机碳的降解,有机碳含量持续下降,其胶结作用明显降低;因此在本试验中不同粒级土壤团聚体有机碳含量表现为大团聚体(R>0.25 mm)高于微团聚体(R<0.25 mm),各土层表现为表层(0~20 cm)土壤高于深层(20~40 cm)土壤。本研究还发现针对土壤扰动影响较大的两种耕作措施Ts和Tf,Tf处理使土壤各粒级团聚体中的有机碳含量高于Ts处理,但差异不显著(p<0.05),这主要是由于Tf处理对土壤的扰动程度大于Ts处理,导致表层秸秆等有机物能够翻到深土层,使得深层土壤有机碳含量有所增加。
土壤有机碳的矿化受土壤颗粒组成、土壤温度、湿度、pH值、施肥措施、土地类型和质地等因素的影响,不同组分有机碳的土壤物理、化学、生物和非生物等性质差异会导致土壤有机碳含量发生改变,甚至不同组分间会出现显著性差异,从而对不同组分土壤有机碳的累积矿化量产生不同程度的影响[36]。本研究结果表明,与自然撂荒地(对照)相比,4种耕作措施均提高了各土层各粒级团聚体的有机碳累积矿化量,其中Ts和Tf处理含量较高,在0~20 cm土层,Ts和Tf处理使土壤团聚体有机碳矿化累积量显著增加,且随着团聚体粒级的减小而增加。一方面说明免耕使土壤耕层受到较少的物理扰动,土壤结构保持良好,减缓了土壤有机质矿化,而深松、翻耕等耕作措施改善了土壤通气能力,促进了有机质分解与矿化[37-38];另一方面Ts和Tf处理的土壤微团聚体含量较高,大团聚体含量较低。而微团聚体中的有机碳不稳定,易矿化分解,不利于有机碳的储存固定,大团聚体的有机碳较稳定,不易分解,利于固定。这与魏亚伟 和刘晶等[36, 39]的研究结果类似,但与Elliott等[40]的研究结果不一致。Elliott等[40]研究发现土壤大团聚体中的有机碳更为年轻化,因而比微团聚体中的有机碳更易矿化;相反微团聚体中的有机碳比大团聚体中的有机碳老化程度更严重,不易被矿化[41]。总之,有机碳累积矿化量在各级别土壤团聚体中的差异化表现,可能与各粒级土壤团聚体中的有机碳含量、微生物以及土壤母质、地上植被和生态环境的差异紧密相关。
4. 结论
综上所述,耕作可促进耕层土壤团聚体的形成,增加土壤团聚体的稳定性。其中,免耕和少耕能够减少机械的破坏作用,提高各土层及表层土壤大团聚体的有机碳含量,免耕更利于有机碳的固定,少耕更利于土壤团聚体的结构稳定性。综合分析,免耕和少耕可作为民勤绿洲区农田最适宜的土壤耕作措施。
-
表 1 编码基因密码子不同位置GC含量
Table 1 GC content in various positions of codon in chloroplast gene
基因 Gene GC1/% GC2/% GC3/% GCall/% ENC 基因 Gene GC1/% GC2/% GC3/% GCall/% ENC rps12 52.42 47.58 25.00 41.67 43.84 petA 53.89 35.51 26.17 38.53 48.43 psbA 49.44 43.22 32.20 41.62 43.05 rps18 37.25 45.1 30.39 37.58 43.24 atpA 54.72 40.35 23.43 39.50 43.05 rpl20 36.67 44.17 29.17 36.67 54.03 atpF 45.95 33.51 28.65 36.04 48.25 clpP 57.56 37.07 28.29 40.98 56.80 atpI 48.39 36.69 24.60 36.56 45.27 psbB 53.83 46.17 31.04 43.68 48.68 rps2 43.22 40.25 27.97 37.15 47.67 petB 47.69 41.2 30.09 39.66 43.21 rpoC2 45.91 37.29 27.30 36.83 48.68 petD 50.61 37.8 24.39 37.60 43.52 rpoC1 50.15 38.09 29.85 39.36 49.05 rpoA 45.00 34.71 26.47 35.39 48.64 rpoB 49.30 38.38 28.10 38.59 48.53 rps11 53.24 51.8 23.74 42.93 43.04 psbD 52.26 42.94 30.23 41.81 42.10 rps8 40.15 37.12 18.94 32.07 40.85 psbC 53.38 45.99 33.97 44.44 45.89 rpl14 53.66 35.77 27.64 39.02 43.44 rps14 43.56 46.53 27.72 39.27 41.37 rpl16 52.21 52.94 25.74 43.63 41.93 psaA 52.06 43.41 32.22 42.57 49.71 rps3 44.29 33.79 22.83 33.64 45.82 ycf3 47.34 39.05 24.85 37.08 53.43 rpl22 41.40 35.67 22.93 33.33 44.99 rps4 48.02 38.12 30.20 38.78 51.44 ycf2 42.53 35.21 37.34 38.36 53.23 ndhJ 47.17 37.74 30.82 38.57 53.38 ndhB 41.88 39.92 32.29 38.03 47.85 ndhK 41.53 42.80 30.08 38.14 50.43 rps7 53.85 46.15 23.72 41.24 47.64 ndhC 49.59 34.71 29.75 38.02 51.42 ccsA 34.37 38.08 29.10 33.85 47.69 atpE 50.37 42.22 28.15 40.25 44.72 ndhE 43.14 33.33 30.39 35.62 54.50 atpB 55.29 41.32 31.74 42.78 51.77 ndhG 40.11 36.16 25.42 33.9 45.14 rbcL 57.69 42.31 28.54 42.85 47.79 ndhA 42.03 37.09 20.05 33.06 42.54 accD 36.44 34.99 25.67 32.37 44.91 ndhH 48.22 35.79 27.66 37.23 47.50 ycf4 43.78 41.08 31.89 38.92 48.68 ycf1 35.72 26.67 25.69 29.36 46.69 cemA 41.74 26.52 33.04 33.77 47.70 平均值 AVG 47.00 39.41 27.99 38.13 47.27 表 2 编码基因密码子参数的相关性
Table 2 Correlations among individual related parameters of genes
项目 Items GC1 GC2 GC3 GCall ENC N GC1 1.000 GC2 0.390** 1.000 GC3 0.047 0.035 1.000 GCall 0.802** 0.758** 0.396** 1.000 ENC 0.022 −0.277 0.455** 0.029 N −0.126 −0.286 0.296* −0.118 0.196 1.000 注:*表示在0.05水平上显著相关;**表示在0.01水平上极显著相关。
Note: * means significant correlation at 0.05 level; ** means extremely significant correlation at 0.01 level.表 3 相对同义密码子使用度分析
Table 3 RSCU analysis on protein coding region
氨基酸Amino acid 密码子 Coden 数量 Number RSCU 氨基酸 Amino acid 密码子 Coden 数量 Number RSCU Phe UUU 649 1.37 Ser UCU 410 1.71 UUC 359 0.63 UCC 231 0.98 Leu UUA 584 2.09 UCA 283 1.25 UUG 379 1.16 UCG 114 0.43 CUU 384 1.31 Pro CCU 287 1.47 CUC 127 0.38 CCC 172 0.89 CUA 259 0.74 CCA 206 1.15 CUG 126 0.32 CCG 75 0.31 AUU 770 1.48 Thr ACU 379 1.69 Ile AUC 312 0.57 ACC 154 0.68 AUA 492 0.95 ACA 289 1.16 Met AUG 432 1.00 ACG 103 0.47 Val GUU 369 1.45 Ala GCU 444 1.82 GUC 119 0.39 GCC 133 0.59 GUA 377 1.44 GCA 309 1.23 GUG 165 0.73 GCG 93 0.36 Tyr UAU 527 1.68 Cys UGU 157 1.51 UAC 130 0.32 UGC 53 0.36 Ter UAA 22 1.40 Ter UGA 11 0.70 UAG 14 0.89 Trp UGG 322 0.87 His CAU 330 1.45 Arg CGU 260 1.60 CAC 97 0.47 CGC 63 0.37 Gln CAA 528 1.59 CGA 246 1.31 CAG 162 0.41 CGG 83 0.43 Asn AAU 695 1.50 AGA 359 1.71 AAC 190 0.45 AGG 116 0.59 Lys AAA 772 1.50 Ser AGU 292 1.31 AAG 249 0.41 AGC 75 0.31 Asp GAU 638 1.62 Gly GGU 405 1.30 GAC 131 0.38 GGC 122 0.41 Glu GAA 822 1.52 GGA 499 1.68 GAG 271 0.48 GGG 206 0.62 注:Phe:苯丙氨酸,Leu:亮氨酸,Ile:异亮氨酸,Met:蛋氨酸, Val:缬氨酸,Tyr:酪氨酸,His:组氨酸,Gln:谷氨酰胺, Asn:天冬酰胺,Lys:赖氨酸,Asp:天冬氨酸,Glu:谷氨酸,Ser:丝氨酸, Pro:脯氨酸,Thr:苏氨酸,Ala:丙氨酸,Cys:半胱氨酸,Trp:色氨酸,Arg:精氨酸,Gly:甘氨酸,Ter:终止密码子。表5同。
Note: Phe: phenylalanine; Leu: leucine; Ile: isoleucine; Met: methionine; Val: valine; Tyr: tyrosine; His: histidine; Gln: glutamine; Asn: asparagine; Lys: lysine; Asp: aspartic acid; Glu: glutamic acid; Ser: serine; Pro: proline; Thr: threonine; Ala: alanine; Cys: cysteine; Trp: tryptophane; Arg: argnine; Gly: glycine; Ter: termination codon. Same for Table 5.表 4 ENC比值频数分布
Table 4 Distribution of ENC ratios
组限
Class limits组中值
Class mid value组数
Frequency number组频
Frequency−0.25~−0.15 −0.20 1 0.02 −0.15~−0.05 −0.75 5 0.11 −0.05~0.05 0.00 22 0.47 0.05~0.15 0.10 19 0.40 合计 total 47 1.00 表 5 最优密码子分析
Table 5 Analysis on preferred codon
氨基酸
Amino acid密码子
Coden高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 氨基酸
Amino acid密码子
Coden高表达基因
High expressed gene低表达基因
Low expressed geneΔRSCU 数量
NumberRSCU 数量
NumberRSCU 数量
NumberRSCU 数量
NumberRSCU Phe UUU* 53 1.61 25 1.46 0.15 Ser UCU*** 28 1.86 11 1.15 0.71 UUC 19 0.39 9 0.54 -0.15 UCC* 12 1.14 9 0.93 0.21 Leu UUA 47 1.70 21 1.89 -0.19 UCA 16 1.07 14 1.55 −0.48 UUG* 17 1.05 9 0.81 0.24 UCG 2 0.28 5 0.57 −0.29 CUU* 26 1.76 18 1.48 0.28 Pro CCU*** 21 1.89 10 0.91 0.98 CUC 6 0.43 5 0.37 0.06 CCC*** 10 1.03 3 0.29 0.74 CUA 24 0.84 10 0.90 −0.06 CCA 11 0.93 9 0.90 0.03 CUG 8 0.22 7 0.56 −0.34 CCG 2 0.15 3 0.29 −0.14 Ile AUU* 47 1.36 28 1.22 0.14 Thr ACU* 23 1.53 13 1.36 0.17 AUC 10 0.30 13 0.61 −0.31 ACC* 11 0.97 8 0.76 0.21 AUA* 39 1.33 26 1.16 0.17 ACA* 18 1.37 9 1.14 0.23 Met AUG 27 1.00 20 1.00 0.00 ACG 3 0.13 6 0.74 −0.61 Val GUU*** 27 2.19 17 1.55 0.64 Ala GCU 37 1.69 27 2.08 −0.39 GUC* 10 0.41 4 0.29 0.12 GCC 6 0.47 8 0.52 −0.05 GUA 20 1.15 14 1.43 −0.28 GCA*** 26 1.50 14 0.93 0.57 GUG 4 0.24 5 0.73 −0.49 GCG 6 0.34 6 0.47 −0.13 Tyr UAU* 31 1.88 30 1.67 0.21 Cys UGU 10 1.73 5 1.73 0.00 UAC 4 0.12 6 0.33 −0.21 UGC 2 0.27 2 0.27 0.00 Ter UAA 3 1.80 3 1.80 0.00 Ter UGA 1 0.60 1 0.60 0.00 UAG 1 0.60 1 0.60 0.00 Trp UGG 28 1.00 13 0.80 0.20 His CAU 15 1.50 15 1.54 −0.04 Arg CGU** 21 1.60 12 1.21 0.39 CAC 2 0.50 5 0.46 0.04 CGC 3 0.26 3 0.36 −0.10 Gln CAA 28 1.61 24 1.75 −0.14 CGA* 21 1.64 13 1.38 0.26 CAG* 7 0.39 4 0.25 0.14 CGG 1 0.08 7 0.79 −0.71 Asn AAU 27 1.50 33 1.82 −0.32 AGA** 22 1.83 15 1.50 0.33 AAC** 10 0.50 4 0.18 0.32 AGG 7 0.60 8 0.76 −0.16 Lys AAA 34 1.61 23 1.61 0.00 Ser AGU** 17 1.37 12 1.21 0.16 AAG 10 0.39 7 0.39 0.00 AGC 2 0.28 5 0.59 −0.16 Asp GAU 18 0.97 31 1.92 −0.95 Gly GGU*** 35 1.31 7 0.68 0.63 GAC* 8 0.24 2 0.08 0.16 GGC 13 0.44 8 0.60 −0.16 Glu GAA*** 44 1.76 32 1.03 0.73 GGA 29 1.73 18 1.92 −0.19 GAG 6 1.03 15 0.97 0.06 GGG 11 0.52 5 0.79 −0.27 注:_表示RSCU 值大于1 的密码子; *表示△RSCU≥0.08; **表示ΔRSCU≥0.3; ***表示ΔRSCU≥0.5。
Note: Underlining indicates RSCU>1; * means △RSCU≥0.08; ** means △RSCU≥0.3; *** means ΔRSCU≥0.5. -
[1] CHEN S C, WOOD J J, ORMEROD P. Liparis L. C. Richard. In: WU Z Y, RAVEN P H, HONG D. (Eds.) Flora of China, vol. 25[M]. Science Press, Beijing & Missouri Botanical Garden Press, St. Louis, 2009: 211–228.
[2] 乔永刚, 贺嘉欣, 王勇飞, 等. 药用植物苦参的叶绿体基因组及其特征分析 [J]. 药学学报, 2019, 54(11):2106−2112. QIAO Y G, HE J X, WANG Y F, et al. Analysis of chloroplast genome and its characteristics of medicinal plant Sophora flavescens [J]. Acta Pharmaceutica Sinica, 2019, 54(11): 2106−2112.(in Chinese)
[3] DANIELL H, KHAN M S, ALLISON L. Milestones in chloroplast genetic engineering: an environmentally friendly era in biotechnology [J]. Trends in Plant Science, 2002, 7(2): 84−91. DOI: 10.1016/S1360-1385(01)02193-8
[4] LIANG W, GUO X, NAGLE D G, et al. Genus Liparis: A review of its traditional uses in China, phytochemistry and pharmacology [J]. Journal of Ethnopharmacology, 2019, 234: 154−171. DOI: 10.1016/j.jep.2019.01.021
[5] 国家中医药管理局编委会. 中华本草[M]. 上海: 上海科学技术出版社, 1999. [6] 方志先, 廖朝林. 湖北恩施药用植物志(下册)[M]. 武汉: 湖北科学技术出版社, 2006. [7] 吴润. 镰翅羊耳蒜中化学成分的研究[D]. 成都: 西南交通大学, 2017. WU R. Study on chemical constituents of Liparis bootanensis[D]. Chengdu: Southwest Jiaotong University, 2017. (in Chinese).
[8] 林爱英. 福建3种野生兰科植物繁殖生物学的初步研究[D]. 福州: 福建师范大学, 2015. LIN A Y. A preliminary study on reproductive biology of three wild orchids in Fujian province[D]. Fuzhou: Fujian Normal University, 2015. (in Chinese).
[9] LIU J F. The complete chloroplast genome sequence of Liparis bootanensis (Orchidaceae) [J]. Mitochondrial DNA Part B, 2020, 5(3): 2058−2059. DOI: 10.1080/23802359.2020.1763866
[10] BULMER M. The selection-mutation-drift theory of synonymous codon usage [J]. Genetics, 1991, 129: 897−907. DOI: 10.1093/genetics/129.3.897
[11] ROMERO H, ZAVALA A, MUSTO H. Codon usage in Chlamydia trachomatis is the result of strand-specific mutational biases and a complex pattern of selective forces [J]. Nucleic Acids Research, 2000, 28(10): 2084−2090. DOI: 10.1093/nar/28.10.2084
[12] HIRAOKA Y, KAWAMATA K, HARAGUCHI T, et al. Codon usage bias is correlated with gene expression levels in the fission yeast Schizosaccharomyces pombe [J]. Genes to Cells, 2009, 14(4): 499−509. DOI: 10.1111/j.1365-2443.2009.01284.x
[13] SHARP P M, EMERY L R, ZENG K. Forces that influence the evolution of Codon bias [J]. Philosophical Transactions of the Royal Society of London Series B, Biological Sciences, 2010, 365(1544): 1203−1212. DOI: 10.1098/rstb.2009.0305
[14] 杨国锋, 苏昆龙, 赵怡然, 等. 蒺藜苜蓿叶绿体密码子偏好性分析 [J]. 草业学报, 2015, 24(12):171−179. DOI: 10.11686/cyxb2015016 YANG G F, SU K L, ZHAO Y R, et al. Analysis of Codon usage in the chloroplast genome of Medicago truncatula [J]. Acta Prataculturae Sinica, 2015, 24(12): 171−179.(in Chinese) DOI: 10.11686/cyxb2015016
[15] 王婧, 王天翼, 王罗云, 等. 沙枣叶绿体全基因组序列及其使用密码子偏性分析 [J]. 西北植物学报, 2019, 39(9):1559−1572. WANG J, WANG T Y, WANG L Y, et al. Assembling and analysis of the whole chloroplast genome sequence of Elaeagnus angustifolia and its Codon usage bias [J]. Acta Botanica Boreali-Occidentalia Sinica, 2019, 39(9): 1559−1572.(in Chinese)
[16] WRIGHT F. The ‘effective number of codons’ used in a gene [J]. Gene, 1990, 87(1): 23−29. DOI: 10.1016/0378-1119(90)90491-9
[17] JIANG Y, DENG F, WANG H L, et al. An extensive analysis on the global Codon usage pattern of baculoviruses [J]. Archives of Virology, 2008, 153(12): 2273−2282. DOI: 10.1007/s00705-008-0260-1
[18] SUEOKA N. Directional mutation pressure and neutral molecular evolution [J]. Proceedings of the National Academy of Sciences of the United States of America, 1988, 85(8): 2653−2657. DOI: 10.1073/pnas.85.8.2653
[19] SUEOKA N. Near homogeneity of PR2-bias fingerprints in the human genome and their implications in phylogenetic analyses [J]. Journal of Molecular Evolution, 2001, 53(4/5): 469−476.
[20] 胡莎莎, 罗洪, 吴琦, 等. 苦荞叶绿体基因组密码子偏爱性分析 [J]. 分子植物育种, 2016, 14(2):309−317. HU S S, LUO H, WU Q, et al. Analysis of Codon bias of chloroplast genome of Tartary buckwheat [J]. Molecular Plant Breeding, 2016, 14(2): 309−317.(in Chinese)
[21] 续晨, 贲爱玲, 蔡晓宁. 蝴蝶兰叶绿体基因组密码子使用的相关分析 [J]. 分子植物育种, 2010, 8(5):945−950. DOI: 10.3969/mpb.008.000945 XU C, BEN A L, CAI X N. Analysis of synonymous Codon usage in chloroplast genome of Phalaenopsis aphrodite subsp. Formosana [J]. Molecular Plant Breeding, 2010, 8(5): 945−950.(in Chinese) DOI: 10.3969/mpb.008.000945
[22] 李冬梅, 吕复兵, 朱根发, 等. 文心兰叶绿体基因组密码子使用的相关分析 [J]. 广东农业科学, 2012, 39(10):61−65. DOI: 10.3969/j.issn.1004-874X.2012.10.019 LI D M, LV F B, ZHU G F, et al. Analysis on Codon usage of chloroplast genome of Oncidium Gower Ramsey [J]. Guangdong Agricultural Sciences, 2012, 39(10): 61−65.(in Chinese) DOI: 10.3969/j.issn.1004-874X.2012.10.019
[23] 刘慧, 王梦醒, 岳文杰, 等. 糜子叶绿体基因组密码子使用偏性的分析 [J]. 植物科学学报, 2017, 35(3):362−371. DOI: 10.11913/PSJ.2095-0837.2017.30362 LIU H, WANG M X, YUE W J, et al. Analysis of Codon usage in the chloroplast genome of Broomcorn millet (Panicum miliaceum L.) [J]. Plant Sclence Journal, 2017, 35(3): 362−371.(in Chinese) DOI: 10.11913/PSJ.2095-0837.2017.30362
[24] INGVARSSON P K. Gene expression and protein length influence Codon usage and rates of sequence evolution in Populus tremula [J]. Molecular Biology and Evolution, 2007, 24(3): 836−844.
[25] 陆奇丰, 骆文华, 黄至欢. 两种梧桐叶绿体基因组密码子使用偏性分析 [J]. 广西植物, 2020, 40(2):173−183. DOI: 10.11931/guihaia.gxzw201811035 LU Q F, LUO W H, HUANG Z H. Codon usage bias of chloroplast genome from two species of Firmiana Marsili [J]. Guihaia, 2020, 40(2): 173−183.(in Chinese) DOI: 10.11931/guihaia.gxzw201811035
-
期刊类型引用(1)
1. 邓子正,黄明镜,张吴平,王国芳. 旱作条件下保护性耕作对土壤结构和容重影响试验研究. 土壤通报. 2023(01): 46-55 .  百度学术
百度学术
其他类型引用(4)




 下载:
下载: