Transcriptome Analysis on Cryotherapy-treated Virus-free Plantlets of Red Bud Taro at Yanshan, Jiangxi Province
-
摘要:目的 探究江西铅山红芽芋对超低温疗法脱毒的转录组响应机制,为江西铅山红芽芋超低温疗法脱毒苗的规模化应用奠定理论依据。方法 以江西铅山红芽芋超低温疗法脱毒苗(VF组)和江西铅山红芽芋超低温疗法带毒苗(V组)试验材料进行转录组比较分析。结果 VF组Clean reads为42 406 188,GC含量为54.09%;V组Clean reads为46 818 060,GC含量为50.36%。VF组和V组表达量FPKM的对数值在0~2,表达量密度在0~0.8。VF组和V组表达的共有基因数为23 820,V组单独表达的基因数为10 298,VF组单独表达的基因数为4 477。VF组和V组表达量的相关系数为0.287,样本间相关性较差。VF组和V组共产生差异表达基因5 282 个,与V组比较,VF组上调基因3 011,下调基因2 271。GO 富集分析显示,差异基因主要注释到过氧化氢分解过程、过氧化氢代谢过程、一元羧酸生物合成过程、多糖分解代谢过程、金属离子输运、细胞外区、细胞壁、外部封装结构、膜的固有成分、质膜固有成分、核酸结合转录因子活性、序列特异性DNA结合转录因子活性、单加氧酶活性、铁离子结合、血红素结合等功能。KEGG 富集分析显示,差异基因主要注释到苯丙酸生物合成、类黄酮生物合成、二苯乙烯类和二芳基庚烷类以及姜辣素的生物合成、MAPK信号通路-植物、淀粉和蔗糖代谢、酪氨酸代谢、植物激素信号转导、戊糖和葡萄糖醛酸的相互转化、过氧化物酶体、α-亚麻酸代谢、苯丙氨酸代谢、氰胺酸代谢、类胡萝卜素生物合成、异喹啉生物碱的生物合成、泛醌和其他萜类醌的生物合成、甘氨酸、丝氨酸和苏氨酸代谢、植物昼夜节律、果糖和甘露糖代谢、半乳糖代谢、氨基糖和核苷酸糖代谢等途径。结论 F-box家族蛋白、脱落酸受体PYR1、乙烯受体2、生长素响应因子1、BZIP转录因子家族蛋白、过氧化物酶、过氧化氢酶1、超氧化物歧化酶[Cu-Zn]、植物抗病反应蛋白、抗病蛋白、抗病蛋白RPS2、抗病蛋白RPS5、抗病蛋白RGA2、抗病反应蛋白、转座子TNT的逆转录病毒相关Pol多蛋白、逆转录病毒相关Pol多聚蛋白系1、类烟草病毒增殖蛋白3、烟草病毒增殖蛋白1、转座子RE1的逆转录病毒相关Pol多蛋白、蔗糖合成酶等基因是江西铅山红芽芋的超低温疗法脱毒的主要响应基因。Abstract:Objective Mechanism of red bud taro in response to cryotherapy was studied by transcriptome analysis for extended application of the virus-free seedlings at Yanshan, Jiangxi.Method Transcriptomes of virus-free (VF) and virus-carrying (V) red bud taro plantlets that had been cryo-therapeutically treated were analyzed and compared.Result In the VF plantlets, the clean reads were 42 406 188 with a GC content of 54.09%; while, in the V plantlets, the clean reads were 46 818 060 with a GC content of 50.36%. For either group, the FPKM was between 0-2, and the expression density between 0-0.8. There were 23 820 genes commonly expressed in both VF and V, while 4 477 independently expressed in VF, and 10 298 in V. There was a low correlation between VF and V with a coefficient of 0.287, and were 5 282 differentially expressed genes (DEG) in them. Compared to V, VF had 3 011 genes were up-regulated and 2 271 down-regulated. The Go enrichment analysis showed that the differential genes were annotated mainly in the processes of hydrogen peroxide catabolism, hydrogen peroxide metabolism, monocarboxylic acid biosynthesis, and polysaccharide catabolism, metal ion transport, extracellular region, cell wall, external encapsulating structure, intrinsic components of membrane and plasma membrane as well as the activities of nucleic acid binding transcription factor, transcription factor, sequence-specific DNA binding, monooxygenase, iron ion binding, heme binding, etc. The KEGG enrichment analysis indicated that the differential genes were annotated largely to the biosynthesis of phenylpropanoid, flavonoid, stilbenoid, diarylheptanoid, gingerol, carotenoid, isoquinoline alkaloid, ubiquinone, and other terpenoid-quinones, the metabolisms of starch, sucrose, tyrosine, alpha-Linolenic acid, phenylalanine, cyanoamino acid, glycine, serine, threonine, fructose, mannose, galactose, amino sugar, and nucleotide sugar, and the pathways of MAPK signaling-plant, plant hormone signal transduction, pentose and glucuronate interconversions, peroxisome, circadian rhythm-plant, and others,Conclusion The genes of F-box family protein, abscisic acid receptor PYR1, ethylene receptor 2, auxin response factor 1, BZIP transcription factor family protein, peroxidase, catalase 1, superoxide dismutase [Cu-Zn], plant disease resistance response protein, disease resistance protein, disease resistance protein RPS2, disease resistance protein RPS5, disease resistance protein RGA2, disease resistance response protein, retrovirus-related Pol polyprotein from transposon TNT, retrovirus-related Pol polyprotein LINE-1, tobamovirus multiplication protein 3-like, tobamovirus multiplication protein 1, retrovirus-related Pol polyprotein from transposon RE1, sucrose synthase, etc. were the predominant genes in the virus-free red bud taro plantlets that responded to the cryotherapy.
-
0. 引言
【研究意义】江西铅山红芽芋(Colocasia esculenta L. Schott Var. Cormosus CV. Hongyayu),属天南星科(Araceae)芋属(Colocasia)多年生宿根性草本植物,多作为一年收获的粮菜兼用作物,属江西省上饶市铅山县特产,2013年4月15日被核准为国家地理标志农产品(登记证书编号:AGI01110)[1]。铅山红芽芋药食兼优。食用,粉而不粘,细致松滑,糯香可口,淀粉颗粒细小易吸收,品质风味居芋类之首[2];药用,含天然多糖,补脾益肠止泻;富微量元素,增强人体免疫;氟含量较高,洁齿防龋护齿;具膳食纤维,润肠通便止秘[3]。江西铅山红芽芋多采用无性繁殖,易积累病毒(如芋花叶病毒、黄瓜花叶病毒等),导致种性退化、早衰严重、抗性不足、产量下降、品质变劣,影响红芽芋种质保存和新品种选育,更影响红芽芋商品性和生产效益,给芋农带来极大损失[4-6]。【前人研究进展】基于超低温保存(Cryopreservation)的超低温疗法(Cryotherapy)是新兴植物脱毒技术[7],可同步实现种质资源“快速脱毒”和“长期保存”双重目标[8-12]。超低温疗法已成为国内外植物脱毒的研究前沿和学术热点。“超低温疗法脱毒”的成功实现主要依赖于“超低温保存技术体系”的建立,理论上只要能利用“超低温”保存种质的植物均可实现“超低温疗法脱毒”,应用前景广阔。目前,国内外的种质资源超低温保存专家开始关注“超低温疗法脱毒”在其他植物(马铃薯、苹果等)上的适用性和广谱性,取得了一系列研究成果[13-14]。病毒侵染对植物来说是一种生物胁迫[15]。从植物转录组数据着手,获得转录组与生物胁迫之间的关系,是研究植物抗逆性的重要手段[16]。转录组测序(RNA-Seq)随着第 2 代测序技术的快速发展已成为植物分子生物学研究的重要手段,一般用于功能基因挖掘、分子标记开发、代谢通路和调控机制研究等方面[17-19]。【本研究切入点】超低温疗法脱毒是感染病毒的一个逆向过程,是解除病毒这种生物胁迫的过程。超低温疗法脱毒后植物在转录组水平上的分子响应机制还有待深入研究。【拟解决的关键问题】本试验以江西铅山红芽芋为研究对象,通过超低温疗法对其进行脱毒,然后采用RNA-Seq技术,对超低温疗法脱毒苗和超低温疗法带毒苗进行转录组测序,筛选差异表达显著的基因进行序列分析、比较和功能聚类,初步了解江西铅山红芽芋“超低温疗法脱毒”的响应机制,为江西铅山红芽芋超低温疗法脱毒苗的规模化应用提供依据。
1. 材料与方法
1.1 试验材料
江西铅山红芽芋超低温疗法脱毒苗(VF组)和江西铅山红芽芋超低温疗法带毒苗(V组)由上饶师范学院生命科学学院上饶市药食同源植物资源保护与利用重点实验室制备。材料获得方法:切取江西铅山红芽芋1 cm左右的茎尖,进行超低温保存,最后对其再生苗进行病毒RT-PCR检测,获得江西铅山红芽芋超低温疗法脱毒苗(VF组)和江西铅山红芽芋超低温疗法带毒苗(V组)。
1.2 试验方法
1.2.1 RNA提取
参照说明书用Trizol试剂提取江西铅山红芽芋超低温疗法脱毒苗(VF组)和江西铅山红芽芋超低温疗法带毒苗(V组)总RNA。所提RNA的质量由Nanodrop 2000和琼脂糖凝胶电泳检测,Agilent 2100 测定RIN值。单次建库若RNA总量5 μg,浓度≥200 ng·μL−1,OD260/280为1.8~2.2即为可用。
1.2.2 Illumina Hiseq测序
RNA测序送由上海美吉生物科技股份有限公司完成。
1.2.3 数据分析
使用Trinity软件对clean reads进行de novo 拼接,形成Unigene序列。利用Trinity软件组装得到转录本与参考蛋白质数据库NR进行BLAST比对。使用软件Trans Decode对组装结果进行CDS预测。再将序列注释到Swiss-Protein、KEGG和GO数据库中获得基因功能信息。根据错误发现率(False discovery rate,FDR≤0.001)和表达量倍数变化(|Fold change|>1.5)的阈值筛选出2个样本间差异表达的基因(Degs)。对显著性差异表达基因进行KEGG Pathway显著性富集分析。
1.2.4 差异基因的qRT-PCR验证
根据转录组试验结果,采用Primer BLAST在线工具对相关差异表达基因TRINITY_DN22900_c0_g1(F-box family protein)、TRINITY_DN22529_c0_g1(Abscisic acid receptor PYR1)、TRINITY_DN2690_c0_g1(Ethylene receptor 2)、TRINITY_DN3279_c0_g1(Auxin response factor 1)、TRINITY_DN7214_c1_g1(BZIP transcription factor family protein)、TRINITY_DN7681_c0_g2(Peroxidase)、TRINITY_DN298_c0_g2(Catalase 1)、TRINITY_DN7108_c0_g1(Superoxide dismutase [Cu-Zn])、TRINITY_DN22649_c0_g1(Plant disease resistance response protein)、TRINITY_DN15482_c0_g1(Disease resistance protein)、TRINITY_DN14303_c0_g2(Disease resistance protein RPS2)、TRINITY_DN17886_c0_g2(Disease resistance protein RPS5)、TRINITY_DN8674_c0_g5(Disease resistance protein RGA2)、TRINITY_DN183_c1_g1(Disease resistance response protein)、TRINITY_DN4074_c1_g1(Retrovirus-related Pol polyprotein from transposon TNT)、TRINITY_DN10839_c0_g1(Retrovirus-related Pol polyprotein LINE-1)、TRINITY_DN7938_c0_g1(Tobamovirus multiplication protein 3-like)、TRINITY_DN2141_c0_g2(Tobamovirus multiplication protein 1)、TRINITY_DN27902_c0_g1(Retrovirus-related Pol polyprotein from transposon RE1)、TRINITY_DN8401_c0_g1(Sucrose synthase)进行引物设计(表1),每个样品以GAPDH为内参,3个技术重复,采用1.2.1的方法提取2种材料的总RNA,按照Prime Script TM RT reagent Kit with gDNA Eraser(Perfect Real Time)试剂盒说明书合成cDNA的第1条链,在荧光定量PCR仪中进行扩增反应,每个反应重复3次。数据采用SPSS 19.0进行显著性分析。
表 1 qRT-PCR引物序列Table 1. Primers designed for qRT-PCR基因
Gene方向
Direction引物序列
Primer sequence(5′-3′)大小
Size/bpTM值
TM value抗病反应蛋白 Disease resistance response protein F TTCGCGACGAAAGGTAAAACGA 105 55 R CACCCACAGGATGGACCAGAGG 抗病蛋白RGA2 Disease resistance protein RGA2 F CGGTTCAGAAGGGAGCCTATTGT 199 53.3 R AAACTCTTATACCTTTGGGGGGC 乙烯受体2 Ethylene receptor 2 F TCCCTTTTGACCTTGTTCTTTTG 137 52 R TACATTCTCCCGTGTACTTGCAG 过氧化氢酶1 Catalase 1 F CAGTGTCAAGACACGGTCGCATG 198 53.8 R CTAGTAACCGCTAGGGCCGTTGG 蔗糖合成酶 Sucrose synthase F GCCCCGTCTTATTCATACCCTCG 173 54.6 R CATGCTTTTCCCATTTTGTCCTCA 抗病蛋白RPS5 Disease resistance protein RPS5 F GTCCCAACTTATCCGTCATTTCT 168 51.4 R TAAAGGAGCTGCCACTCACAGTC 抗病蛋白 Disease resistance protein F TGTGTCTCCCTCCAGTGCTCA 145 53.6 R TTTCTCGCCCCCCCTGTTCA 类烟草病毒增殖蛋白3 Tobamovirus multiplication protein 3-like F GGTGTCTCGTTGTTTGCTGCTC 103 52.1 R GTTTCTTGCGTCGCCCTTTC 植物抗病反应蛋白 Plant disease resistance response protein F TGCCCTGAGCGTTCCCTAC 178 58.3 R ACCCGCCTCCACTTCTTCC F-box家族蛋白 F-box family protein F CGCCAAGAGGGAAATGAAACC 195 58.5 R CGGGAACATGGAGCAGTGGTA 超氧化物歧化酶[Cu-Zn] Superoxide dismutase [Cu-Zn] F CTGAGGCAACGATTGTGGATA 130 53.2 R CCCGTGCTAAGGCTAAGTTCAT 逆转录病毒相关Pol多聚蛋白系-1
Retrovirus-related Pol polyprotein line-1F CCCTTCTTTGTTAATGCCCACCA 156 49.9 R CGTCCTTAATGCACGGAAGCA 转座子RE1的逆转录病毒相关Pol多蛋白
Retrovirus-related Pol polyprotein from transposon RE1F GTCCAGATTGAAGTCGCTCCC 117 53.5 R CCTTGTACTGTGACAACAATGCC 转座子TNT逆转录病毒相关Pol多蛋白
Retrovirus-related Pol polyprotein from transposon TNTF GATAGACCATCAGGCACTTTAGG 151 48.5 R CTAAGTCATTCTTGGGACACTGTAA BZIP转录因子家族蛋白 BZIP transcription factor family protein F CATCGCCGAGCACTTGCACCGTCTC 95 55.6 R GCTTCTGGCATCTTTACCACTTTC 过氧化物酶 Peroxidase F CCAACTCCCAGGACTTCTTCTTCC 136 57.9 R CTGGCTGGCTAGTGGTGCTTGTT 脱落酸受体PYR1 Abscisic acid receptor PYR1 F CTCCATTTCGCCCTGATTCCATT 139 54.6 R TCAAACATCCCTTTCACCATTCCTAT 抗病蛋白RPS2 Disease resistance protein RPS2 F ATCCATTCCTTGCCAGATGAC 115 53.4 R ACCTTATTAACCGCAGCACCA 生长素反应因子1 Auxin response factor 1 F CTTTCACGCCCAATCGACCAC 169 57.6 R TCGGCTTCTTGCTTTCTGCTGTC 烟草病毒增殖蛋白1 Tobamovirus multiplication protein 1 F CTGTGGGAAGACTTCTTGCCTGAT 108 50.9 R TCGCTTTGGTATTATTGGACCTG 甘油醛-3-磷酸脱氢酶 GAPDH (内参 Reference gene) F ATCAAGCCCTCAACAATGCCAAA 179 54.8 R GCCAAGAAGGTCGTCATCTCAGC 2. 结果与分析
2.1 江西铅山红芽芋超低温疗法脱毒苗测序数据产出分析
碱基质量值(Quality Score 或 Q-score)是碱基识别(Base Calling))出错概率映射。碱基质量值越高表明碱基识别越可靠。通常Quality scores大于20 即满足分析对测序质量的要求。经过测序质量控制,江西铅山红芽芋超低温疗法脱毒苗(VF组)和江西铅山红芽芋超低温疗法带毒苗(V组)的文库构建质量和测序质量较好,可用于后续的转录组分析(表2)。
表 2 V组和VF组测序数据统计Table 2. Statistical sequencing data on V and VF plantlets样本 Sample 带毒苗 V 脱毒苗 VF 原始序列
Raw reads47 418 842 43 025 444 原始总碱基数
Raw bases7 160 245 142 6 496 842 044 过滤后序列
Clean reads46 818 060 42 406 188 质控后总碱基数
Clean bases6 964 653 736 6 305 931 529 测序错误率
Error rate/%0.023 1 0.024 6 大于20的碱基数占总碱基的百分比
Q20/%98.83 98.26 大于30的碱基数占总碱基的百分比
Q30/%96.08 94.50 G、C碱基的总数量占总碱基数量的百分比
GC content/%50.36 54.09 2.2 江西铅山红芽芋超低温疗法脱毒苗测序数据与组装结果
将每个样本的 clean reads 与Trinity组装获得的参考序列进行比对,获得每个样本的mapping结果,也是后续进行各样本基因和转录本定量的基础。一般能定位到组装转录本上的 clean reads 所占百分比大于70%即为比对结果较好。由表3可知,测序数据与组装结果比对结果较好,可以进行后续VF组和V组样本基因和转录本定量等工作。
表 3 测序数据与组装结果比对统计Table 3. Comparison on sequencing data and assembly results样本
Sample过滤后测序
数据的条数
Clean reads能比对到组装
转录本上的
Clean reads数
Mapped reads能定位到组装转
录本上的Clean
reads所占百分比
Mapped ratio/%脱毒苗VF 42 406 188 34 683 956 81.79 带毒苗V 46 818 060 38 036 872 81.24 2.3 江西铅山红芽芋超低温疗法脱毒苗表达量分析
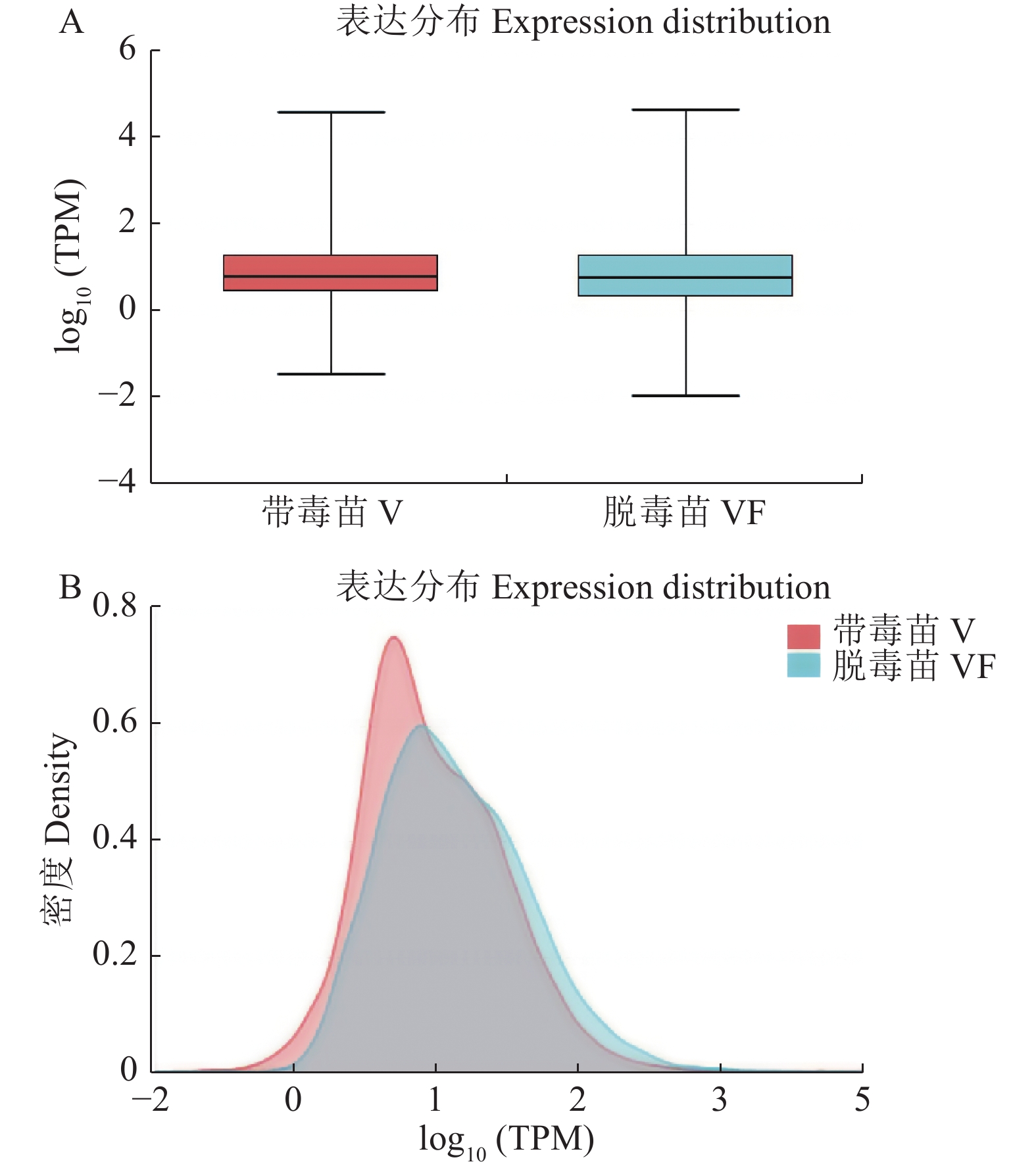
TPM(Transcripts Per Million reads):即每百万读段中来自某转录本的读段数。TPM和FPKM一样,都对基因长度和测序深度进行了均一化。但不同的是,FPKM是先对测序深度进行均一化,然后对基因长度进行均一化;而TPM是先对基因长度进行均一化,然后再对测序深度进行均一化。TPM的均一化过程使得不同样本中的总表达量一致,这样可以更直观地进行表达量的比较。由图1-A,VF组和V组表达量FPKM的对数值在0~2。由图1-B展示的VF组和V组样本unigene/transcript的表达量密度分布情况可知,VF组和V组表达量密度在0~0.8。
2.4 江西铅山红芽芋超低温疗法脱毒苗表达量样本间Venn分析
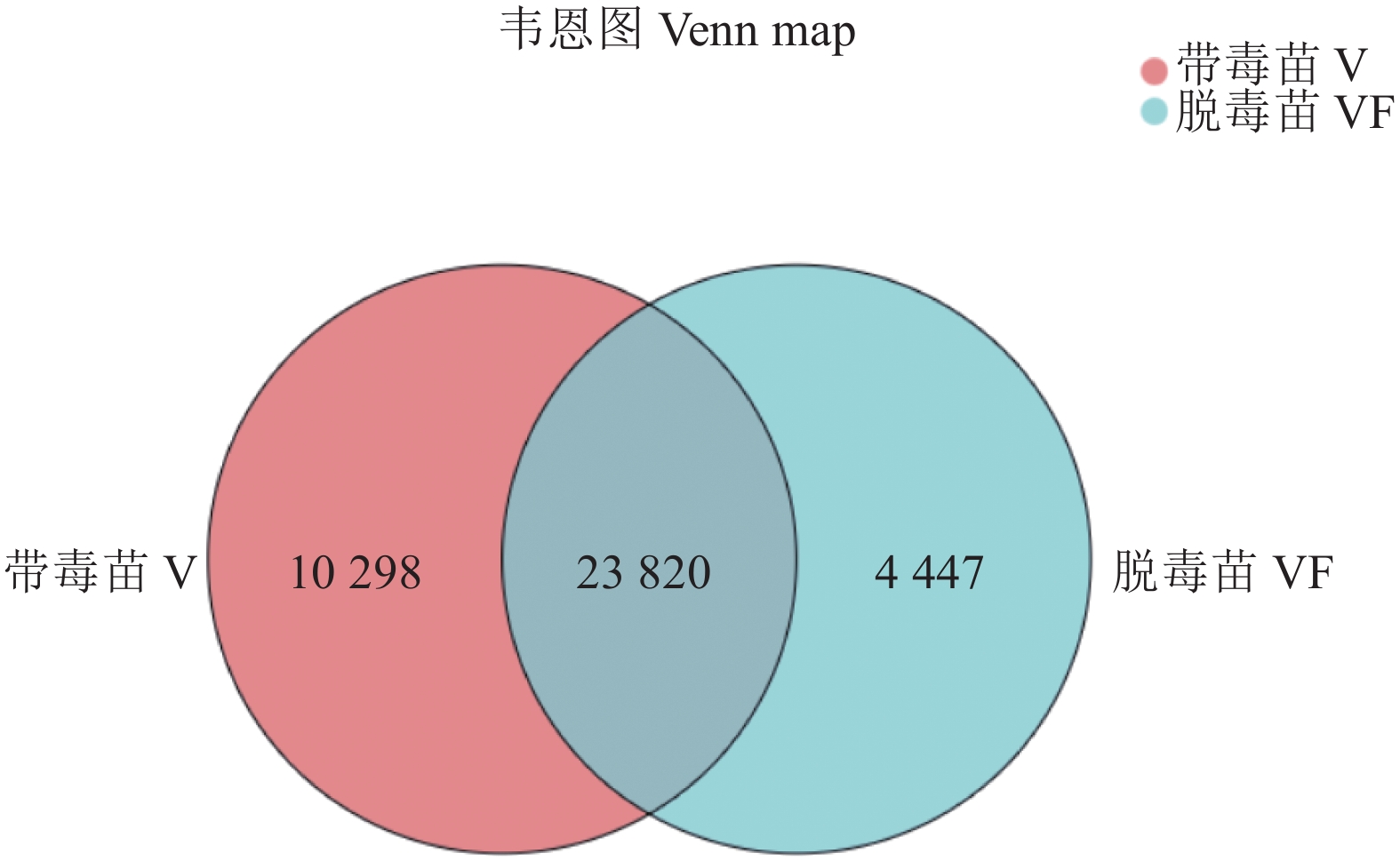
从图2可知,VF组和V组表达的共有基因数为23820,V组单独表达的基因数为10 298,VF组单独表达的基因数为4 477。VF组单独表达的基因数远远少于V组单独表达的基因数。
2.5 江西铅山红芽芋超低温疗法脱毒苗表达量样本间相关性分析
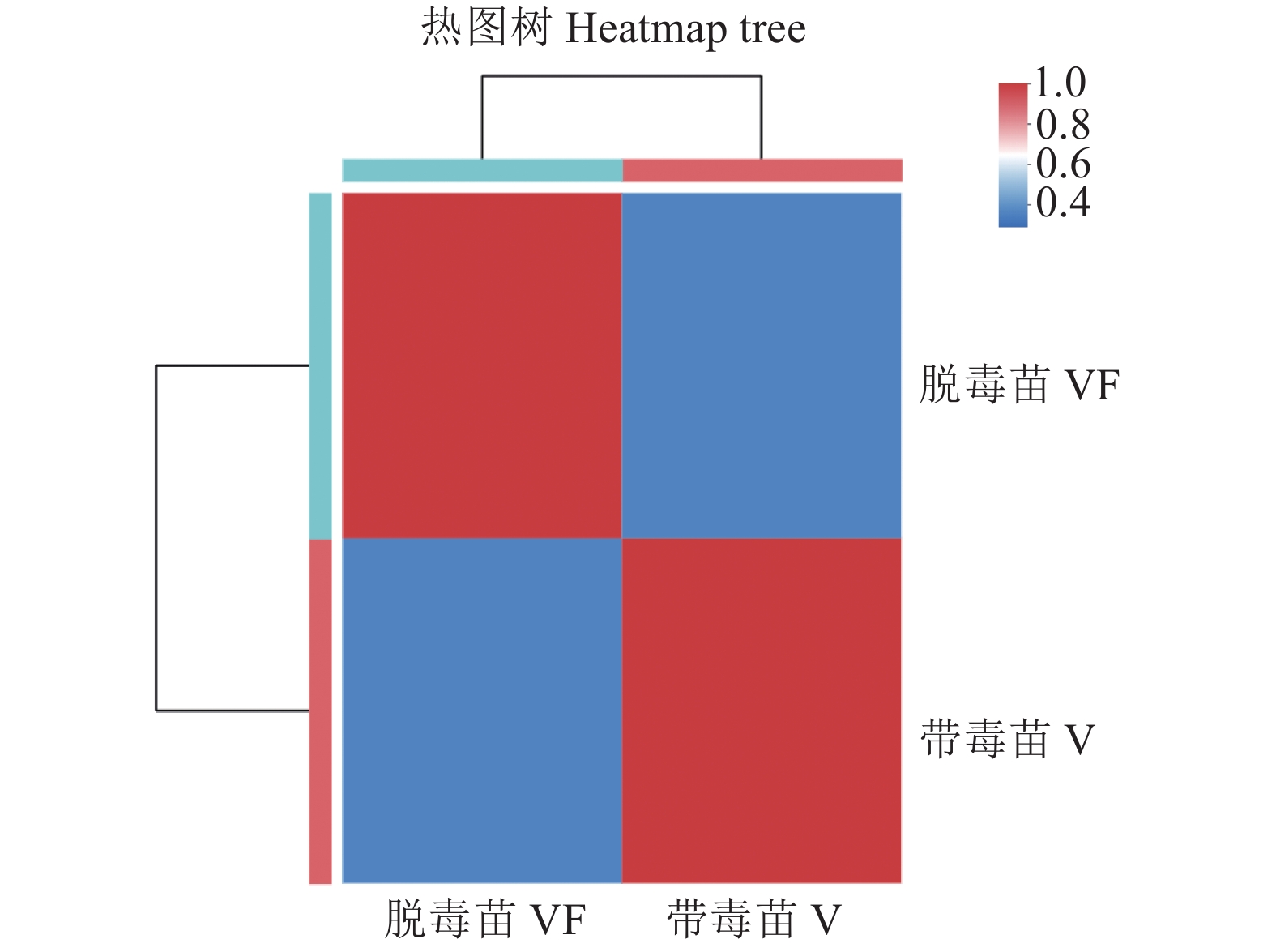
由图3可知,VF组和V组表达量的相关系数为0.287,表明基因在VF组和V组2个样本间的表达量相似度较低,即样本间相关性不大。
2.6 江西铅山红芽芋超低温疗法脱毒苗差异表达分析
VF组和V组共产生差异表达基因(Differentially expressed gene, DEG)5 282 个,与V组比较,VF组上调基因3011,下调基因2271(图4)。从表4可知,VF组和V组前10个差异表达基因大多为假设蛋白和外壳蛋白。
表 4 VF组和V组表达量差异统计(前10)Table 4. Top 10 statistical expression differences between VF and V plantlets基因编号
Gene_ID差异表达倍数
FC(VF/V)差异表达倍数
以2为底的
对数值
log2FC
(VF/V)P值
P value矫正后 P值
P adjust显著性
Significance调节
Regulate带毒苗
表达量
Expression
in V脱毒苗
表达量
Expression
in VFNR描述
NR
descriptionTRINITY_DN56_c0_g1 5.15×10−6 −17.57 5.30×10−41 1.65×10−36 是 Yes 下调
Down33445.52 0.12 假设蛋白质
Hypothetical proteinTRINITY_DN56_c1_g1 5.39×10−6 −17.50 8.29×10−41 1.65×10−36 是 Yes 下调
Down2710.44 0.01 假设蛋白质
MIMGU_mgv1a026582mg,
hypothetical protein
MIMGU_mgv1a026582mgTRINITY_DN388_c0_g1 1.36×10−5 −16.16 4.43×10−40 5.88×10−36 是 Yes 下调
Down37524.60 0.40 假设蛋白质
Hypothetical proteinTRINITY_DN56_c0_g2 2.62×10−5 −15.22 7.54×10−40 7.52×10−36 是Yes 下调
Down24571.29 0.53 假设蛋白质
Hypothetical proteinTRINITY_DN1368_c0_g1 2.82×10−5 −15.11 8.27×10−39 6.59×10−35 是 Yes 下调
Down16978.15 0.39 未注释 No TRINITY_DN863_c0_g2 2.14×10−5 −15.51 4.02×10−38 2.67×10−34 是 Yes 下调
Down2861.27 0.05 假设蛋白质
MIMGU_mgv1a026582mg hypothetical protein MIMGU_mgv1a026582mgTRINITY_DN1333_c0_g1 2.42×10−5 −15.33 1.41×10−37 8.01×10−34 是 Yes 下调
Down8296.82 0.16 未注释 No TRINITY_DN893_c0_g2 2.93×10−6 −18.38 3.72×10−35 1.86×10−31 是 Yes 下调
Down11335.30 0.00 假设蛋白质
Hypothetical proteinTRINITY_DN4426_c0_g1 8.51×10−5 −13.52 7.01×10−35 3.11×10−31 是 Yes 下调
Down31665.60 4.91 假设蛋白质
Hypothetical proteinTRINITY_DN6139_c0_g1 6.48×10−5 −13.91 4.83×10−34 1.93×10−30 是 Yes 下调
Down17471.00 0.87 外壳蛋白
Coat protein2.7 江西铅山红芽芋超低温疗法脱毒苗差异表达基因GO富集分析
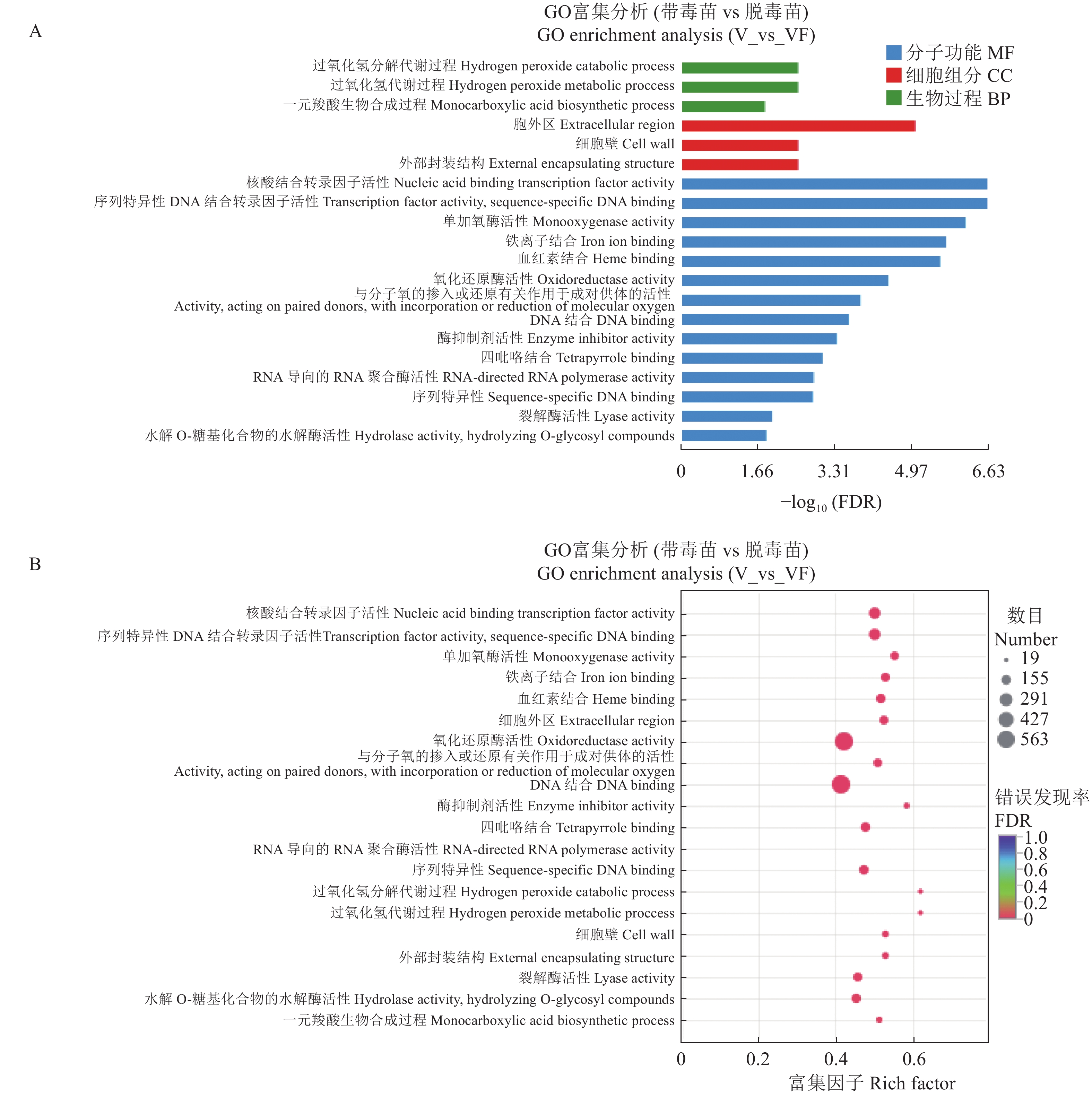
从图5和表5可知,通过富集,过氧化氢分解过程、过氧化氢代谢过程、一元羧酸生物合成过程、多糖分解代谢过程、金属离子输运、细胞外区、细胞壁、外部封装结构、膜的固有成分、质膜固有成分、核酸结合转录因子活性、序列特异性DNA结合转录因子活性、单加氧酶活性、铁离子结合、血红素结合等GO term富集显著。这可能是江西铅山红芽芋响应超低温疗法脱毒的主要功能变化。
表 5 部分差异表达基因GO富集结果Table 5. GO enrichment results of some differentially expressed genes富集到该GO term的基因
转录本数目
Number of genes enriched to the GO termGO Term对应
的编号
ID Corresponding
to Go termGO三大分类
Three categories
of GOGO功能描述
Go function description该GO在目
标基因集中
占有的比例
The proportion of the GO in the target gene set/%该GO在背景基因转录本中占有的比例
The proportion of the GO in the background gene/%未经校正
的P值
P value
uncorrected校正后
的P值
P value
corrected37 GO:0042744 生物过程 BP 过氧化氢分解过程
Hydrogen peroxide catabolic process0.62 0.35 0.00 0.00 37 GO:0042743 生物过程 BP 过氧化氢代谢过程
Hydrogen peroxide metabolic process0.62 0.35 0.00 0.00 65 GO:0072330 生物过程 BP 一元羧酸生物合成工艺
Monocarboxylic acid biosynthetic process1.09 0.75 0.00 0.02 48 GO:0000272 生物过程 BP 多糖分解代谢过程
Polysaccharide catabolic process0.80 0.54 0.00 0.04 54 GO:0030001 生物过程 BP 金属离子输运
Metal ion transport0.91 0.62 0.00 0.04 128 GO:0005576 细胞组分 CC 细胞外区
Extracellular region2.15 1.45 0.00 0.00 73 GO:0005618 细胞组分 CC 细胞壁 Cell wall 1.23 0.82 0.00 0.00 73 GO:0030312 细胞组分 CC 外部封装结构
External encapsulating structure1.23 0.82 0.00 0.00 2136 GO:0031224 细胞组分 CC 膜的固有成分
Intrinsic component of membrane35.88 34.47 0.00 0.15 37 GO:0031226 细胞组分 CC 质膜固有成分
Intrinsic component of plasma membrane0.62 0.43 0.00 0.18 209 GO:0001071 分子功能 MF 核酸结合转录因子活性
Nucleic acid binding transcription factor activity3.51 2.48 0.00 0.00 209 GO:0003700 分子功能 MF 序列特异性DNA结合转录因子活性
Transcription factor activity, sequence-specific DNA binding3.51 0.48 0.00 0.00 118 GO:0004497 分子功能 MF 单加氧酶活性
Monooxygenase activity1.98 1.27 0.00 0.00 137 GO:0005506 分子功能 MF 铁离子结合
Iron ion binding2.30 1.54 0.00 0.00 147 GO:0020037 分子功能 MF 血红素结合
Heme binding2.47 1.69 0.00 0.00 2.8 江西铅山红芽芋超低温疗法脱毒苗差异表达基因KEGG注释
从图6和表6可知,VF组和V组差异表达基因富集的KEGG通路主要有苯丙酸生物合成、类黄酮生物合成、二苯乙烯类和二芳基庚烷类以及姜辣素的生物合成、MAPK信号通路-植物、淀粉和蔗糖代谢、酪氨酸代谢、植物激素信号转导、戊糖和葡萄糖醛酸的相互转化、过氧化物酶体、α-亚麻酸代谢、苯丙氨酸代谢、氰胺酸代谢、类胡萝卜素生物合成、异喹啉生物碱的生物合成、泛醌和其他萜类醌的生物合成、甘氨酸、丝氨酸和苏氨酸代谢、植物昼夜节律、果糖和甘露糖代谢、半乳糖代谢、氨基糖和核苷酸糖代谢等。其中植物激素信号转导(基因数92)、苯丙酸生物合成(基因数78)、MAPK信号通路-植物(基因数71)、淀粉和蔗糖代谢(基因数63)、氨基糖和核苷酸糖代谢(基因数50)、过氧化物酶体(基因数39)以及甘氨酸、丝氨酸和苏氨酸代谢(基因数32)注释的基因数均超过30,这可能是江西铅山红芽芋响应超低温疗法脱毒的主要机制。
表 6 差异表达基因KEGG富集部分分析Table 6. Partial results of analysis on KEGG enrichment of differentially expressed genes基因数目
Gene number通路编号
Pathway ID描述
Description该KEGG在目标基因
集中占有的比例
The proportionof KEGG in
target gene concentration/%该KEGG在背景中
占有的比例
The proportion of the
KEGG in the background/%未经校正的P值
P value uncorrected校正后的P值
P value corrected78 map00940 苯丙酸生物合成
Phenylpropanoid biosynthesis2.71 1.36 0.00 0.00 26 map00941 类黄酮生物合成
Flavonoid biosynthesis0.90 0.38 0.00 0.00 17 map00945 二苯乙烯类、二芳基庚烷类和姜辣素的生物合成
Stilbenoid, diarylheptanoid and gingerol biosynthesis0.59 0.26 0.00 0.00 71 map04016 MAPK信号通路-植物
MAPK signaling pathway-plant2.47 1.67 0.00 0.00 63 map00500 淀粉和蔗糖代谢
Starch and sucrose metabolism2.19 1.52 0.00 0.01 26 map00350 酪氨酸代谢
Tyrosine metabolism0.90 0.52 0.00 0.01 92 map04075 植物激素信号转导
Plant hormone signal transduction3.20 2.42 0.00 0.01 26 map00040 戊糖和葡萄糖醛酸的相互转化
Pentose and glucuronate interconversions0.90 0.53 0.00 0.01 39 map04146 过氧化物酶体
Peroxisome1.36 0.88 0.00 0.01 28 map00592 α-亚麻酸代谢
alpha-Linolenic acid metabolism0.97 0.60 0.00 0.02 20 map00360 苯丙氨酸代谢
Phenylalanine metabolism0.69 0.40 0.00 0.03 24 map00460 氰胺酸代谢
Cyanoamino acid metabolism0.83 0.50 0.00 0.03 16 map00906 类胡萝卜素生物合成
Carotenoid biosynthesis0.56 0.31 0.00 0.05 16 map00950 异喹啉生物碱的生物合成
Isoquinoline alkaloid biosynthesis0.56 0.31 0.00 0.04 23 map00130 泛醌和其他萜类醌的生物合
Ubiquinone and other terpenoid-quinone biosynthesis0.80 0.50 0.00 0.05 32 map00260 甘氨酸、丝氨酸和苏氨酸代谢
Glycine, serine and threonine metabolism1.11 0.78 0.00 0.06 23 map04712 植物昼夜节律
Circadian rhythm-plant0.80 0.52 0.00 0.06 28 map00051 果糖和甘露糖代谢
Fructose and mannose metabolism0.97 0.65 0.00 0.07 22 map00052 半乳糖代谢
Galactose metabolism0.76 0.50 0.01 0.10 50 map00520 氨基糖和核苷酸糖代谢
Amino sugar and nucleotide sugar metabolism1.74 1.34 0.01 0.10 2.9 江西铅山红芽芋超低温疗法脱毒苗相关差异表达基因的qRT-PCR分析验证
从RNA-seq数据中挑选20个在KEGG通路注释数量较多的差异表达基因TRINITY_DN22900_c0_g1(F-box family protein)、TRINITY_DN22529_c0_g1(abscisic acid receptor PYR1)、TRINITY_DN2690_c0_g1(ethylene receptor 2)、TRINITY_DN3279_c0_g1(auxin response factor 1)、TRINITY_DN7214_c1_g1(BZIP transcription factor family protein)、TRINITY_DN7681_c0_g2(Peroxidase)、TRINITY_DN298_c0_g2(catalase 1)、TRINITY_DN7108_c0_g1(superoxide dismutase [Cu-Zn])、TRINITY_DN22649_c0_g1(Plant disease resistance response protein)、TRINITY_DN15482_c0_g1(disease resistance protein)、TRINITY_DN14303_c0_g2(Disease resistance protein RPS2)、TRINITY_DN17886_c0_g2(Disease resistance protein RPS5)、TRINITY_DN8674_c0_g5(disease resistance protein RGA2)、TRINITY_DN183_c1_g1(Disease resistance response protein)、TRINITY_DN4074_c1_g1(Retrovirus-related Pol polyprotein from transposon TNT)、TRINITY_DN10839_c0_g1(Retrovirus-related Pol polyprotein LINE-1)、TRINITY_DN7938_c0_g1(tobamovirus multiplication protein 3-like)、TRINITY_DN2141_c0_g2(tobamovirus multiplication protein 1)、TRINITY_DN27902_c0_g1(Retrovirus-related Pol polyprotein from transposon RE1)、TRINITY_DN8401_c0_g1(sucrose synthase),并利用qRT-PCR技术对其表达量进行验证(表7),结果显示,20个基因表达量的变化趋势均与RNA-seq结果一致,说明本次转录组测序数据可信度高。qRT-PCR也进一步验证了F-box家族蛋白、脱落酸受体PYR1、乙烯受体2、生长素响应因子1、BZIP转录因子家族蛋白、过氧化物酶、过氧化氢酶1、超氧化物歧化酶[Cu-Zn]、植物抗病反应蛋白、抗病蛋白、抗病蛋白RPS2、抗病蛋白RPS5、抗病蛋白RGA2、抗病反应蛋白、转座子TNT的逆转录病毒相关Pol多蛋白、逆转录病毒相关Pol多聚蛋白系1、类烟草病毒增殖蛋白3、烟草病毒增殖蛋白1、转座子RE1的逆转录病毒相关Pol多蛋白、蔗糖合成酶等基因是江西铅山红芽芋的超低温疗法脱毒的主要响应基因。
表 7 20个差异表达基因qRT-PCR的RQ值Table 7. RQ values of qRT-PCR for 20 differentially expressed genes基因
Gene正常种
RQ值
RQ value in CK平原种植
退化种 RQ值
RQ value in BZ调节
RegulationRNA-seq调节
Regulation in
RNA-seq抗病反应蛋白 Disease resistance response protein 1.00 b 216.68 a 上调 Up 上调 Up 抗病蛋白 RGA2 Disease resistance protein RGA2 1.00 b 6.29 a 上调 Up 上调 Up 乙烯受体2 Ethylene receptor 2 1.00 a 0.48 b 下调 Down 下调 Down 过氧化氢酶1 Catalase 1 1.00 b 8.23 a 上调 Up 上调 Up 蔗糖合成酶 Sucrose synthase 1.00 b 10.50 a 上调 Up 上调 Up 抗病蛋白 RPS5 Disease resistance protein RPS5 1.00 b 10.02 a 上调 Up 上调 Up 抗病蛋白 Disease resistance protein 1.00 b 10.04 a 上调 Up 上调 Up 类烟草病毒增殖蛋白3 Tobamovirus multiplication protein 3-like 1.00 a 0.78 b 下调 Down 下调 Down 植物抗病反应蛋白 Plant disease resistance response protein 1.00 b 10.01 a 上调 Up 上调 Up F-box家族蛋白 F-box family protein 1.00 b 7.815577 a 上调 Up 上调 Up 超氧化物歧化酶 [Cu-Zn] Superoxide dismutase [Cu-Zn] 1.00 b 9.217215 a 上调 Up 上调 Up 逆转录病毒相关 Pol多聚蛋白系-1 Retrovirus-related Pol polyprotein LINE-1 1.00 a 0.13 b 下调 Down 下调 Down 转座子 RE1的逆转录病毒相关 Pol多蛋白 Retrovirus-related Pol polyprotein from transposon RE1 1.00 a 0.01 b 下调 Down 下调 Down 转座子 TNT逆转录病毒相关 Pol多蛋白 Retrovirus-related Pol polyprotein from transposon TNT 1.00 a 0.06 b 下调 Down 下调 Down BZIP转录因子家族蛋白 BZIP transcription factor family protein 1.00 a 0.46 b 下调 Down 下调 Down 过氧化物酶 Peroxidase 1.00 b 16.00 a 上调 Up 上调 Up 脱落酸受体 PYR1 Abscisic acid receptor PYR1 1.00 a 0.25 b 下调 Down 下调 Down 抗病蛋白 RPS2 Disease resistance protein RPS2 1.00 b 5.40 a 上调 Up 上调 Up 生长素反应因子1 Auxin response factor 1 1.00 a 0.50 b 下调 Down 下调 Down 烟草病毒增殖蛋白1 Tobamovirus multiplication protein 1 1.00 a 0.49 b 下调 Down 下调 Down 甘油醛-3-磷酸脱氢酶 GAPDH 1.00 a 1.00 a - - 注:同行数据后不同小写字母表示显著性差异(P<0.05)。
Note: Different lowercase letters in each line indicate significant difference at 0.05 level.3. 讨论与结论
与常规茎尖培养脱毒法相比,茎尖超低温疗法脱毒率显著提高,且脱毒率不受茎尖大小影响,操作简便,试验周期短、成本低,可同时实现种质保存和病毒脱除双重目的[20-22]。目前,超低疗法脱毒苗的转录组分析一般专注于超低温保存程序本身。百子莲胚性愈伤组织超低温保存前脱水和恢复培养材料的转录组分析表明,差异表达基因主要参与到外源刺激响应(517个)、胁迫响应(302个)、植物激素信号转导(207个)等,逆境响应主要表现在细胞膜质结构、还原性物质和植物激素的变化、细胞结构修复及发育相关物质合成代谢等方面[23]。冷冻胁迫下MLP2-1 拟南芥与野生型拟南芥植株差异表达基因的KEGG代谢通路富集在植物激素与信号转导代谢通路[24]。本试验结果专注于超低温保存后脱毒苗的内在分子响应机制,与上述结果的出发点不同。在本试验中,江西铅山红芽芋超低温疗法脱毒苗(VF组)和江西铅山红芽芋超低温疗法带毒苗(V组)差异表达基因富集的KEGG通路主要有植物激素信号转导(基因数92)、苯丙酸生物合成(基因数78)、MAPK信号通路-植物(基因数71)、淀粉和蔗糖代谢(基因数63)、氨基糖和核苷酸糖代谢(基因数50)、过氧化物酶体(基因数39)以及甘氨酸、丝氨酸和苏氨酸代谢(基因数32)等通路,这可能是江西铅山红芽芋响应超低温疗法脱毒的主要机制。
植物激素对于江西铅山红芽芋超低温疗法脱毒后的生长发育起着重要作用。植物激素的作用在于形成植物激素信号传导系统,响应内外因素诱导植物激素基因表达[25],激素受体或组件因互作或串话产生协同或拮抗作用从而使植物激素信号转导途径形成网络化[26],最终综合产生效应,使江西铅山红芽芋经过超低温疗法脱毒恢复种性。苯丙氨酸类代谢能合成次生酚类物质和抗菌作用产物(如木质素、绿原酸和植保素等)[27],阻止病原菌生长繁殖,苯丙氨酸解氨酶(PAL)是苯丙氨酸类代谢的定速酶[28]。江西铅山红芽芋超低温疗法脱毒后,会通过提高PAL的活性,促使木质素和绿原酸的积累和植保素的合成,以增强江西铅山红芽芋超低温疗法脱毒苗的抗病性。MAPK(丝裂原活化蛋白激酶)信号通路主要参与各种植物激素信号转导和各种内外因素的应激反应,调控生物非生物胁迫、激素、细胞分裂及植物生长发育[29]。研究表明。MAPK信号通路与植物激素信号传导系统可能存在交叉调控[30-31]。江西铅山红芽芋超低温疗法脱毒苗可增强感知和传递刺激信号的能力,通过MAPK级联通路将信号传递给细胞,启动信号传导系统,对信号级联放大,最终导致植株内部的生理生化变化。江西铅山红芽芋球茎的生长发育(包括形态建成和贮藏物质的积累)是通过光合作用蔗糖的不断合成来完成的[32]。蔗糖和淀粉是变态器官(如球茎)糖类的主要积累形式,可调节和平衡碳水化合物的形态[33]。超低温疗法脱毒可能促使江西铅山红芽芋“淀粉-蔗糖”代谢酶(蔗糖合成酶、蔗糖磷酸合成酶、酸性转化酶、中性转化酶、Q酶)的活性升高,进而对江西铅山红芽芋球茎的发育及其品质起到调控作用。氨基糖最常见的形式是葡糖胺和半乳糖胺,氨基糖代谢与植物生长发育及与环境适应性密切相关。核苷酸糖是糖类合成或相互转换时的活化形式。超低温疗法脱毒可能促使江西铅山红芽芋氨基糖和核苷酸糖各类代谢酶的活性升高,对江西铅山红芽芋球茎的形态建成及质量起到调控作用。过氧化物酶体是过氧化氢产生的细胞器[34],是活性氧类(ROS)的重要来源[35]。研究表明,ROS作为植物激素信号的第二信使,调节植物发育过程和应答反应[36]。过氧化物酶体可能在江西铅山红芽芋超低温疗法脱毒苗中调控ROS,感知环境变化,平衡氧化还原反应,调控细胞各类代谢途径,参与体内不同的重要生理反应。氨基酸是植物新陈代谢关键的参与者[37]。甘氨酸、丝氨酸和苏氨酸代谢可提供一碳单位,合成其他氨基酸、核苷酸、脂质以及合成还原性物质,调控核酸、蛋白质和脂质的合成[38],使江西铅山红芽芋超低温疗法脱毒苗细胞内氧化还原处于动态平衡状态,对维持江西铅山红芽芋超低温疗法脱毒苗的生物代谢及其表观遗传稳定十分重要。本研究表明,F-box家族蛋白、脱落酸受体PYR1、乙烯受体2、生长素响应因子1、BZIP转录因子家族蛋白、过氧化物酶、过氧化氢酶1、超氧化物歧化酶[Cu-Zn]、植物抗病反应蛋白、抗病蛋白、抗病蛋白RPS2、抗病蛋白RPS5、抗病蛋白RGA2、抗病反应蛋白、转座子TNT的逆转录病毒相关Pol多蛋白、逆转录病毒相关Pol多聚蛋白系1、类烟草病毒增殖蛋白3、烟草病毒增殖蛋白1、转座子RE1的逆转录病毒相关Pol多蛋白、蔗糖合成酶等基因是江西铅山红芽芋的超低温疗法脱毒的主要响应基因。
-
表 1 qRT-PCR引物序列
Table 1 Primers designed for qRT-PCR
基因
Gene方向
Direction引物序列
Primer sequence(5′-3′)大小
Size/bpTM值
TM value抗病反应蛋白 Disease resistance response protein F TTCGCGACGAAAGGTAAAACGA 105 55 R CACCCACAGGATGGACCAGAGG 抗病蛋白RGA2 Disease resistance protein RGA2 F CGGTTCAGAAGGGAGCCTATTGT 199 53.3 R AAACTCTTATACCTTTGGGGGGC 乙烯受体2 Ethylene receptor 2 F TCCCTTTTGACCTTGTTCTTTTG 137 52 R TACATTCTCCCGTGTACTTGCAG 过氧化氢酶1 Catalase 1 F CAGTGTCAAGACACGGTCGCATG 198 53.8 R CTAGTAACCGCTAGGGCCGTTGG 蔗糖合成酶 Sucrose synthase F GCCCCGTCTTATTCATACCCTCG 173 54.6 R CATGCTTTTCCCATTTTGTCCTCA 抗病蛋白RPS5 Disease resistance protein RPS5 F GTCCCAACTTATCCGTCATTTCT 168 51.4 R TAAAGGAGCTGCCACTCACAGTC 抗病蛋白 Disease resistance protein F TGTGTCTCCCTCCAGTGCTCA 145 53.6 R TTTCTCGCCCCCCCTGTTCA 类烟草病毒增殖蛋白3 Tobamovirus multiplication protein 3-like F GGTGTCTCGTTGTTTGCTGCTC 103 52.1 R GTTTCTTGCGTCGCCCTTTC 植物抗病反应蛋白 Plant disease resistance response protein F TGCCCTGAGCGTTCCCTAC 178 58.3 R ACCCGCCTCCACTTCTTCC F-box家族蛋白 F-box family protein F CGCCAAGAGGGAAATGAAACC 195 58.5 R CGGGAACATGGAGCAGTGGTA 超氧化物歧化酶[Cu-Zn] Superoxide dismutase [Cu-Zn] F CTGAGGCAACGATTGTGGATA 130 53.2 R CCCGTGCTAAGGCTAAGTTCAT 逆转录病毒相关Pol多聚蛋白系-1
Retrovirus-related Pol polyprotein line-1F CCCTTCTTTGTTAATGCCCACCA 156 49.9 R CGTCCTTAATGCACGGAAGCA 转座子RE1的逆转录病毒相关Pol多蛋白
Retrovirus-related Pol polyprotein from transposon RE1F GTCCAGATTGAAGTCGCTCCC 117 53.5 R CCTTGTACTGTGACAACAATGCC 转座子TNT逆转录病毒相关Pol多蛋白
Retrovirus-related Pol polyprotein from transposon TNTF GATAGACCATCAGGCACTTTAGG 151 48.5 R CTAAGTCATTCTTGGGACACTGTAA BZIP转录因子家族蛋白 BZIP transcription factor family protein F CATCGCCGAGCACTTGCACCGTCTC 95 55.6 R GCTTCTGGCATCTTTACCACTTTC 过氧化物酶 Peroxidase F CCAACTCCCAGGACTTCTTCTTCC 136 57.9 R CTGGCTGGCTAGTGGTGCTTGTT 脱落酸受体PYR1 Abscisic acid receptor PYR1 F CTCCATTTCGCCCTGATTCCATT 139 54.6 R TCAAACATCCCTTTCACCATTCCTAT 抗病蛋白RPS2 Disease resistance protein RPS2 F ATCCATTCCTTGCCAGATGAC 115 53.4 R ACCTTATTAACCGCAGCACCA 生长素反应因子1 Auxin response factor 1 F CTTTCACGCCCAATCGACCAC 169 57.6 R TCGGCTTCTTGCTTTCTGCTGTC 烟草病毒增殖蛋白1 Tobamovirus multiplication protein 1 F CTGTGGGAAGACTTCTTGCCTGAT 108 50.9 R TCGCTTTGGTATTATTGGACCTG 甘油醛-3-磷酸脱氢酶 GAPDH (内参 Reference gene) F ATCAAGCCCTCAACAATGCCAAA 179 54.8 R GCCAAGAAGGTCGTCATCTCAGC 表 2 V组和VF组测序数据统计
Table 2 Statistical sequencing data on V and VF plantlets
样本 Sample 带毒苗 V 脱毒苗 VF 原始序列
Raw reads47 418 842 43 025 444 原始总碱基数
Raw bases7 160 245 142 6 496 842 044 过滤后序列
Clean reads46 818 060 42 406 188 质控后总碱基数
Clean bases6 964 653 736 6 305 931 529 测序错误率
Error rate/%0.023 1 0.024 6 大于20的碱基数占总碱基的百分比
Q20/%98.83 98.26 大于30的碱基数占总碱基的百分比
Q30/%96.08 94.50 G、C碱基的总数量占总碱基数量的百分比
GC content/%50.36 54.09 表 3 测序数据与组装结果比对统计
Table 3 Comparison on sequencing data and assembly results
样本
Sample过滤后测序
数据的条数
Clean reads能比对到组装
转录本上的
Clean reads数
Mapped reads能定位到组装转
录本上的Clean
reads所占百分比
Mapped ratio/%脱毒苗VF 42 406 188 34 683 956 81.79 带毒苗V 46 818 060 38 036 872 81.24 表 4 VF组和V组表达量差异统计(前10)
Table 4 Top 10 statistical expression differences between VF and V plantlets
基因编号
Gene_ID差异表达倍数
FC(VF/V)差异表达倍数
以2为底的
对数值
log2FC
(VF/V)P值
P value矫正后 P值
P adjust显著性
Significance调节
Regulate带毒苗
表达量
Expression
in V脱毒苗
表达量
Expression
in VFNR描述
NR
descriptionTRINITY_DN56_c0_g1 5.15×10−6 −17.57 5.30×10−41 1.65×10−36 是 Yes 下调
Down33445.52 0.12 假设蛋白质
Hypothetical proteinTRINITY_DN56_c1_g1 5.39×10−6 −17.50 8.29×10−41 1.65×10−36 是 Yes 下调
Down2710.44 0.01 假设蛋白质
MIMGU_mgv1a026582mg,
hypothetical protein
MIMGU_mgv1a026582mgTRINITY_DN388_c0_g1 1.36×10−5 −16.16 4.43×10−40 5.88×10−36 是 Yes 下调
Down37524.60 0.40 假设蛋白质
Hypothetical proteinTRINITY_DN56_c0_g2 2.62×10−5 −15.22 7.54×10−40 7.52×10−36 是Yes 下调
Down24571.29 0.53 假设蛋白质
Hypothetical proteinTRINITY_DN1368_c0_g1 2.82×10−5 −15.11 8.27×10−39 6.59×10−35 是 Yes 下调
Down16978.15 0.39 未注释 No TRINITY_DN863_c0_g2 2.14×10−5 −15.51 4.02×10−38 2.67×10−34 是 Yes 下调
Down2861.27 0.05 假设蛋白质
MIMGU_mgv1a026582mg hypothetical protein MIMGU_mgv1a026582mgTRINITY_DN1333_c0_g1 2.42×10−5 −15.33 1.41×10−37 8.01×10−34 是 Yes 下调
Down8296.82 0.16 未注释 No TRINITY_DN893_c0_g2 2.93×10−6 −18.38 3.72×10−35 1.86×10−31 是 Yes 下调
Down11335.30 0.00 假设蛋白质
Hypothetical proteinTRINITY_DN4426_c0_g1 8.51×10−5 −13.52 7.01×10−35 3.11×10−31 是 Yes 下调
Down31665.60 4.91 假设蛋白质
Hypothetical proteinTRINITY_DN6139_c0_g1 6.48×10−5 −13.91 4.83×10−34 1.93×10−30 是 Yes 下调
Down17471.00 0.87 外壳蛋白
Coat protein表 5 部分差异表达基因GO富集结果
Table 5 GO enrichment results of some differentially expressed genes
富集到该GO term的基因
转录本数目
Number of genes enriched to the GO termGO Term对应
的编号
ID Corresponding
to Go termGO三大分类
Three categories
of GOGO功能描述
Go function description该GO在目
标基因集中
占有的比例
The proportion of the GO in the target gene set/%该GO在背景基因转录本中占有的比例
The proportion of the GO in the background gene/%未经校正
的P值
P value
uncorrected校正后
的P值
P value
corrected37 GO:0042744 生物过程 BP 过氧化氢分解过程
Hydrogen peroxide catabolic process0.62 0.35 0.00 0.00 37 GO:0042743 生物过程 BP 过氧化氢代谢过程
Hydrogen peroxide metabolic process0.62 0.35 0.00 0.00 65 GO:0072330 生物过程 BP 一元羧酸生物合成工艺
Monocarboxylic acid biosynthetic process1.09 0.75 0.00 0.02 48 GO:0000272 生物过程 BP 多糖分解代谢过程
Polysaccharide catabolic process0.80 0.54 0.00 0.04 54 GO:0030001 生物过程 BP 金属离子输运
Metal ion transport0.91 0.62 0.00 0.04 128 GO:0005576 细胞组分 CC 细胞外区
Extracellular region2.15 1.45 0.00 0.00 73 GO:0005618 细胞组分 CC 细胞壁 Cell wall 1.23 0.82 0.00 0.00 73 GO:0030312 细胞组分 CC 外部封装结构
External encapsulating structure1.23 0.82 0.00 0.00 2136 GO:0031224 细胞组分 CC 膜的固有成分
Intrinsic component of membrane35.88 34.47 0.00 0.15 37 GO:0031226 细胞组分 CC 质膜固有成分
Intrinsic component of plasma membrane0.62 0.43 0.00 0.18 209 GO:0001071 分子功能 MF 核酸结合转录因子活性
Nucleic acid binding transcription factor activity3.51 2.48 0.00 0.00 209 GO:0003700 分子功能 MF 序列特异性DNA结合转录因子活性
Transcription factor activity, sequence-specific DNA binding3.51 0.48 0.00 0.00 118 GO:0004497 分子功能 MF 单加氧酶活性
Monooxygenase activity1.98 1.27 0.00 0.00 137 GO:0005506 分子功能 MF 铁离子结合
Iron ion binding2.30 1.54 0.00 0.00 147 GO:0020037 分子功能 MF 血红素结合
Heme binding2.47 1.69 0.00 0.00 表 6 差异表达基因KEGG富集部分分析
Table 6 Partial results of analysis on KEGG enrichment of differentially expressed genes
基因数目
Gene number通路编号
Pathway ID描述
Description该KEGG在目标基因
集中占有的比例
The proportionof KEGG in
target gene concentration/%该KEGG在背景中
占有的比例
The proportion of the
KEGG in the background/%未经校正的P值
P value uncorrected校正后的P值
P value corrected78 map00940 苯丙酸生物合成
Phenylpropanoid biosynthesis2.71 1.36 0.00 0.00 26 map00941 类黄酮生物合成
Flavonoid biosynthesis0.90 0.38 0.00 0.00 17 map00945 二苯乙烯类、二芳基庚烷类和姜辣素的生物合成
Stilbenoid, diarylheptanoid and gingerol biosynthesis0.59 0.26 0.00 0.00 71 map04016 MAPK信号通路-植物
MAPK signaling pathway-plant2.47 1.67 0.00 0.00 63 map00500 淀粉和蔗糖代谢
Starch and sucrose metabolism2.19 1.52 0.00 0.01 26 map00350 酪氨酸代谢
Tyrosine metabolism0.90 0.52 0.00 0.01 92 map04075 植物激素信号转导
Plant hormone signal transduction3.20 2.42 0.00 0.01 26 map00040 戊糖和葡萄糖醛酸的相互转化
Pentose and glucuronate interconversions0.90 0.53 0.00 0.01 39 map04146 过氧化物酶体
Peroxisome1.36 0.88 0.00 0.01 28 map00592 α-亚麻酸代谢
alpha-Linolenic acid metabolism0.97 0.60 0.00 0.02 20 map00360 苯丙氨酸代谢
Phenylalanine metabolism0.69 0.40 0.00 0.03 24 map00460 氰胺酸代谢
Cyanoamino acid metabolism0.83 0.50 0.00 0.03 16 map00906 类胡萝卜素生物合成
Carotenoid biosynthesis0.56 0.31 0.00 0.05 16 map00950 异喹啉生物碱的生物合成
Isoquinoline alkaloid biosynthesis0.56 0.31 0.00 0.04 23 map00130 泛醌和其他萜类醌的生物合
Ubiquinone and other terpenoid-quinone biosynthesis0.80 0.50 0.00 0.05 32 map00260 甘氨酸、丝氨酸和苏氨酸代谢
Glycine, serine and threonine metabolism1.11 0.78 0.00 0.06 23 map04712 植物昼夜节律
Circadian rhythm-plant0.80 0.52 0.00 0.06 28 map00051 果糖和甘露糖代谢
Fructose and mannose metabolism0.97 0.65 0.00 0.07 22 map00052 半乳糖代谢
Galactose metabolism0.76 0.50 0.01 0.10 50 map00520 氨基糖和核苷酸糖代谢
Amino sugar and nucleotide sugar metabolism1.74 1.34 0.01 0.10 表 7 20个差异表达基因qRT-PCR的RQ值
Table 7 RQ values of qRT-PCR for 20 differentially expressed genes
基因
Gene正常种
RQ值
RQ value in CK平原种植
退化种 RQ值
RQ value in BZ调节
RegulationRNA-seq调节
Regulation in
RNA-seq抗病反应蛋白 Disease resistance response protein 1.00 b 216.68 a 上调 Up 上调 Up 抗病蛋白 RGA2 Disease resistance protein RGA2 1.00 b 6.29 a 上调 Up 上调 Up 乙烯受体2 Ethylene receptor 2 1.00 a 0.48 b 下调 Down 下调 Down 过氧化氢酶1 Catalase 1 1.00 b 8.23 a 上调 Up 上调 Up 蔗糖合成酶 Sucrose synthase 1.00 b 10.50 a 上调 Up 上调 Up 抗病蛋白 RPS5 Disease resistance protein RPS5 1.00 b 10.02 a 上调 Up 上调 Up 抗病蛋白 Disease resistance protein 1.00 b 10.04 a 上调 Up 上调 Up 类烟草病毒增殖蛋白3 Tobamovirus multiplication protein 3-like 1.00 a 0.78 b 下调 Down 下调 Down 植物抗病反应蛋白 Plant disease resistance response protein 1.00 b 10.01 a 上调 Up 上调 Up F-box家族蛋白 F-box family protein 1.00 b 7.815577 a 上调 Up 上调 Up 超氧化物歧化酶 [Cu-Zn] Superoxide dismutase [Cu-Zn] 1.00 b 9.217215 a 上调 Up 上调 Up 逆转录病毒相关 Pol多聚蛋白系-1 Retrovirus-related Pol polyprotein LINE-1 1.00 a 0.13 b 下调 Down 下调 Down 转座子 RE1的逆转录病毒相关 Pol多蛋白 Retrovirus-related Pol polyprotein from transposon RE1 1.00 a 0.01 b 下调 Down 下调 Down 转座子 TNT逆转录病毒相关 Pol多蛋白 Retrovirus-related Pol polyprotein from transposon TNT 1.00 a 0.06 b 下调 Down 下调 Down BZIP转录因子家族蛋白 BZIP transcription factor family protein 1.00 a 0.46 b 下调 Down 下调 Down 过氧化物酶 Peroxidase 1.00 b 16.00 a 上调 Up 上调 Up 脱落酸受体 PYR1 Abscisic acid receptor PYR1 1.00 a 0.25 b 下调 Down 下调 Down 抗病蛋白 RPS2 Disease resistance protein RPS2 1.00 b 5.40 a 上调 Up 上调 Up 生长素反应因子1 Auxin response factor 1 1.00 a 0.50 b 下调 Down 下调 Down 烟草病毒增殖蛋白1 Tobamovirus multiplication protein 1 1.00 a 0.49 b 下调 Down 下调 Down 甘油醛-3-磷酸脱氢酶 GAPDH 1.00 a 1.00 a - - 注:同行数据后不同小写字母表示显著性差异(P<0.05)。
Note: Different lowercase letters in each line indicate significant difference at 0.05 level. -
[1] 余志平, 林海红, 余俊红, 等. 铅山红芽芋产业发展概况 [J]. 长江蔬菜, 2016(20):34−36. DOI: 10.3865/j.issn.1001-3547.2016.20.015 YU Z P, LIN H H, YU J H, et al. Development of red bud taro industry in Yanshan [J]. Journal of Changjiang Vegetables, 2016(20): 34−36.(in Chinese) DOI: 10.3865/j.issn.1001-3547.2016.20.015
[2] 姜绍通, 程元珍, 郑志, 等. 红芽芋营养成分分析及评价 [J]. 食品科学, 2012, 33(11):269−272. JIANG S T, CHENG Y Z, ZHENG Z, et al. Analysis and evaluation of nutritional components of red bud taro (Colocasia esulenla L. Schott) [J]. Food Science, 2012, 33(11): 269−272.(in Chinese)
[3] 山娜, 杨文俊, 李斌. 举全县之力, 打造铅山红芽芋支柱产业 [J]. 长江蔬菜, 2016(4):9−10. DOI: 10.3865/j.issn.1001-3547.2016.04.005 SHAN N, YANG W J, LI B. Take the efforts of the whole county to build the pillar industry of red bud taro [J]. Journal of Changjiang Vegetables, 2016(4): 9−10.(in Chinese) DOI: 10.3865/j.issn.1001-3547.2016.04.005
[4] 李火金. 铅山红芽芋茎尖脱毒组培繁育及高产栽培 [J]. 中国蔬菜, 2012(2):45−46. LI H J. Virus free tissue culture and high yield cultivation of the shoot tips of the red bud taro [J]. China Vegetables, 2012(2): 45−46.(in Chinese)
[5] 江芹, 廖华俊, 董玲, 等. 红芽芋的丛生芽诱导和再生体系建立 [J]. 分子植物育种, 2015, 13(3):675−679. JIANG Q, LIAO H J, DONG L, et al. Induction of multiple shoot and establishment of regeneration system in Clocasia escalenta schott [J]. Molecular Plant Breeding, 2015, 13(3): 675−679.(in Chinese)
[6] 邓接楼, 曹昊玮, 李玲, 等. 脱毒红芽芋试管芋盆栽种植的农艺性状分析 [J]. 分子植物育种, 2019, 17(22):7500−7506. DENG J L, CAO H W, LI L, et al. Analysis on agronomic traits of virus-free test-tube taro planted in pots [J]. Molecular Plant Breeding, 2019, 17(22): 7500−7506.(in Chinese)
[7] 胡国君, 董雅凤, 张尊平, 等. 植物类病毒脱除技术进展 [J]. 植物保护学报, 2017, 44(2):177−184. HU G J, DONG Y F, ZHANG Z P, et al. Progress in plant viroid elimination techniques [J]. Acta Phytophylacica Sinica, 2017, 44(2): 177−184.(in Chinese)
[8] YANG X X, POPOVA E, SHUKLA M R, et al. Root cryopreservation to biobank medicinal plants: A case study for Hypericum perforatum L [J]. In Vitro Cellular & Developmental Biology-Plant, 2019, 55(4): 392−402.
[9] KULUS D, REWERS M, SEROCKA M, et al. Cryopreservation by encapsulation-dehydration affects the vegetative growth of Chrysanthemum but does not disturb its chimeric structure [J]. Plant Cell, Tissue and Organ Culture (PCTOC), 2019, 138(1): 153−166. DOI: 10.1007/s11240-019-01614-6
[10] LI W B, HARTUNG J S, LEVY L. Quantitative real-time PCR for detection and identification of Candidatus Liberibacter species associated with Citrus huanglongbing [J]. Journal of Microbiological Methods, 2006, 66(1): 104−115. DOI: 10.1016/j.mimet.2005.10.018
[11] WANG Q C, VALKONEN J P T. Cryotherapy of shoot tips: Novel pathogen eradication method [J]. Trends in Plant Science, 2009, 14(3): 119−122. DOI: 10.1016/j.tplants.2008.11.010
[12] HELLIOT B, PANIS B, POUMAY Y, et al. Cryopreservation for the elimination of cucumber mosaic and banana streak viruses from banana (Musa spp.) [J]. Plant Cell Reports, 2002, 20(12): 1117−1122. DOI: 10.1007/s00299-002-0458-8
[13] 李涵, 陆琳, 瞿素萍, 等. 杂交兰种苗超低温脱毒技术研究 [J]. 中国农业科技导报, 2018, 20(1):147−153. LI H, LU L, QU S P, et al. Study of hybrid orchid seedlings on virus elimination using cryopreservation technology [J]. Journal of Agricultural Science and Technology, 2018, 20(1): 147−153.(in Chinese)
[14] 李艳林, 渠慎春, 栾雨婷, 等. 苹果茎尖超低温脱毒体系的建立 [J]. 分子植物育种, 2019, 17(9):2982−2995. LI Y L, QU S C, LUAN Y T, et al. Establishment of cryopreservation detoxification system of apple shoot-tips [J]. Molecular Plant Breeding, 2019, 17(9): 2982−2995.(in Chinese)
[15] 周金鑫, 胡新文, 张海文, 等. ABA在生物胁迫应答中的调控作用 [J]. 农业生物技术学报, 2008, 16(1):169−174. DOI: 10.3969/j.issn.1674-7968.2008.01.032 ZHOU J X, HU X W, ZHANG H W, et al. Regulatory role of ABA in plant response to biotic stresses [J]. Journal of Agricultural Biotechnology, 2008, 16(1): 169−174.(in Chinese) DOI: 10.3969/j.issn.1674-7968.2008.01.032
[16] XANTHOPOULOU A, GANOPOULOS I, PSOMOPOULOS F, et al. De novo comparative transcriptome analysis of genes involved in fruit morphology of pumpkin cultivars with extreme size difference and development of EST-SSR markers [J]. Gene, 2017, 622: 50−66. DOI: 10.1016/j.gene.2017.04.035
[17] UNAMBA C I N, NAG A, SHARMA R K. Next generation sequencing technologies: The doorway to the unexplored genomics of non-model plants [J]. Frontiers in Plant Science, 2015, 6: 1074.
[18] COSTA V, ANGELINI C, DE FEIS I, et al. Uncovering the complexity of transcriptomes with RNA-seq [J]. Journal of Biomedicine and Biotechnology, 2010, 2010: 1−19.
[19] WANG Z, GERSTEIN M, SNYDER M. RNA-Seq: a revolutionary tool for transcriptomics [J]. Nature Reviews Genetics, 2009, 10(1): 57−63. DOI: 10.1038/nrg2484
[20] VIEIRA R L, SILVA A L, ZAFFARI G R, et al. Efficient elimination of virus complex from garlic (Allium sativum L.) by cryotherapy of shoot tips [J]. Acta Physiologiae Plantarum, 2014, 37(1): 1−11.
[21] WANG B, WANG R R, CUI Z H, et al. Potential applications of cryogenic technologies to plant genetic improvement and pathogen eradication [J]. Biotechnology Advances, 2014, 32(3): 583−595. DOI: 10.1016/j.biotechadv.2014.03.003
[22] WANG Q C, PANIS B, ENGELMANN F, et al. Cryotherapy of shoot tips: A technique for pathogen eradication to produce healthy planting materials and prepare healthy plant genetic resources for cryopreservation [J]. Annals of Applied Biology, 2009, 154(3): 351−363. DOI: 10.1111/j.1744-7348.2008.00308.x
[23] 何广深. 百子莲胚性愈伤组织超低温保存中钙离子的分布变化及逆境应答机制初探 [D]. 上海: 上海交通大学, 2014. HE G S. Distribution change of Ca2+ and stress response mechanism research of Agapanthus praecox embryogenic callus during cryopreservation [D]. Shanghai: Shanghai Jiaotong University, 2014.(in Chinese)
[24] 滕进婧, 李梦芸, 郭纯, 等. 冷冻胁迫转金柑MLP2-1基因拟南芥的转录组测序和代谢通路 [J]. 湖南农业大学学报(自然科学版), 2018, 44(4):376−381. TENG J J, LI M Y, GUO C, et al. Transcriptome sequencing and metabolic pathway analysis of transgenic MLP2-1 gene in Arabidopsis under cold stress [J]. Journal of Hunan Agricultural University (Natural Sciences Edition), 2018, 44(4): 376−381.(in Chinese)
[25] 苏谦, 安冬, 王库. 植物激素的受体和诱导基因 [J]. 植物生理学通讯, 2008, 44(6):1202−1208. SU Q, AN D, WANG K. Phytohormone receptors and induced genes in plants [J]. Plant Physiology Communications, 2008, 44(6): 1202−1208.(in Chinese)
[26] OHRI P, BHARDWAJ R, BALI S G, et al. The common molecular players in plant hormone crosstalk and signaling [J]. Current Protein & Peptide Science, 2015, 16(5): 369−388.
[27] 杨家书, 吴畏, 吴友三, 等. 植物苯丙酸类代谢与小麦对白粉病抗性的关系 [J]. 植物病理学报, 1986, 16(3):169−174. YANG J S, WU W, WU Y S, et al. Relation of metabolism of plant phenylalanine and resistance of wheat to powdery mildew [J]. Acta Phytopathologica Sinica, 1986, 16(3): 169−174.(in Chinese)
[28] 陈建中, 盛炳成, 刘克均. 苯丙酸类代谢与苹果对轮纹病抗性的关系 [J]. 果树科学, 1986, 16(3):169−174. CHEN J Z, SHENG B C, LIU K J, et al. The relation between metabolism of phenylalanine and resistance to Physalospora piricola Nose in apple trees [J]. Acta Phytopathologica Sinica, 1986, 16(3): 169−174.(in Chinese)
[29] DUAN P G, RAO Y C, ZENG D L, et al. SMALL GRAIN 1, which encodes a mitogen-activated protein kinase kinase 4, influences grain size in rice [J]. The Plant Journal, 2014, 77(4): 547−557. DOI: 10.1111/tpj.12405
[30] LIU S Y, HUA L, DONG S J, et al. OsMAPK6, a mitogen-activated protein kinase, influences rice grain size and biomass production [J]. The Plant Journal, 2015, 84(4): 672−681. DOI: 10.1111/tpj.13025
[31] LI Y B, FAN C C, XING Y Z, et al. Natural variation in GS5 plays an important role in regulating grain size and yield in rice [J]. Nature Genetics, 2011, 43(12): 1266. DOI: 10.1038/ng.977
[32] 苑智华, 何秀丽, 徐哲, 等. 唐菖蒲球茎形成期蔗糖和淀粉代谢及其相关酶活性 [J]. 林业科学, 2008, 44(8):47−51. DOI: 10.3321/j.issn:1001-7488.2008.08.008 YUAN Z H, HE X L, XU Z, et al. Metabolism and related enzymes activities of sucrose and starch in the stages of bulb formation of Gladiolus hybridus [J]. Scientia Silvae Sinicae, 2008, 44(8): 47−51.(in Chinese) DOI: 10.3321/j.issn:1001-7488.2008.08.008
[33] 孙红梅, 何玲, 王微微, 等. IBA与GA3调控百合鳞片扦插繁殖的 “淀粉-蔗糖” 代谢机制 [J]. 中国农业科学, 2011, 44(4):798−806. DOI: 10.3864/j.issn.0578-1752.2011.04.018 SUN H M, HE L, WANG W W, et al. Mechanism of starch-sucrose metabolism regulated by IBA as well as GA3 during scale cutting propagation in Lilium [J]. Scientia Agricultura Sinica, 2011, 44(4): 798−806.(in Chinese) DOI: 10.3864/j.issn.0578-1752.2011.04.018
[34] HU J P, BAKER A, BARTEL B, et al. Plant peroxisomes: Biogenesis and function [J]. The Plant Cell, 2012, 24(6): 2279−2303. DOI: 10.1105/tpc.112.096586
[35] SANDALIO L M, RODRÍGUEZ-SERRANO M, ROMERO-PUERTAS M C, et al. Role of peroxisomes as a source of reactive oxygen species (ROS) signaling molecules[C]//Peroxisomes and Their Key Role in Cellular Signaling and Metabolism, 2013: 231-225.
[36] MITTLER R, VANDERAUWERA S, SUZUKI N, et al. ROS signaling: The new wave? [J]. Trends in Plant Science, 2011, 16(6): 300−309. DOI: 10.1016/j.tplants.2011.03.007
[37] 于妍, 宋万坤, 刘春燕, 等. 植物天冬氨酸代谢途径关键酶基因研究进展 [J]. 生物技术通报, 2008(S1):7−11, 17. YU Y, SONG W K, LIU C Y, et al. Research development of key enzymes gene on aspartic acid metabolic pathway in plants [J]. Biotechnology Bulletin, 2008(S1): 7−11, 17.(in Chinese)
[38] 马瑞肖, 张慧杰, 李馨慧, 等. 丝氨酸/甘氨酸代谢在肿瘤中的研究进展 [J]. 现代肿瘤医学, 2020, 28(6):1021−1024. DOI: 10.3969/j.issn.1672-4992.2020.06.033 MA R X, ZHANG H J, LI X H, et al. The progress of serine/Glycine metabolism in tumor [J]. Journal of Modern Oncology, 2020, 28(6): 1021−1024.(in Chinese) DOI: 10.3969/j.issn.1672-4992.2020.06.033
-
期刊类型引用(3)
1. 尹明华,肖心怡,方雅轩,万静,木也赛尔·吐鲁洪,陈悦. 壳寡糖浸种对低温下江西铅山红芽芋脱毒试管芋萌发及生理代谢的影响. 农业工程学报. 2024(01): 320-330 .  百度学术
百度学术
2. 曾珍,陈万生,肖莹. 植物必需金属离子对药用植物次生代谢产物生物合成的作用. 植物生理学报. 2022(04): 597-606 .  百度学术
百度学术
3. 张艺欣,邓雨晴,张清招,曾会英,聂学文,龚寅,陈丽丽,侯莹,陈小波,陆春西,韦宏默,尹明华. 江西铅山红芽芋超低温疗法脱毒苗的离体保存. 江苏农业科学. 2021(21): 58-66 .  百度学术
百度学术
其他类型引用(0)




 下载:
下载: