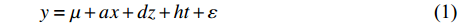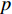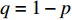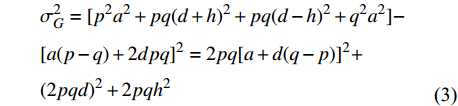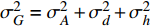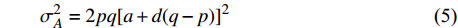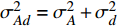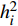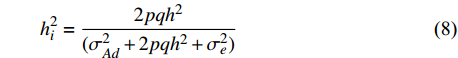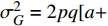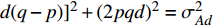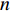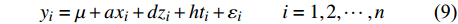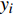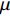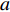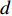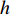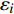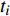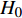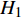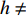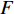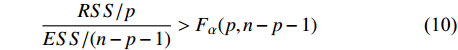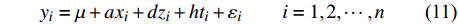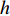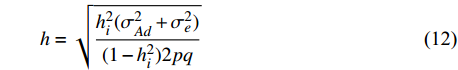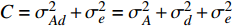A Preliminary Study on Single Imprinted QTL Mapping Based on Random Mating of Natural Populations
-
摘要:目的 探索基于自然群体随机交配的单个印迹QTL的定位方法,分析影响定位准确性的关键因素。方法 若印迹QTL决定的某一性状为数量性状,假设该性状与标记之间的关系存在线性关系,可以采用最小二乘法进行印迹QTL定位和遗传参数的估计。利用计算机模拟单点模拟标记、水稻真实自然群体标记进行印迹QTL定位,比较在不同最小等位基因频率(Minor allele frequency, MAF)、不同遗传率、不同随机交配轮数下的统计功效与参数估计精度,印迹QTL的显著性采用F检验和t检验。结果 通过模拟研究,证明该试验设计对于检测单个印迹QTL是有效的,在MAF大于5%时,印迹遗传率大于10%时,定位与遗传参数估计趋于无偏。结论 采用自然群体随机交配产生作图群体,可以用来进行单个印迹QTL的定位,定位的结果较好,是一种有效的试验设计,为下一步进行多个印迹QTL奠定了基础。Abstract:Objective To explore the application of single imprinted QTL mapping based on random mating of natural populations and analyze key factors affecting the mapping accuracy.Method Assuming the trait determined by the imprinted QTL to be quantitative and a linear relationship between the trait and the markers existed, then the least squares method could be used to estimate the QTL mapping and genetic parameters. The imprinted QTL mapping was generated by computer simulating the single-point markers as well as a real data set of natural population markers. The statistical power and parameter estimation accuracy of different genotype frequencies, heritability rates, and random mating rounds were compared. The significance of imprinted QTLs was determined by using F- and t-tests.Result Through a simulation study, it was proven that the experimental design was effective in detecting a single imprinted QTL. When the MAF was greater than 5% and the imprinted heritability greater than 10%, the iQTLs positions and genetic parameters were presumably unbiasedConclusion Using random mating in the nature to generate mapping populations could conceivably be used to locate single-imprinted QTLs with acceptable results of localization. It seemed to be an effective design for the next step for QTL mapping.
-
Keywords:
- Natural population /
- association analysis /
- F-test /
- imprinted QTL /
- random mating
-
0. 引言
【研究意义】自然群体往往不是严格的遗传群体,如:种质资源、野生种等,它一般不用通过先构建亲本再进行交配产生F2代以后再用自交、回交等方法来产生。相对于结构群体,它的遗传背景一般并不清晰,往往采用关联分析来进行QTL定位。全基因组关联分析[1]主要基于基因的连锁不平衡,用以判断表型性状与遗传标记之间的关联性。在全基因组上对逐点扫描遗传标记,依次判断每个标记与相关表型性状的关联水平。关联分析方法出现以后,有力地推动了数量遗传学的发展,对于水稻[2-3]、玉米[4-5]等作物以及猪[6-7]、牛[8]等家畜的育种改良起了重要的推动作用。印迹效应是一种表观遗传效应,指的是由于亲本产生的配子根据其来源父本或母本,使得配子中某些基因呈现特异的表达或沉默,造成子代体细胞中某些等位基因表达活性不同的现象。印迹在某些植物的胚乳形成[9]、哺乳动物的胚胎发育[10]、人类的一些罕见疾病[11]的形成中起着重要的作用。【前人研究进展】由于全基因组数据往往十分庞大,关联分析对计算的要求较高。为了简化计算,早期研究人员进行关联分析时往往将只考虑加性效应,而忽略显性与印迹等效应。近些年来,有部分研究人员将印迹效应引入到全基因组关联分析中。Marcos等[12]采用SNP作为遗传标记,采用回归方法来定位与猪背膘、日增重等性状相关联的SNP,并估计加性、显性和印迹3种遗传效应值。Hu等[13]采用GWAS技术分析了印迹基因组对小鼠BMI指数的影响,研究表明印迹可以世代传播,对小鼠的BMI指数具有一定的影响。Steven等[14]通过研究发现:基因组印迹对在小鼠对于锥虫的抗性具有重要的作用,并在小鼠染色体上定位到了与锥虫抗性相关的3个印迹QTL。Wen等[15]利用永久F2群体来进行印迹QTL定位,可以无偏估计印迹QTL位置和加性、显性和印迹3种遗传效应。【本研究切入点】自然群体的构建需要花费大量的人力与物力,若将自然群体随机交配产生后代,则相对可以用较低廉的价格获得遗传信息丰富的群体。目前,基于自然群体随机交配的方法来定位印迹QTL目前尚未见到报道。由于基因组印迹具有亲本特异的特点,所以,在进行印迹QTL定位时,需要知道某个株系的父本或母本。直接采用自然群体不能够直接进行印迹QTL的定位,但可以将自然群体作为初始群体,进行随机交配,产生F2群体,可以用较小的初始群体来获得一个大的F2群体,该群体包含的遗传重组事件较为丰富,可以用作印迹QTL定位。【拟解决的关键问题】建立性状与加性、显性、印迹关系的遗传模型、给出单个印迹QTL定位与印迹QTL显著性检验的方法。
1. 试验设计
假设有1个已知标记基因型的自然群体,将自然群体植株间进行随机交配产生随机交配群体,采用随机区组设计,调查二倍体植株性状表型值,生成随机交配群体如图1所示。
2. 遗传模型与定位方法
假设基因组上某个位置上存在与表型性状关联的位点,这个位点控制某个性状,那么我们可以定义下列遗传模型:
y=μ+ax+dz+ht+ε (式1) 式中,y为表型值,μ为群体均值,a代表加性效应,d代表显性效应,h代表印迹效应,ε为随机误差,服从正态分布N(0,
σ2ε );x、z和t为哑变量,假设两个等位基因A和a,A的基因频率为p ,a的基因频率为q=1−p ,在随机交配群体中,若考虑加性效应,显性效应和印迹效应,有4种基因型,各自的频率如表1所示。表 1 自然群体中基因型的频率与哑变量取值Table 1. Frequency of genotypes and value of dummy variables in natural populations基因型 Genotype 频率 Frequency x z t a d h A/A p2 1 0 0 A/a pq 0 1 1 a/A pq 0 1 −1 A/A q2 −1 0 0 我们可以推导出,它的理论均值为:
M=a(p−q)+2pqd (式2) 则其遗传方差为:
σ2G=[p2a2+pq(d+h)2+pq(d−h)2+q2a2]−[a(p−q)+2dpq]2=2pq[a+d(q−p)]2+(2pqd)2+2pqh2 (式3) 等位基因替代效应的平均效应(the average effect of allele substitution)可以通过式4计算得到:
A=a+d(q−p) (式4) 则总遗传方差为
σ2G=σ2A+σ2d+σ2h ,其中根据文献[16-17],加性方差、显性方差和印迹方差可以采用下列公式,加性方差为:σ2A=2pq[a+d(q−p)]2 (式5) 显性方差为:
σ2d=(2pqd)2 (式6) 印迹方差为:
σ2h=2pqh2 (式7) 设
σ2Ad=σ2A+σ2d ,若设印迹效应的遗传率为h2i ,则h2i=2pqh2(σ2Ad+2pqh2+σ2e) (式8) 若不考虑印迹效应,则其遗传方差为
σ2G=2pq[a+ d(q−p)]2+(2pqd)2=σ2Ad 。3. 单标记印迹QTL定位方法
假设有一个自然群体进行随机交配后,产生了个体数目为
n 的群体,为此我们建立了以下线性模型yi=μ+axi+dzi+hti+εii=1,2,⋯,n (式9) 式中,
yi 为表型值,μ 为群体均值,a 代表加性效应,d 代表显性效应,h 代表印迹效应,εi 为随机误差,服从正态分布N(0,σ2e) ;xi、zi 和ti 为哑变量,其值的确定见表1。为了简单起见,本研究采取单标记关联分析,采用最小二乘法回归分析,对基因组上的标记进行逐点扫描,对于式9,我们提出假设
H0 ∶a=d=h= 0 ,其备择假设H1 ∶a,d 和h 不全为零。若仅检验印迹效应,我们提出假设H0 ∶h=0 ,其备择假设H1 ∶h≠ 0,我们采用F 检验来检测标记的显著性,因为是单个标记的检验,直接用0.05或0.01下的F 临界值进行判断。由于这里有3种遗传效应,那么显然的,如果:对于给点的显著性水平,可以构建统计量,该统计量服从F(p, n−p−1)的F分布,则有式10。
RSS/pESS/(n−p−1)>Fα(p,n−p−1) (式10) 式中,RSS代表随机残差平方和,ESS为回归平方和,n为样本数,p为变量个数,此处取值为3。如果方程总体通过了显著性检验以后,再逐个对x、z、t做t检验,检验各变量是否显著。F统计量与t统计量计算简单,使用计算机运算速度较快,可以逐点计算全基因组上每个位点的F值,并对每个位点的3种效应估计值进行t检验,将F最大(超过临界值,且通过t检验)的点作为可能的印迹QTL位点。
4. 模拟研究
为了验证该方法的可行性,我们采用计算机来模拟验证,由于本文仅是基于自然群体随机交配的进行印迹QTL定位方法的初步研究,目的在探讨其可行性,所以进行2个模拟:第一是采用单点模拟,假设已知QTL位置,仅考察其参数估计的准确度和精度,及其是否达到显著性;第二是采用实际的水稻的基因型数据进行模拟,验证其可行性。
4.1 单点模拟研究
由于自然群体有别于结构群体,自然群体的遗传结构是不清晰的,在群体中有大量的进化事件掺杂其中,群体结构较为复杂。在群体中,等位基因A,a的基因频率往往不是1∶1,甚至有可能是很极端的,如可能在0.05以下,就是罕见变异。那么自然群体中在不同基因频率下,基于自然群体随机交配产生的群体,我们所建立的遗传模型和提出相应地印迹QTL检测方法与效应估计方法是否还能有效,我们通过模拟来进行检验。在这里,仅考察所建立的遗传模型在不同的MAF下相应地参数估计和显著检验方法是否可行。
我们假定仅有单点标记,在遗传模型中
yi=μ+axi+dzi+hti+εii=1,2,⋯,n 进行参数估计,在此基础上,首先我们对方程进行F检验,看其是否存在QTL(
a,d 和h 不全为零),在此基础上进一步对印迹效应做显著性检验(h≠0 )。根据式9可以计算出印迹效应的大小如式12所示。
h=√h2i(σ2Ad+σ2e)(1−h2i)2pq (式11) 若令
C=σ2Ad+σ2e=σ2A+σ2d+σ2e ,在模拟中可以采用如下设置:C 取一常数,给定印迹遗传率,从而计算出印迹效应,如式13所示:h=√Ch2i(1−h2i)2pq (式12) 再计算环境方差,最终产生表型值,这样就可以考虑遗传率的不同,随机交配次数不同的功效,参数估计等。
文献[18]根据印迹与加性、显性的关系将它们分成5种印迹类型,如表2所示。
表 2 印迹类型与参数设置与缩写Table 2. Imprint types, parameter settings, and abbreviations印迹类型
Imprinting type简写
abbreviation参数
parameter来源于父本的表达
Parental expression, PaternalPEP d=0且a=h 来源于母本的表达
Parental expression, MaternalPEM d=0且a=−h 显性印迹(双相)
Dominance imprinting, BipolarDIB a=0且d=0 显性印迹(单相)超显性
Dominance imprinting, Polar, Over-dominanceDIPOD a=0且d=h 显性印迹(单相)低于显性
Dominance imprinting, Polar, Under dominanceDIPUD a=0且d=−h 对这5种类型的iQTL进行模拟定位并对模拟结果进行讨论,由于在GWAS研究中,研究人员关心的往往是MAF大于0.05的这部分标记,所以,将MAF,即
p 设置0.05、0.1两种情况,设q=1−p,常数C 取4、10两种情况,iQTL的遗传率设置为5%、10%、15%三个水平,印迹效应根据式12进行推算。根据表2中的5种印迹类型得出a 、d的值。假设自然群体作为初始群体,包含100个株系,利用该自然群体进行随机交配6、20轮产生F2群体。每种情况重复100次,100次参数估计的标准误偏差提供了参数估计误差标准,显著性水平取为0.05,限于篇幅,此处仅列出C=10,交配20轮的结果,结果见表3。表 3 单点模拟5种类型的iQTL定位结果Table 3. Single-point simulation of 5 types of iQTL mapping resultsMAF/% H/% DIPOD DIPUD DIB PEM PEP a d h P/% a d h P/% a d h P/% a d h P/% a d h P/% RV 0 2.35 2.35 0 2.35 −2.35 0 0 2.35 2.35 0 −2.35 2.35 0 2.35 5 5 0.18 2.2 2.4 85 0.07 2.25 −2.4 93 0.12 −0.1 2.35 88 2.41 −0.06 −2.35 91 2.16 0.24 2.35 87 (1.17) (1.22) (0.31) (1.22) (1.23) (0.33) (1.19) (1.24) (0.37) (1.16) (1.22) (0.31) (1.04) (1.13) (0.29) RV 0 3.42 3.42 0 3.42 −3.42 0 0 3.42 3.42 0 −3.42 3.42 0 3.42 5 10 0.09 3.31 3.45 83 0 3.47 −3.38 82 −0.02 −0.02 3.43 86 3.46 0.03 −3.45 92 3.37 0.1 3.38 86 (1.19) (1.21) (0.35) (1.01) (1.06) (0.31) (1.19) (1.25) (0.35) (1.34) (1.35) (0.35) (1.03) (1.05) (0.29) RV 0 4.31 4.31 0 4.31 −4.31 0 0 4.31 4.31 0 −4.31 4.31 0 4.31 5 15 0.07 4.28 4.34 90 −0.01 4.31 −4.35 90 0.03 −0.05 4.25 90 4.36 −0.05 −4.31 88 4.42 −0.08 4.33 85 (1.2) (1.25) (0.27) (1.18) (1.26) (0.29) (1.24) (1.24) (0.33) (1.03) (1.03) (0.3) (1.03) (1.08) (0.32) RV 0 1.71 1.71 0 1.71 −1.71 0 0 1.71 1.71 0 −1.71 1.71 0 1.71 10 5 −0.02 1.75 1.72 100 −0.04 1.75 −1.73 100 −0.04 0.06 1.72 100 1.64 0.06 −1.67 100 1.69 −0.02 1.69 100 (0.53) (0.6) (0.21) (0.53) (0.55) (0.21) (0.57) (0.63) (0.23) (0.47) (0.58) (0.24) (0.57) (0.63) (0.22) RV 0 2.48 2.48 0 2.48 −2.48 0 0 2.48 2.48 0 −2.48 2.48 0 2.48 10 10 −0.05 2.52 2.5 100 −0.05 2.53 −2.51 100 −0.02 0.03 2.51 100 2.54 −0.04 −2.51 100 2.59 −0.06 2.52 100 (0.55) (0.61) (0.23) (0.57) (0.63) (0.25) (0.61) (0.7) (0.26) (0.51) (0.56) (0.2) (0.6) (0.62) (0.23) RV 0 3.13 3.13 0 3.13 −3.13 0 0 3.13 3.13 0 −3.13 3.13 0 3.13 10 15 −0.04 3.18 3.12 100 −0.08 3.22 −3.13 100 −0.08 0.06 3.13 100 3.24 −0.05 −3.12 100 3.18 0.01 3.12 100 (0.49) (0.57) (0.23) (0.52) (0.57) (0.23) (0.62) (0.65) (0.26) (0.47) (0.53) (0.22) (0.55) (0.59) (0.24) 注:表中括号内为标准差,此处列出的为C=10,交配轮数为20的结果,表中H代表遗传率(Heritability),RV代表真值(Real value),P代表统计功效(Power)
Note: The number in parentheses in the table is the standard deviation,C=10 and cross rounds is 20, H represents heritability, RV means real value and p means statistical power.结果表明:在MAF较小,为0.05时,即使iQTL的遗传率达到15%,其检测功效和参数估计的标准误都较高,当MAF达到0.1以上时,其检测功效就较高了;随着MAF的增大,遗传率的增加,其估计的标准误逐渐减小。在和遗传率相同的条件下,随着C值的增加,其参数估计的标准误也增加,其原因还有待于进一步的研究。即使在MAF高于0.05时,当遗传率为5%时,效应值估计有一定的误差,检测功效也较低,当遗传率在10%以上的时候,效应估计就较为精准。所以可以看出基因频率和遗传率是影响iQTL定位效果的因素。此外,随着交配次数的增加,iQTL的检测功效随之增加,参数估计的标准误随之减少,从5种iQTL来看,PEP、PEM、DIPOD、DIPUD在MAF、遗传率、C值相同的情况下,其iQTL检测效果相似,然而DIB的iQTL检测效果略差些,这说明加性效应和显性效应影响了iQTL定位。
综上所述,如果某个性状的表型值由一个iQTL确定,那么不论它是上述五种类型中的哪种类型,只要在MAF大于0.1且遗传率大于10%时,其检测功效均较好,说明我们的提出的方法在自然群体随机交配产生的F2群体上定位单个iQTL是有效的。
4.2 基于水稻基因型数据单个位点的模拟研究
为了验证该方法在实际中的实施情况,我们采用韩斌院士课题组发布的水稻自然群体的数据集[19],该数据集收集了960多种水稻种质资源。为了简化研究的问题,对原始数据进行了预处理,把其中粳稻的254个株系取出,并将MAF小于0.05的SNP位点去掉,并去除了所有的缺失位点,最终得到了一个包含16 382个SNP,在本模拟中,假定这些SNP是等间距分布在一条染色体上,每个SNP间的距离为1 kb,254个株系的数据集作为原始群体。原始群体采用随机交配的方式生成随机交配群体,在进行模拟前,统计各个位点的SNP频率。从图2可知,在自然群体中,每个位置上的SNP的MAF并不是均衡的。
假定一个位于470 kb位置的这个SNP与某个表型性状关联,群体均值为μ=10。模拟前,先计算该位点的SNP频率p和q,印迹效应根据公式12进行推算(C值取2,4,6三种组合)。根据表2中的5种印迹类型得出a, d的值,根据前面的模拟结果,iQTL的遗传力水平设置为10%和15%,利用自然群体进行3、5、7次随机交配,产生随机交配群体。每种模拟情况重复100次,100次参数估计的标准误偏差提供了参数估计误差标准,阈值可用Bonferroni校正,显著性水平为0.05。此处,限于篇幅,仅列出C值取2、6,交配轮数为7次的结果,见表4,交配以后得到的随机交配群体中470 kb处的SNP的频率p为0.54,则q为0.46,接近于1∶1。由于在模拟时选择的iQTL位置上的MAF接近于0.5,从真实数据的模拟结果来看:当遗传率为10%、15%时,5种类型的iQTL定位都具有较好的结果,iQTL所在位置的估计较为准确,各参数估计的准确度和精度达到较好的效果,而且检测功效也较高,在15%时所有类型的iQTL检测功效均为100%。结果表明,随着交配次数的增加,参数估计的精度也得到提高,这符合我们的构想,当样本量增加时,其估计的准确度和精度得到提高;对于常数C值,从表可以看出,随着C值的增加,其参数估计的精度逐渐下降,其原因还有待于进一步研究;从5种类型的印迹QTL来看,在相同的条件下,DIB的检测功效较差,而其余4种DIPOD,DIPUD,PEM,PEP检测的功效相近,由此可见:加性效应和显性效应对iQTL的检测具有一定的影响。
表 4 不同C值下5种印迹类型的iQTL定位结果Table 4. iQTL mapping results of 5 imprint types under varied C valuesH C DIPOD DIPUD DIB PEM PEP POS/kbp a d h POS/kbp a d h POS/kbp a d h POS/Kbp a d h POS/kbp a d h P/% RV 470 0 0.73 0.73 470 0 0.73 −0.73 470 0 0 0.73 470 0.73 0 −0.73 470 0.73 0 0.73 10 2 470 −0.01 0.74 0.73 470 0.01 0.74 −0.72 470 0 0 0.74 470 0.73 0 −0.73 470 −0.01 0.74 0.73 100 0 (0.1) (0.13) (0.07) 0 (0.09) (0.13) (0.07) 0 (0.1) (0.14) (0.08) 0 (0.1) (0.12) (0.08) 0 (0.1) (0.1) (0.1) RV 470 0 1.26 1.26 470 0 1.26 −1.26 470 0 0 1.26 470 1.26 0 −1.26 470 1.26 0 1.26 10 6 470 0.02 1.25 1.28 470 −0.04 1.25 −1.28 470.02 0.01 0.01 1.26 470 1.27 0 −1.25 470 0.02 1.25 1.28 100 0 (0.18) (0.23) (0.11) 0 (0.16) (0.18) (0.13) (0.2) (0.16) (0.22) (0.15) 0 (0.17) (0.21) (0.12) 0 (0.18) (0.2) (0.1) RV 470 0 0.93 0.93 470 0 0.93 −0.93 470 0 0 0.93 470 0.93 0 −0.93 470 0.93 0 0.93 15 2 470 −0.01 0.92 0.93 470 0 0.9 −0.93 470 −0.01 0 0.92 470 0.95 0 −0.92 470 −0.01 0.92 0.93 100 0 (0.1) (0.12) (0.07) 0 (0.1) (0.13) (0.07) 0 (0.11) (0.13) (0.09) 0 (0.1) (0.14) (0.07) 0 (0.1) (0.1) (0.1) RV 470 0 1.6 1.6 470 0 1.6 −1.6 470 0 0 1.6 470 1.6 0 −1.6 470 1.3 0 1.6 15 6 470 0 1.59 1.59 470 0 1.62 −1.63 470 0.01 0.01 1.6 470 1.59 −0.03 −1.62 470 0 1.59 1.59 100 0 (0.17) (0.2) (0.13) 0 (0.18) (0.22) (0.13) 0 (0.16) (0.19) (0.12) 0 (0.17) (0.2) (0.12) 0 (0.17) (0.2) (0.1) 注:表中括号内为标准差,此处列出C=2、6,交配轮数为7次的结果,表中H代表遗传率,H代表遗传率(Heritability),C代表C值(C value),RV代表真值(Real value),P代表统计功效(Power),POS代表iQTL在染色体上的位置(iQTL position)。
Note: The number in parentheses in the table is the standard deviation,the results listed here are the C values are 2 and 4,the number of mating rounds is 7, C represents C-value, RV represents real value, P represents statistical power and POS means the iQTL’s position on chromosome.5. 讨论
我们提出了基于自然群体随机交配产生的F2群体进行iQTL定位的思想,构建其初步的定位方法,通过单点模拟,全基因组的单点模拟说明基于自然群体随机交配产生的群体进行iQTL定位是可行的,当然这里没有考虑其他因素(如群体结构等)的干扰。模拟表明,在不考虑其他因素的影响下,该iQTL定位方法的检测功效,以及参数估计的准确度和精度都较好。
虽然自然群体的群体遗传结构不清晰,但在GWAS时代,测序成本不断降低,甚至可以直接对个体进行测序。所以,基因型频率可以看成已知的,基于自然群体随机交配产生的群体,只要交配次数足够,就可以产生较大的样本;从初步研究模拟也可以看出,随着交配次数的增加,其检测功效,参数估计的精度也增加。
在全基因组关联分析(GWAS)中,是以长期重组后保留下来的基因或位点间连锁不平衡为基础,在获得群体表型数据和基因型数据后,采用统计方法检测标记与性状之间关联的分析方法,一般以自然群体或种质资源为研究材料,遗传结构较为复杂。在进行GWAS时,要求遗传背景一致或者相似的群体,但在未知群体结构的关联分析研究中,由不同亚群引起的MAF的差异会造成假关联。 通常采用基因组控制、基于家系的关联分析检验和主成分分析等方法控制。一般而言:基于自然群体随机交配产生的群体会产生结构分层,所以在这种情况下,必须要控制因为结构分层造成的假关联,在后续的工作中,可以利用混合线性模型,传递不平衡分析等其他方法来进行印迹QTL定位方法的研究。
-
表 1 自然群体中基因型的频率与哑变量取值
Table 1 Frequency of genotypes and value of dummy variables in natural populations
基因型 Genotype 频率 Frequency x z t a d h A/A p2 1 0 0 A/a pq 0 1 1 a/A pq 0 1 −1 A/A q2 −1 0 0 表 2 印迹类型与参数设置与缩写
Table 2 Imprint types, parameter settings, and abbreviations
印迹类型
Imprinting type简写
abbreviation参数
parameter来源于父本的表达
Parental expression, PaternalPEP d=0且a=h 来源于母本的表达
Parental expression, MaternalPEM d=0且a=−h 显性印迹(双相)
Dominance imprinting, BipolarDIB a=0且d=0 显性印迹(单相)超显性
Dominance imprinting, Polar, Over-dominanceDIPOD a=0且d=h 显性印迹(单相)低于显性
Dominance imprinting, Polar, Under dominanceDIPUD a=0且d=−h 表 3 单点模拟5种类型的iQTL定位结果
Table 3 Single-point simulation of 5 types of iQTL mapping results
MAF/% H/% DIPOD DIPUD DIB PEM PEP a d h P/% a d h P/% a d h P/% a d h P/% a d h P/% RV 0 2.35 2.35 0 2.35 −2.35 0 0 2.35 2.35 0 −2.35 2.35 0 2.35 5 5 0.18 2.2 2.4 85 0.07 2.25 −2.4 93 0.12 −0.1 2.35 88 2.41 −0.06 −2.35 91 2.16 0.24 2.35 87 (1.17) (1.22) (0.31) (1.22) (1.23) (0.33) (1.19) (1.24) (0.37) (1.16) (1.22) (0.31) (1.04) (1.13) (0.29) RV 0 3.42 3.42 0 3.42 −3.42 0 0 3.42 3.42 0 −3.42 3.42 0 3.42 5 10 0.09 3.31 3.45 83 0 3.47 −3.38 82 −0.02 −0.02 3.43 86 3.46 0.03 −3.45 92 3.37 0.1 3.38 86 (1.19) (1.21) (0.35) (1.01) (1.06) (0.31) (1.19) (1.25) (0.35) (1.34) (1.35) (0.35) (1.03) (1.05) (0.29) RV 0 4.31 4.31 0 4.31 −4.31 0 0 4.31 4.31 0 −4.31 4.31 0 4.31 5 15 0.07 4.28 4.34 90 −0.01 4.31 −4.35 90 0.03 −0.05 4.25 90 4.36 −0.05 −4.31 88 4.42 −0.08 4.33 85 (1.2) (1.25) (0.27) (1.18) (1.26) (0.29) (1.24) (1.24) (0.33) (1.03) (1.03) (0.3) (1.03) (1.08) (0.32) RV 0 1.71 1.71 0 1.71 −1.71 0 0 1.71 1.71 0 −1.71 1.71 0 1.71 10 5 −0.02 1.75 1.72 100 −0.04 1.75 −1.73 100 −0.04 0.06 1.72 100 1.64 0.06 −1.67 100 1.69 −0.02 1.69 100 (0.53) (0.6) (0.21) (0.53) (0.55) (0.21) (0.57) (0.63) (0.23) (0.47) (0.58) (0.24) (0.57) (0.63) (0.22) RV 0 2.48 2.48 0 2.48 −2.48 0 0 2.48 2.48 0 −2.48 2.48 0 2.48 10 10 −0.05 2.52 2.5 100 −0.05 2.53 −2.51 100 −0.02 0.03 2.51 100 2.54 −0.04 −2.51 100 2.59 −0.06 2.52 100 (0.55) (0.61) (0.23) (0.57) (0.63) (0.25) (0.61) (0.7) (0.26) (0.51) (0.56) (0.2) (0.6) (0.62) (0.23) RV 0 3.13 3.13 0 3.13 −3.13 0 0 3.13 3.13 0 −3.13 3.13 0 3.13 10 15 −0.04 3.18 3.12 100 −0.08 3.22 −3.13 100 −0.08 0.06 3.13 100 3.24 −0.05 −3.12 100 3.18 0.01 3.12 100 (0.49) (0.57) (0.23) (0.52) (0.57) (0.23) (0.62) (0.65) (0.26) (0.47) (0.53) (0.22) (0.55) (0.59) (0.24) 注:表中括号内为标准差,此处列出的为C=10,交配轮数为20的结果,表中H代表遗传率(Heritability),RV代表真值(Real value),P代表统计功效(Power)
Note: The number in parentheses in the table is the standard deviation,C=10 and cross rounds is 20, H represents heritability, RV means real value and p means statistical power.表 4 不同C值下5种印迹类型的iQTL定位结果
Table 4 iQTL mapping results of 5 imprint types under varied C values
H C DIPOD DIPUD DIB PEM PEP POS/kbp a d h POS/kbp a d h POS/kbp a d h POS/Kbp a d h POS/kbp a d h P/% RV 470 0 0.73 0.73 470 0 0.73 −0.73 470 0 0 0.73 470 0.73 0 −0.73 470 0.73 0 0.73 10 2 470 −0.01 0.74 0.73 470 0.01 0.74 −0.72 470 0 0 0.74 470 0.73 0 −0.73 470 −0.01 0.74 0.73 100 0 (0.1) (0.13) (0.07) 0 (0.09) (0.13) (0.07) 0 (0.1) (0.14) (0.08) 0 (0.1) (0.12) (0.08) 0 (0.1) (0.1) (0.1) RV 470 0 1.26 1.26 470 0 1.26 −1.26 470 0 0 1.26 470 1.26 0 −1.26 470 1.26 0 1.26 10 6 470 0.02 1.25 1.28 470 −0.04 1.25 −1.28 470.02 0.01 0.01 1.26 470 1.27 0 −1.25 470 0.02 1.25 1.28 100 0 (0.18) (0.23) (0.11) 0 (0.16) (0.18) (0.13) (0.2) (0.16) (0.22) (0.15) 0 (0.17) (0.21) (0.12) 0 (0.18) (0.2) (0.1) RV 470 0 0.93 0.93 470 0 0.93 −0.93 470 0 0 0.93 470 0.93 0 −0.93 470 0.93 0 0.93 15 2 470 −0.01 0.92 0.93 470 0 0.9 −0.93 470 −0.01 0 0.92 470 0.95 0 −0.92 470 −0.01 0.92 0.93 100 0 (0.1) (0.12) (0.07) 0 (0.1) (0.13) (0.07) 0 (0.11) (0.13) (0.09) 0 (0.1) (0.14) (0.07) 0 (0.1) (0.1) (0.1) RV 470 0 1.6 1.6 470 0 1.6 −1.6 470 0 0 1.6 470 1.6 0 −1.6 470 1.3 0 1.6 15 6 470 0 1.59 1.59 470 0 1.62 −1.63 470 0.01 0.01 1.6 470 1.59 −0.03 −1.62 470 0 1.59 1.59 100 0 (0.17) (0.2) (0.13) 0 (0.18) (0.22) (0.13) 0 (0.16) (0.19) (0.12) 0 (0.17) (0.2) (0.12) 0 (0.17) (0.2) (0.1) 注:表中括号内为标准差,此处列出C=2、6,交配轮数为7次的结果,表中H代表遗传率,H代表遗传率(Heritability),C代表C值(C value),RV代表真值(Real value),P代表统计功效(Power),POS代表iQTL在染色体上的位置(iQTL position)。
Note: The number in parentheses in the table is the standard deviation,the results listed here are the C values are 2 and 4,the number of mating rounds is 7, C represents C-value, RV represents real value, P represents statistical power and POS means the iQTL’s position on chromosome. -
[1] 凃欣, 石立松, 汪樊, 等. 全基因组关联分析的进展与反思 [J]. 生理科学进展, 2010, 41(2):87−94. TU X, SHI LS, WANG F, WANG Q. Genomewide Association Study: Advances, Challenges and Deliberation [J]. Progress in Physiological Sciences, 2010, 41(2): 87−94.(in Chinese)
[2] LI F M, XIE J Y, ZHU X Y, et al. Genetic basis underlying correlations among growth duration and yield traits revealed by GWAS in rice(Oryza sativa L.) [J]. Frontiers in Plant Science, 2018, 9: 650. DOI: 10.3389/fpls.2018.00650
[3] WANG H R, XU X, VIEIRA F G, et al. The power of inbreeding: NGS-based GWAS of rice reveals convergent evolution during rice domestication [J]. Molecular Plant, 2016, 9(7): 975−985. DOI: 10.1016/j.molp.2016.04.018
[4] 田润苗, 张雪海, 汤继华, 等. 玉米种子萌发相关性状的全基因组关联分析 [J]. 作物学报, 2018, 44(5):672−685. DOI: 10.3724/SP.J.1006.2018.00672 TIAN R M, ZHANG X H, TANG J H, et al. Genome-wide association studies of seed germination related traits in maize [J]. Acta Agronomica Sinica, 2018, 44(5): 672−685.(in Chinese) DOI: 10.3724/SP.J.1006.2018.00672
[5] 邵晓宇, 宋希云, 潘顺祥, 等. 玉米穗粗性状的全基因组关联分析及QTL元分析 [J]. 植物生理学报, 2017, 53(12):2091−2102. SHAO XY, SONG XY, PAN S X, et al. Genome-wide association study and Meta-QTL analysis of ear diameter trait in maize [J]. Plant Physiology Communications, 2017, 53(12): 2091−2102.(in Chinese)
[6] WANG K J, LIU Y F, XU Q, et al. A post-GWAS confirming GPAT3 gene associated with pig growth and a significant SNP influencing its promoter activity [J]. Animal Genetics, 2017, 48(4): 478−482. DOI: 10.1111/age.12567
[7] UZZAMAN M R, PARK J E, LEE K T, et al. A genome-wide association study of reproductive traits in a Yorkshire pig population [J]. Livestock Science, 2018, 209: 67−72. DOI: 10.1016/j.livsci.2018.01.005
[8] IBEAGHA-AWEMU E M, PETERS S O, AKWANJI K A, et al. High density genome wide genotyping-by-sequencing and association identifies common and low frequency SNPs, and novel candidate genes influencing cow milk traits [J]. Scientific Reports, 2016, 6: 31109. DOI: 10.1038/srep31109
[9] JESSICA A R, DANIEL Z. Evolution and function of genomic imprinting in plants [J]. Genes & Development, 2015, 29(24): 2517−2531.
[10] PILVAR D, REIMAN M, PILVAR A, et al. Parent-of-origin-specific allelic expression in the human placenta is limited to established imprinted loci and it is stably maintained across pregnancy [J]. Clinical Epigenetics, 2019, 11: 94. DOI: 10.1186/s13148-019-0692-3
[11] PETERS J. The role of genomic imprinting in biology and disease: an expanding view [J]. Nature Reviews Genetics, 2014, 15(8): 517−530. DOI: 10.1038/nrg3766
[12] LOPES M S, BASTIAANSEN J W M, JANSS L, et al. Estimation of additive, dominance, and imprinting genetic variance using genomic data [J]. G3-Genes Genomes Genetics, 2015, 5(12): 2629−2637.
[13] HU Y D, ROSA G J, GIANOLA D. A GWAS assessment of the contribution of genomic imprinting to the variation of body mass index in mice [J]. BMC Genomics, 2015, 16: 576. DOI: 10.1186/s12864-015-1721-z
[14] CLAPCOTT S J, TEALE A J, KEMP S J. Evidence for genomic imprinting of the Major QTL controlling susceptibility to trypanosomiasis in mice [J]. Parasite Immunology, 2000, 22(5): 259−263. DOI: 10.1046/j.1365-3024.2000.00308.x
[15] WEN Y X, WU W R. Mapping of imprinted quantitative trait loci using immortalized F2 populations [J]. PLoS One, 2014, 9(3): e92989. DOI: 10.1371/journal.pone.0092989
[16] DE KONING D J, BOVENHUIS H, VAN ARENDONK J A. On the detection of imprinted 513 quantitative trait loci in experimental crosses of outbred species [J]. Genetics, 2002, 161(2): 931−938.
[17] FALCONER D S, MACKAY T F C. An Introduction to Quantitative Genetics[M]. Ed. 4. Longman Group, Essex, UK, 1996:12-28.
[18] CHEVERUD J M, HAGER R, ROSEMAN C, et al. Genomic imprinting effects on adult body composition in mice [J]. Proceedings of the National Academy of Sciences of the United States of America, 2008, 105(11): 4253−4258. DOI: 10.1073/pnas.0706562105
[19] HUANG X H, ZHAO Y, WEI X H, et al. Genome-wide association study of flowering time and grain yield traits in a worldwide collection of rice germplasm [J]. Nature Genetics, 2012, 44(1): 32−39. DOI: 10.1038/ng.1018
-
期刊类型引用(2)
1. 郑珂晖,黎哲镇,温永仙. 基于自然群体随机交配的多iQTL定位的模拟. 分子植物育种. 2023(20): 6793-6800 .  百度学术
百度学术
2. 黎哲镇,郑珂辉,周富杰,叶景山,温永仙. LASSO算法在基于自然群体随机交配的多iQTL定位中的应用. 福建农林大学学报(自然科学版). 2020(03): 418-424 .  百度学术
百度学术
其他类型引用(4)





 下载:
下载: