16S rRNA and COI Gene Barcodes Applied for Identification of Ornamental Fishes
-
摘要: 应用通用引物测定观赏鱼市场上价值较高的红尾金龙鱼、黄尾龙鱼、青龙鱼、珍珠龙鱼、银龙鱼、神仙鱼、非洲珠宝鱼、接吻鱼、珍珠毛足鲈、蓝钻石孔雀、孔雀鱼、锦鲤等12种观赏鱼的16S rRNA基因和COI基因部分序列。应用DNA序列分析软件,对这些基因进行同源性比较和系统发育分析。通过GenBank中的核酸BLAST和BOLD SYSTEMS数据库对这些基因序列进行比对,结果表明16S rRNA基因和COI基因可以在物种水平作为基因条码对亚洲龙鱼、珍珠龙鱼、银龙鱼和神仙鱼等观赏鱼进行准确的鉴别,而且应用COI基因构建的系统发育树比16S rRNA基因更加接近现有的鱼类分类体系。但对形态十分相似的慈鲷等观赏鱼,以及红尾金龙鱼、黄尾龙鱼、青龙鱼等同一个物种不同品系的观赏鱼依靠16S rRNA基因或COI基因都无法准确鉴定,需要研究进化速度更快的DNA基因条码。Abstract: Partial sequences of 16S rRNA genes and mitochondrial cytochrome oxidase subunit I (COI) genes from 12 highly valuable ornamental fish species were amplified using universal primers. The species included the red-tail golden arowana, the yellow-tail arowana, the green arowana, Scleropages formosus, Scleropages jardinii, Osteoglossum bicirrhosum, Pterophyllum scalare, Hemichromis letourneuxi, Aulonocara stuartgranti, Helostoma temminkii, Poecilia reticulate and Trichopodus leerii. The homology and phylogenetic analyses on the genes were carried out by using DNA sequence analysis software, and compared with the databases at GenBank and Bold Systems. The sequence analysis indicated that the information on 16S rRNA and COI genes might be applicable for distinguishing S. formosus, S. jardinii, O. bicirrhosum and P. reticulatefrom one another. However, the phylogenetic tree constructed with the COI gene would be more closely related to the conventional classification than 16S rRNA. Regardless, the use of 16S rRNA and/or COI gene was not deemed sufficient to differentiate or identify very similar genera or species, such as cichlids or different lines of a same species, nor among the red-tail arowana, the yellow-tail arowana and the green arowana. It was suggested that DNA barcoding with higher evolution rate be studied to overcome the shortcoming of the current method.
-
Keywords:
- gene barcode /
- ornamental fish /
- species identification /
- 16S rRNA /
- COI
-
随着全球观赏鱼产业快速发展,我国已成为全球最大的观赏鱼繁育和消费市场。我国每年从世界20多个国家和地区进口各类淡水、海水观赏鱼近千万尾,同时中国优势的金鱼、锦鲤和部分热带鱼也大量出口到全球近30个国家或地区[1]。由于观赏鱼的价值因种类和品质而存在巨大差异,如同属于亚洲龙鱼的红龙、金龙以及青龙等龙鱼,其幼鱼形态十分相似,但长大后欣赏价值差异巨大,因此其经济价值也有很大差异,有必要对这些高价值的观赏鱼品种进行准确的鉴定。传统的鱼类分类方法依据鱼体的形态结构,由于鱼类的表型特征具有一定的可塑性和生长发育阶段性,因此仅仅依据形态特征进行分类很容易出错。尤其对一些形态差异很小、亲缘关系很近的观赏鱼类,很难进行准确的鉴定。
DNA条形码是通过对一个标准目的基因的DNA序列作为标记对物种进行快速、准确和自动化鉴定。Hebert于2003年首次提出了DNA条形码的概念[2]。该技术依据物种内在的具有特异性和稳定性的遗传物质组成进行鉴定,避免了传统分类时因为趋同或者生境影响而产生表型差异引起的物种鉴定差错。线粒体细胞色素C氧化酶I (cytochrome coxidase I,COI) 基因和16S rRNA基因常常被用于物种的分类和鉴定。尤其是COI基因,因其拥有适宜的长度,具有相对高的种内变异和低度的种间变化,富含较多的系统发育信号,容易被通用引物扩增,成为主要的DNA条形码之一。生命条码数据系统(barcode of life data system,BOLD) 正是基于COI基因650 bp长度的序列建立的。Steinke等通过COI基因对加拿大进口的391种观赏鱼种进行鉴定,除了3种怀疑是杂交或新物种的鱼无法通过DNA条形码区分外,98%的鱼可准确鉴别,并通过遗传距离分析发现了9种隐藏鱼种,说明COI基因用于鱼种鉴定具有快速、准确的优势[3]。Kim等用DNA条形码技术鉴定了286种淡水鱼和1 339种海水鱼,解决了传统分类方法的局限性[4]。Breman等对非洲坦噶尼喀湖的96种形态种慈鲷进行研究,尽管DNA条形码识别技术在复合物种水平的作用有限,但仍具有发现慈鲷新物种的潜力[5]。美国食品和药物管理局(FDA) 于2011年正式采用DNA条形码技术作为鱼种鉴定的官方方法[6]。近年来,还有许多研究者应用DNA条形码技术对世界各地的鱼类进行鉴定,都证明该技术不仅能够对已知鱼种进行鉴定,而且还能够发现新鱼种,可以广泛适用于鱼类物种鉴别[7-9]。我国的DNA条形码技术近年来也取得一些进展。王中铎等用COI基因对南海硬骨鱼类40个物种89个样本进行了分析,结果表明COI序列广泛适用硬骨鱼类物种鉴别,并可用于低级分类阶元的系统进化分析[10]。程磊等研究了鲫属Carassius某些种和亚种中的COI基因变异,认为欧亚大陆与日本列岛的鲫鱼C.auratus有明显分化。银鲫C.auratus gibelio应被视为鲫的一个亚种,而不是一个有效物种[11]。陈双雅等对台湾海峡常见的8种石斑鱼进行了16S rRNA基因和COI基因的序列测定,结果表明COI基因较16S rRNA基因有更大的分辨率,2个基因序列有差异,建议在进行鱼种鉴定时同时使用16S rRNA基因和COI基因[12]。王茜等报告了4种养殖鲤科鱼类的COI基因部分序列,认为COI基因不但可以作为物种辨识的良好DNA条码,而且在鲤科鱼类的种间系统发生方面也具有一定的适用性[13]。张馨月等针对部分拖网渔获物进行DNA条形码研究,认为COI基因序列可以作为鱼种鉴定的一种便捷有效方式,同时可以对鱼类形态学分类系统进行补充和修订[14]。Shen等研究了长江中游采集的35种鱼的COI条形码,证明DNA条形码技术是长江中游鲤科鱼类辨识的有效工具[15]。但目前针对观赏鱼基因条码的研究报告还很少。
本研究对慈鲷科Cichlidae的非洲珠宝鱼Hemichromis letourneuxi、接吻鱼Helostoma temminkii、珍珠毛足鲈Trichopodus leerii、蓝钻石孔雀Aulonocara stuartgranti、神仙鱼Pterophyllum scalare,花鳉科Poeciliidae的孔雀鱼Poecilia reticulata,鲤科Cyprinidae的锦鲤Cyprinus carpio,骨舌鱼科Osteoglossidae的银龙Osteoglossum bicirrhosum、珍珠龙Scleropages jardinii以及红尾金龙、黄尾龙鱼、青龙等亚洲龙鱼Scleropages formosus进行研究,探讨16S rRNA和COI基因在观赏鱼种类鉴定中的作用。
1. 材料与方法
1.1 试验材料
供试非洲珠宝鱼、接吻鱼、珍珠毛足鲈、蓝钻石孔雀、孔雀鱼、神仙鱼、锦鲤等观赏鱼购自厦门市观赏鱼市场,红尾金龙、黄尾龙鱼、青龙等亚洲龙鱼以及珍珠龙、银龙等由厦门市某观赏鱼公司提供。每种观赏鱼选择形态色彩一致的10条进行试验,其中青龙和珍珠龙样品数为5条。
1.2 试验仪器和试剂
Veriti PCR仪(美国ABI公司),台式冷冻离心机(美国Thermo公司),FlashGel闪胶系统(Lonza公司);动物组织基因组提取试剂盒(Tiangen公司)。
1.3 方法
1.3.1 DNA提取
用洁净解剖刀取鱼体背部肌肉约100 mg,按照试剂盒方法提取DNA。提取的DNA置于-20℃冰箱备用。
1.3.2 PCR引物
扩增观赏鱼16S rRNA的引物参考Palumbi的方法[16],上游引物序列为16Sar-5:5′-CGC CTG TTT ATC AAA AAC AT-3′,下游引物序列为16Sbr-3:5′-CCG GTC TGA ACT CAG ATC ACG T-3′。扩增片段长约590 bp;扩增COI的引物参考Ivanova的方法[17],上游引物序列为FF2d:5′-TTC TCC ACC AAC CAC AAR GAY ATY GG-3′,下游引物序列为FR1d:5′-CAC CTC AGG GTG TCC GAA RAA YCA RAA-3′。扩增片段长约680 bp。
1.3.3 PCR扩增
采用50 μL反应体系,即在PCR反应管中加入17.5 μL超纯水,5 μL模板DNA,25 μL 2×Taq PCR MasterMix (包含Taq DNA聚合酶、dNTPs、MgCl2、反应缓冲液、PCR反应的增强剂和优化剂以及稳定剂),引物(20 μmol·L-1) 各1.25 μL。反应程序为:先95℃预变性1 min,然后95℃ 15 s、52℃(扩增COI基因) 或54℃(扩增16S rRNA基因)15 s、72℃ 30 s共35个循环,最后72℃延伸7 min,4℃保存。
1.3.4 电泳
为确认扩增效果,将扩增的产物进行电泳。采用LONZA闪胶系统(FlashGelTM DNA System),在预制好的含有缓冲液的琼脂凝胶,加入样品和标记(Marker),观察结果。
1.3.5 PCR扩增产物的序列测定
所有观赏鱼COI基因和16S rRNA基因扩增产物纯化后送美吉生物有限公司测序。
1.3.6 DNA序列分析
利用Chromas软件和CLUSTAL X软件对COI基因和16S rRNA基因测序结果进行拼接和分析。应用MEGA软件[18],选择Kimura-2参数模型制作系统发育树,计算遗传距离。系统分析采用邻接法(neighbor-joining,NJ)。发育树采用重复抽样分析(Bootstrap)1 000次检验各分枝的置信度。
1.3.7 数据库比对
将测序所得到的16S rRNA基因和COI基因序列在GenBank和BOLD SYSTEMS数据库中搜索相似序列,进行鉴定结果分析。
2. 结果与分析
2.1 12种观赏鱼16S rRNA基因和COI基因序列测定结果
12种观赏鱼的16S rRNA基因和COI基因序列测定结果表明,绝大多数种类的种内基因序列差异很小。16S rRNA基因基本相同,而COI基因序列差异稍大,其中非洲珠宝鱼、蓝钻石孔雀、珍珠毛足鲈、锦鲤、接吻鱼、孔雀鱼、珍珠龙和银龙的种内序列差异不超过2个碱基,神仙鱼、红尾金龙、黄尾龙鱼和青龙的种内差异稍大,但也不超过4个碱基。上述观赏鱼的16S rRNA基因和COI基因,选择同种比较一致的序列提交GenBank,其收录序列号见表 1。
表 1 12种观赏鱼基因GenBank登录号Table 1. Genbank accessions on 12 ornamental fish species观赏鱼名称 GenBank登录号 16S rRNA基因 COI基因 非洲珠宝鱼H. letourneuxi KX816035 KX832882 蓝钻石孔雀A. stuartgranti KX816036 KX832884 接吻鱼H. temminkii KX816037 KX832883 孔雀鱼P. reticulata KX816038 KX832885 珍珠毛足鲈T. leerii KX816039 KX832886 神仙鱼P. scalare KX816040 KX832887 锦鲤C.carpio KX866416 KX832888 红尾金龙S. formosus(red-tail golden arowana) KX816041 KX832889 青龙S. formosus (green arowana) KX816042 KX832891 黄尾龙鱼S. formosus(yellow-tail arowana) KX816043 KX832890 珍珠龙S. jardinii KX816044 KX832892 银龙O. bicirrhosum KX816045 KX832893 2.2 12种观赏鱼16S rRNA基因和COI基因系统发育分析
2.2.1 12种观赏鱼的系统发育树
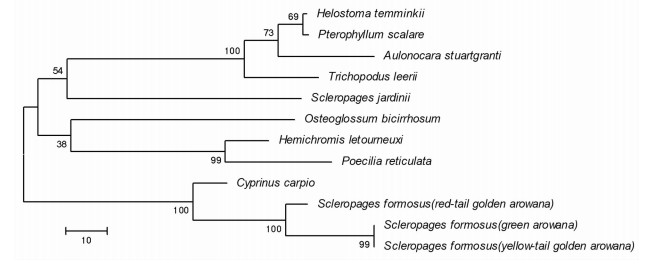
16S rRNA基因的系统发育树(图 1) 显示,3种亚洲龙鱼和锦鲤聚成一支,黄尾龙鱼和青龙鱼的关系最近;接吻鱼与神仙鱼关系最近,并与蓝钻石孔雀、珍珠毛足鲈以及珍珠龙鱼等聚成一支;非洲珠宝鱼与孔雀鱼关系较近,并与银龙聚成一支。
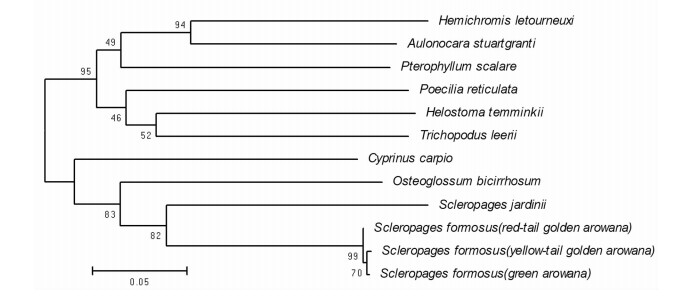
COI基因的系统发育树(图 2) 显示,3个品种的亚洲龙鱼聚成一支,黄尾龙鱼和青龙鱼的关系最近,再依次与珍珠龙鱼、银龙和锦鲤聚在一起;接吻鱼与神仙鱼关系最近,并与蓝钻石孔雀、珍珠毛足鲈以及珍珠龙鱼等聚成一支;非洲珠宝鱼与孔雀鱼关系较近,并与银龙聚成一支;非洲珠宝鱼与蓝钻石孔雀亲缘关系较近,再与神仙鱼聚在一起;接吻鱼和珍珠毛足鲈亲缘关系较近,然后与花鳉科的孔雀鱼聚成一支。再与另外3种慈鲷鱼类聚成一支。
2.2.2 12种观赏鱼的遗传距离分析
12种观赏鱼的16S rRNA序列具有明显差别。以锦鲤的595 bp基因为参照,其他观赏鱼类对应的基因片段长分别为:珍珠毛足鲈591 bp、非洲珠宝鱼583 bp、接吻鱼591 bp、蓝钻石孔雀593 bp、孔雀鱼583 bp、神仙鱼591 bp、七彩神仙592 bp、红尾金龙613 bp、黄尾龙鱼612 bp、青龙612 bp、珍珠龙607 bp、银龙595 bp。根据16S rRNA序列比对结果,除了青龙与黄尾龙鱼的遗传距离只有0.002以外,其他各种观赏鱼之间的16S rRNA遗传距离都很大。
12种观赏鱼的COI基因遗传距离见表 2。红尾金龙、黄尾龙鱼和青龙3种亚洲龙鱼之间的COI基因遗传距离只有0.003,实际上这3种亚洲龙鱼的COI基因只相差1个碱基。其他观赏鱼之间的遗传距离都比较大,可以进行有效的鉴别。
表 2 12种观赏鱼COI基因的遗传距离Table 2. Genetic distances of COI gene from 12 ornamental fish species观赏鱼名称 1 2 3 4 5 6 7 8 9 10 11 1.Hemichromis_letourneuxi 2.Helostoma_temminkii 0.396 3.Aulonocara_stuartgranti 0.234 0.353 4.Poecilia_reticulata 0.330 0.311 0.334 5.Trichopodus leerii 0.299 0.270 0.300 0.289 6.Pterophyllum scalare 0.312 0.290 0.281 0.317 0.352 7. Cyprinus_carpio 0.363 0.334 0.349 0.362 0.392 0.335 8.红尾金龙 0.393 0.363 0.332 0.362 0.329 0.344 0.322 9.黄尾龙鱼 0.399 0.366 0.335 0.365 0.335 0.350 0.325 0.003 10.青龙鱼 0.401 0.366 0.339 0.365 0.328 0.347 0.325 0.003 0.003 11.珍珠龙鱼 0.379 0.395 0.401 0.400 0.387 0.400 0.345 0.243 0.248 0.242 12.银龙鱼 0.379 0.369 0.367 0.370 0.357 0.387 0.298 0.276 0.281 0.282 0.290 2.3 应用16S rRNA基因和COI基因鉴定观赏鱼
将本研究得到的12种观赏鱼的16S rRNA基因和COI基因测序结果在GenBank进行nucleotide BLAST,搜索同源性比较结果见表 3。结果表明,接吻鱼、珍珠毛足鲈、神仙鱼、红尾金龙、青龙、黄尾龙鱼、珍珠龙和银龙的基因序列与数据库中相应的序列同源性大于99%,因此都可以通过16S rRNA基因和COI基因进行鉴别。但红尾金龙、青龙、黄尾龙鱼等亚洲龙鱼的16S rRNA基因和COI基因同源性均大于99.8%,虽然可以鉴定为亚洲龙鱼,但无法再鉴别不同的品种。非洲珠宝鱼、蓝钻石孔雀、孔雀鱼等观赏鱼的基因序列与数据库中的多种观赏鱼类的相应序列同源性均大于99%。其中蓝钻石孔雀与蓝钻石孔雀、乔氏高身丽鱼Alticorpus geoffreyi、斑马拟丽鱼Maylandia zebra、马拉维湖颊丽鲷Genyochromis mento、黑岩非鲫Petrotilapia nigra、犬齿非鲫Cynotilapia afra、白斑若丽鱼Pseudotropheus crabro等多种不同属观赏鱼的序列同源性均超过99%,显然无法通过16S rRNA基因和COI基因进行鉴别;非洲珠宝鱼与同属的双斑伴丽鱼H. bimaculatus、点纹伴丽鱼H. guttatus和撒哈拉伴丽鱼Hemichromis saharae同源性超过99%,因此也无法通过16S rRNA基因和COI基因进行种类鉴别。此外,从同源性比较可知,相比较而言,16S rRNA基因比COI基因相应的同源性更高。
表 3 16S rRNA基因和COI基因与数据库相关序列比对结果Table 3. Sequence alignments on 16S rRNA and COI gene with database观赏鱼名称 GenBank搜索结果 BoldSystems搜索结果 16S rRNA基因 COI基因 COI基因 非洲珠宝鱼H. letourneuxi H. letourneuxi (99%);H. bimaculatus(99%);H. guttatus(99%) H. letourneuxi(100%);H. saharae (99%);H. guttatus (99%) H. letourneuxi(100%);Tilapia guineensis(99.38%);H. bimaculatus(99.38%);H. saharae (99.38%) 蓝钻石孔雀A. stuartgranti A. stuartgranti (100%);Alticorpus geoffreyi(100%);Maylandia zebra(99%);Genyochromis mento(99%);Petrotilapia nigra(99%);Cynotilapia afra(99%);Pseudotropheus crabro(99%) A. stuartgranti(100%);A. geoffreyi(100%);M. zebra(99%);G. mento(99%);P. nigra(99%);C. afra(99%);P. crabro(99%) A. stuartgranti(100%);Metriaclima greyshaki(100%);Pseudotropheus socolofi(100%);Pseudotropheus demasoni(100%);Melano-chromis johannii(100%);Pseudotropheus elongatus(100%);Placidochromis milomo(100%);Maylandia lombardoi(100%);Maylandia hajomaylandi(100%);Maylandia callainos(100%);Labidochromis chisumulae(100%);P. crabro(100%) 接吻鱼H.temminkii H. temminkii (99%) H. temminkii(99%) H. temminkii(100%) 孔雀鱼P. reticulate P. reticulata(99%) P. reticulate(100%);P. wingei(100%) P. reticulata(100%) 珍珠毛足鲈T. leerii T. leerii(100%) T. leerii(99%) T. leerii(99.32%) 神仙鱼P. scalare P. scalare(100%) P. scalare(99%) P. scalare(99.85%) 红尾金龙S. formosus S. formosus(99%) S. formosus(100%) S. formosus(100%) 黄尾龙鱼S. formosus S. formosus(100%) S. formosus(99%) S. formosus(100%) 青龙S. formosus S. formosus (99%) S. formosus(99%) S. formosus(100%) 珍珠龙S. jardinii S. jardinii(100%) S. jardinii(99%) S. jardinii(99.85%) 银龙O. bicirrhosum O. bicirrhosum(100%) O. bicirrhosum(100%) O. bicirrhosum(100%) 3. 讨论与结论
3.1 基因技术在观赏鱼分类中的作用
全球现有约1 800多种观赏鱼,其中约800种为淡水鱼。虽然世界上绝大多数淡水观赏鱼,最初都来自非洲和南美洲,但目前用于贸易的观赏鱼主要来自东南亚,而且80%以上为人工繁育的后代,仅约15%的海水观赏鱼及5%的淡水观赏鱼取自大自然。因此,在我国观赏鱼市场上,绝大部分观赏鱼都是人工繁育的后代。因鱼类在进化上表现出遗传特性的多样性和易变性,很容易出现杂交、变异等现象。同一种观赏鱼因其生长地域的不同,会造成体形、体色上的差别,主要表现在体色方面。人工培养的品种不仅在体色上,甚至在形态上都会发生较大的改变。因此根据外部形态特征对观赏鱼进行分类和鉴别已经被越来越多地证明不够准确,甚至是错误的。本试验结果也表明,通过测定COI基因等基因序列,不仅可以比较准确地鉴别各种形态相近的种类,而且可以明确各种观赏鱼之间的亲缘关系。
3.2 16S rRNA和COI基因在观赏鱼类分类中应用
线粒体基因结构简单,几乎不发生重组,进化速度快,不同区域进化速度存在差异,已成为基因条形码技术研究的最主要目标基因。BOLD已经登录了4 655 540条基因条码序列,其中在物种水平已经有173 232种动物的2 635 468条COI序列。由于16S rRNA基因进化速度相对较慢,在亲缘关系较近的观赏鱼分类中,不同属甚至不同科的观赏鱼的16S rRNA基因同源性常常超过98%,因此不太适合用于这些观赏鱼的分类。Craig等[19]用16S rRNA基因序列研究了42种南北美洲沿岸的石斑鱼系统进化关系,发现16S rRNA基因不能很好地解决属内分支问题,认为应该选取其他进化速度较快的基因。而COI基因的进化速度虽然比16S rRNA基因较快,但仍有一些属的观赏鱼类的COI基因同源性大于98%。BOLD SYSTEMS数据库推荐物种鉴定最小种间遗传距离为2%,因此COI基因作为许多观赏鱼类的基因条码存在明显的不足,需要选择进化速度更快的基因。实际上,COI条形码序列对种以上阶元的物种鉴定存在明显的局限性,针对分化程度低的物种,选择细胞色素b (cytb) 基因或控制区可能更为有效[14]。Mu等[20]对3个品种的亚洲龙鱼的线粒体12S rRNA、16S rRNA和COX3基因的遗传变异和亲缘关系进行了研究,结果表明3个线粒体基因序列呈现多态性,但不适合作为不同品种之间的遗传关系比较的标记。Mohd等[21]也认为亚洲龙鱼体色变化通常与COI或Cyt B基因分化程度没有关系,不能用来确定类群。本试验所用3个品种的亚洲龙鱼,其野生个体来自不同的水域,由于长期的地理隔离等因素,其形态和色彩具有显著的差异,但这些龙鱼的16S rRNA和COI基因差异很小。要鉴别这些龙鱼需要选择进化速度更快的基因深入研究。
此外,在基于16S rRNA和COI基因分别构建的系统发育树中,珍珠龙鱼与同属的亚洲龙鱼的亲缘关系显示出较大的差别,显然COI基因系统发育树与传统的鱼类分类系统更为接近。因此,在应用基因条码鉴定物种时,还需要结合传统的鱼类分类方法,才能得到更加准确的结果。
16S rRNA基因和COI基因作为基因条形码可以在物种水平对亚洲龙鱼、珍珠龙鱼、银龙鱼、神仙鱼、接吻鱼、孔雀鱼和珍珠毛足鲈等观赏鱼进行准确的鉴别。应用COI基因构建的系统发育树比16S rRNA基因更加接近现有的鱼类分类体系。但对形态十分相似的非洲珠宝鱼和蓝钻石孔雀等慈鲷类,以及红尾金龙、黄尾龙鱼、青龙鱼等同属于亚洲龙鱼的不同品系的观赏鱼,依靠16S rRNA基因或COI基因都无法进行准确鉴定,需要研究进化速度更快的DNA基因条码。
-
表 1 12种观赏鱼基因GenBank登录号
Table 1 Genbank accessions on 12 ornamental fish species
观赏鱼名称 GenBank登录号 16S rRNA基因 COI基因 非洲珠宝鱼H. letourneuxi KX816035 KX832882 蓝钻石孔雀A. stuartgranti KX816036 KX832884 接吻鱼H. temminkii KX816037 KX832883 孔雀鱼P. reticulata KX816038 KX832885 珍珠毛足鲈T. leerii KX816039 KX832886 神仙鱼P. scalare KX816040 KX832887 锦鲤C.carpio KX866416 KX832888 红尾金龙S. formosus(red-tail golden arowana) KX816041 KX832889 青龙S. formosus (green arowana) KX816042 KX832891 黄尾龙鱼S. formosus(yellow-tail arowana) KX816043 KX832890 珍珠龙S. jardinii KX816044 KX832892 银龙O. bicirrhosum KX816045 KX832893 表 2 12种观赏鱼COI基因的遗传距离
Table 2 Genetic distances of COI gene from 12 ornamental fish species
观赏鱼名称 1 2 3 4 5 6 7 8 9 10 11 1.Hemichromis_letourneuxi 2.Helostoma_temminkii 0.396 3.Aulonocara_stuartgranti 0.234 0.353 4.Poecilia_reticulata 0.330 0.311 0.334 5.Trichopodus leerii 0.299 0.270 0.300 0.289 6.Pterophyllum scalare 0.312 0.290 0.281 0.317 0.352 7. Cyprinus_carpio 0.363 0.334 0.349 0.362 0.392 0.335 8.红尾金龙 0.393 0.363 0.332 0.362 0.329 0.344 0.322 9.黄尾龙鱼 0.399 0.366 0.335 0.365 0.335 0.350 0.325 0.003 10.青龙鱼 0.401 0.366 0.339 0.365 0.328 0.347 0.325 0.003 0.003 11.珍珠龙鱼 0.379 0.395 0.401 0.400 0.387 0.400 0.345 0.243 0.248 0.242 12.银龙鱼 0.379 0.369 0.367 0.370 0.357 0.387 0.298 0.276 0.281 0.282 0.290 表 3 16S rRNA基因和COI基因与数据库相关序列比对结果
Table 3 Sequence alignments on 16S rRNA and COI gene with database
观赏鱼名称 GenBank搜索结果 BoldSystems搜索结果 16S rRNA基因 COI基因 COI基因 非洲珠宝鱼H. letourneuxi H. letourneuxi (99%);H. bimaculatus(99%);H. guttatus(99%) H. letourneuxi(100%);H. saharae (99%);H. guttatus (99%) H. letourneuxi(100%);Tilapia guineensis(99.38%);H. bimaculatus(99.38%);H. saharae (99.38%) 蓝钻石孔雀A. stuartgranti A. stuartgranti (100%);Alticorpus geoffreyi(100%);Maylandia zebra(99%);Genyochromis mento(99%);Petrotilapia nigra(99%);Cynotilapia afra(99%);Pseudotropheus crabro(99%) A. stuartgranti(100%);A. geoffreyi(100%);M. zebra(99%);G. mento(99%);P. nigra(99%);C. afra(99%);P. crabro(99%) A. stuartgranti(100%);Metriaclima greyshaki(100%);Pseudotropheus socolofi(100%);Pseudotropheus demasoni(100%);Melano-chromis johannii(100%);Pseudotropheus elongatus(100%);Placidochromis milomo(100%);Maylandia lombardoi(100%);Maylandia hajomaylandi(100%);Maylandia callainos(100%);Labidochromis chisumulae(100%);P. crabro(100%) 接吻鱼H.temminkii H. temminkii (99%) H. temminkii(99%) H. temminkii(100%) 孔雀鱼P. reticulate P. reticulata(99%) P. reticulate(100%);P. wingei(100%) P. reticulata(100%) 珍珠毛足鲈T. leerii T. leerii(100%) T. leerii(99%) T. leerii(99.32%) 神仙鱼P. scalare P. scalare(100%) P. scalare(99%) P. scalare(99.85%) 红尾金龙S. formosus S. formosus(99%) S. formosus(100%) S. formosus(100%) 黄尾龙鱼S. formosus S. formosus(100%) S. formosus(99%) S. formosus(100%) 青龙S. formosus S. formosus (99%) S. formosus(99%) S. formosus(100%) 珍珠龙S. jardinii S. jardinii(100%) S. jardinii(99%) S. jardinii(99.85%) 银龙O. bicirrhosum O. bicirrhosum(100%) O. bicirrhosum(100%) O. bicirrhosum(100%) -
[1] 赵增连, 陈华忠, 陈信忠.观赏鱼鉴赏与检疫指南[M].北京:中国质检出版社, 2014:3-16. [2] HEBERT P, CYWINSKA A, BALL S L, et al. Biological identifications through DNA barcodes[J]. Proc Biol Sci, 2003, 270(1512):313-321. DOI: 10.1098/rspb.2002.2218
[3] STEINKE D, ZEMLA K TS, HEBERT P D.Barcoding nemo:DNA-based identifications for the ornamental fish trade[J].P LoS One, 2009, 4(7):e6300. DOI: 10.1371/journal.pone.0006300
[4] KIM S, EO H S, KOO H, et al.DNA barcode-based molecular identification system for fish species[J].Mol Cells, 2010, 30(6):507-512. DOI: 10.1007/s10059-010-0148-2
[5] BREMAN F C, LOIX S, JORDAENS K, et al. Testing the potential of DNA barcoding in vertebrate radiations:the case of the littoral cichlids (Pisces, Perciformes, Cichlidae) from Lake Tanganyika[J]. Mol Ecol Resour, 2016, doi: 10.1111/1755-0998.12523.
[6] 陈双雅, 陈伟玲, 徐敦明.鱼类分子生物学物种鉴定技术[M].厦门:厦门大学出版社, 2012:1-20. [7] MABRAGAÑA E, DÍAZ D E ASTARLOA J M, et al.DNA barcode identifies Argentine fishes from marine and brackish waters[J].PLoS One, 2011, 6(12):e28655. DOI: 10.1371/journal.pone.0028655
[8] MAT JAAFAR T N, TAYLOR M I, MOHD NOR S A, et al.DNA barcoding reveals cryptic diversity within commercially exploited Indo-Malay Carangidae (Teleosteii:Perciformes)[J]. PLoS One, 2012, 7(11):e49623. DOI: 10.1371/journal.pone.0049623
[9] PEREIRA L H, HANNER R, FORESTI F, et al.Can DNA barcoding accurately discriminate megadiverse Neotropical freshwater fish fauna?[J].BMC Qenet, 2013, 14(1):20. https://www.researchgate.net/profile/Luiz_Pereira4/publication/236001515_Can_DNA_barcoding_accurately_discriminate_megadiverse_Neotropical_freshwater_fish_fauna/links/00b4951574912594c1000000.pdf?inViewer=true&disableCoverPage=true&origin=publication_detail
[10] 王中铎, 郭昱嵩, 陈荣玲.南海常见硬骨鱼类COI条码序列[J].海洋与湖沼, 2009, 40(5):608-614. http://www.cnki.com.cn/Article/CJFDTOTAL-HYFZ200905014.htm [11] 程磊, 常玉梅, 鲁翠云, 等.鲫属鱼类DNA条码及种与亚种划分[J].动物学研究, 2012, 33(5):463-472. http://www.cnki.com.cn/Article/CJFDTOTAL-DWXY201205010.htm [12] 陈双雅, 王嘉鹤, 陈伟玲, 等.16S rRNA基因和COI基因序列分析在石斑鱼物种鉴定中的应用[J].生物技术通报, 2012, (10):124-130. http://www.cnki.com.cn/Article/CJFDTOTAL-SWJT201210021.htm [13] 王茜, 金毅成.4种常见鲤科鱼类DNA条形码的研究[J].安徽农业科学, 2014, 42(27):9281-9282, 9316. http://www.cnki.com.cn/Article/CJFDTOTAL-AHNY201427008.htm [14] 张馨月, 刘岩, 张秀梅, 等.基于COI基因的西南大西洋部分经济鱼类DNA条形码鉴定[J].水生生物学报, 2014, 38(6):1161-1167. http://www.cnki.com.cn/Article/CJFDTOTAL-SSWX201406022.htm [15] SHEN Y, GUAN L, WANG D, et al. DNA barcoding and evaluation of genetic diversity in Cyprinidae fish in the midstream of the Yangtze River[J]. Ecol Evol, 2016, 6(9):2702-2713. DOI: 10.1002/ece3.2016.6.issue-9
[16] PALUMBI S R.Nucleic acids Ⅱ:the polymerase chain reaction.In Hillis DM, Moritz C, Mable BK (Eds.).Molecular Systematics. Massachusetts[M].Sunderland:Sinauer & Associates Inc, 1996:205-247.
[17] IVANOVA N V, ZEMLAK T S, HANNER R H, et al. Universal primer cocktails for fish DNA barcoding[J]. Mol Ecol Notes, 2007, 7(4):544-548. DOI: 10.1111/j.1471-8286.2007.01748.x
[18] KUMAR S, TAMURA K, JAKOBSEN I B, et al.MEGA2:molecular evolutionary genetics analysis software[J]. Bioinformatics, 2001, 17(12):1244-1245. DOI: 10.1093/bioinformatics/17.12.1244
[19] CRAIG M T, PONDALLA D J, FRANCK J P, et al. On the status of the Serranid fish genus Epinephelus:evidence for paraphyly based upon 16S rDNA sequence[J]. Mol Phylogenet Evol, 2001, 19(1):121-130. DOI: 10.1006/mpev.2000.0913
[20] MU X D, SONG H M, WANG X J, et al. Genetic variability of the Asian arowana, Scleropages formosus, based on mitochondrial DNA genes[J].Biochemical Systematics and Ecology, 2012, 44:141-148. DOI: 10.1016/j.bse.2012.04.017
[21] MOHD-SHAMSUDIN M I, FARD M Z, MATHER P B, et al. Molecular characterization of relatedness among colour variants of Asian Arowana (Scleropages formosus)[J]. Gene, 2011, 490(1-2):47-53. DOI: 10.1016/j.gene.2011.08.025




 下载:
下载: