Cloning and Expressions of CmCCR1 in Citrus maxima Sanhongmiyou
-
摘要:目的
肉桂酰辅酶A还原酶(cinnamoyl-CoA reductase,CCR)基因是木质素合成的关键基因,本研究旨在探明该基因在柚汁胞粒化过程中的作用机制。
方法通过生物信息学、基因克隆与亚细胞定位、荧光定量PCR技术,筛选克隆三红蜜柚CmCCR基因,及其在两个品种(三红蜜柚和六月早1号柚)不同发育时期果实汁胞中的表达情况,并利用乙酰溴法测定柚汁胞木质素含量。
结果从三红蜜柚汁胞转录组数据中筛选得到5个CmCCR基因,根据果实汁胞成熟期木质素含量与CmCCRs基因的转录水平变化的相关性,初步明确CmCCR1可能参与汁胞木质素代谢;克隆所得CmCCR1基因序列,其开放阅读框长度为
1017 bp;生物信息学分析发现,该基因编码的CmCCR1蛋白分子量为37.5 kDa,等电点(pI)7.06,属于稳定亲水蛋白,含有NAD(P)H/NAD(P)(+) 结合(NADB)域,与甜橙CcCCR1有3个相同的Motif,分布情况也大致相同;进化树分析结果表明,CmCCR1与甜橙CcCCR1的亲缘关系最近,且都归于双子叶植物分组;亚细胞定位结果表明CmCCR1定位于内质网(膜)上,与软件预测的结果相吻合;实时定量PCR结果显示CmCCR1基因的表达量中三红蜜柚汁胞成熟阶段显著上升,在六月早1号柚汁胞发育过程中的相对表达量一直处于较低水平,且显著低于同一发育阶段三红蜜柚汁胞的相对表达量;将CmCCR1瞬时转化至六月早1号柚汁胞,发现瞬时转化的汁胞中CmCCR1基因的表达量显著上升,间苯三酚染色程粉红色,说明汁胞中出现了木质素的积累。结论CmCCR1基因可能参与调控三红蜜柚汁胞木质素等次级代谢产物的生物合成,其表达量的上升与柚汁胞粒化的发生有关。
Abstract:ObjectiveFunctions and expressions of cinnamoyl-CoA reductase genes (CmCCRs) associated with the lignin biosynthesis and juice vesicle granulation in Citrus maxima were investigated.
MethodBioinformatics, gene cloning, subcellular localization, and fluorescence quantitative PCR were applied to select and clone CmCCRs from C. maxima Sanhongmiyou. Expressions of the genes in the juice sacs at different fruit development stages of Sanhongmiyou and Liuyuezaoyihao pomelos were examined. Lignin content in the sacs was determined using the acetyl bromide method.
ResultsFrom the RNA-Seq data on Sanhongmiyou juice sacs, 5 CmCCRs were identified. Since the lignin content in the mature juice sacs was shown to correlate with the gene transcription, CmCCR1 was supposed to be involved in the lignin formation, and thus further analyzed. The ORF of CmCCR1 was 1,017 bp long. The stable hydrophilic protein had a molecular weight of 37.5 kDa and a pI of 7.06 containing an NAD(P)H/NAD(P)(+) binding (NADB) domain and three shared common motifs of CcCCR1 in sweet orange with similar distribution patterns. Phylogenetically, it closely related to CcCCR1 and belonged to the dicotyledonous plants. The gene was located on the endoplasmic reticulum same as predicted previously by software. The expression of CmCCR1 in the Sanhongmiyou juice sacs increased significantly at maturation, but that of Liuyuezaoyihao remained relatively low throughout the entire developmental stages at significantly lower levels. However, when the Liuyuezaoyihao pomelo juice sacs were transiently transformed with CmCCR1, a significantly elevated expression of the gene resulted. Moreover, the phloroglucinol-stained sacs turned pink in color indicating lignin accumulation in them.
ConclusionCmCCR1 was postulated to regulate the biosynthesis of secondary metabolites (e.g., lignin) and the granulation of juice vesicles in pomelos.
-
Keywords:
- Citrus maxima /
- cinnamoyl-CoA reductase /
- gene cloning /
- expression analysis
-
0. 引言
【研究意义】柚[Citrus maxima (Burm.) Merr.]为芸香科柑橘属植物,在我国长江以南及东南亚各国广泛种植。三红蜜柚丰产质优,果大皮薄,汁多可口,酸甜适中,内含丰富的维生素C与类黄酮物质,是公认的具有良好食疗效益的保健型水果[1]。但汁胞粒化现象在柚类果实的成熟与贮藏过程中普遍存在,是柑橘类果实中一种较为常见的生理性病害[2, 3],其粒化进程与木质素的积累密切相关[4]。因此,降低木质素含量,能够有效减轻柚果实的汁胞粒化现象,进而提高其商品价值。【前人研究进展】木质素(1ignin)是一类包围在维管束细胞和厚壁细胞壁外的芳香高聚物,其含量仅次于纤维素,并与纤维素和半纤维素共同构成植物体的基本骨架。木质素在植物生长发育过程中有着不可替代的作用,具有支持加固、增强细胞壁机械强度、提高疏导组织运输水分能力、抵抗外界不良环境及病菌微生物侵袭等多种功能[5−8]。木质素在植物体内的合成途径大致可分为莽草酸途径、苯丙氨酸途径及木质素合成途径3种类型,其中起主要作用的是木质素合成途径[9]。在木质素合成的过程中需要众多酶的参与和共同作用,肉桂酰辅酶A还原酶(cinnamoyl-CoA reductase, CCR)是木质素特异途径中的第一个关键酶[10],它在NADPH的辅助下,催化硫酯肉桂酰辅酶A衍生物还原成相应的肉桂醛[11]。研究表明,CCR可能对木质素生物合成途径的碳流具有潜在的调控作用,是由碳素流向木质素的关键节点[12]。CCR基因首先在桉树(Eucalyptus urophylla)[12]中被鉴定出来,之后从拟南芥(Arabidopsis thaliana)、小麦(Triticum aestivum)、番茄(Solanum lycopersicum)等多种植物中克隆得到了CCR基因的全长或部分编码序列[13]。在转基因杨树(Populus L.)中和拟南芥中,CCR表达量的下降最终可导致木质素含量降低[14, 15]。Chen等[16]通过下调紫花苜蓿(Medicago sativa)中CCR基因的表达,发现木质素含量同样会减少。在挪威云杉(Picea abies)中,抑制CCR的表达也可以降低木质素含量。缺硼胁迫下,香橙(Citrus junos )和枳橙(Citrus × insitorum)的CCR基因表达量均有不同程度的上调表达,木质素含量增加[17]。由此可见,对于大多数植物而言,通过调节CCR基因的表达量可以使得其木质素的结构、组分及含量发生相应的变化。因此,CCR基因被认为是调控木质素合成途径进而提高植物品质的有效途径之一[18]。【本研究切入点】在前期的研究中,我们发现柚汁胞粒化现象与木质素代谢紧密相关。CCR作为木质素特异代谢途径的第一个关键酶,其编码基因成员及其在果实发育过程中的表达规律还未见报道。【拟解决的关键问题】本研究拟根据琯溪蜜柚汁胞RNA-Seq数据获得的CmCCR1基因全长,探究该基因在汁胞发育过程中的表达规律,旨在进一步探究柚果实发育过程中CCR基因在木质素代谢途径中的作用,为解决汁胞粒化问题、改善柚果实品质提供依据。
1. 材料与方法
1.1 试验材料
本试验以易粒化品种三红蜜柚和不粒化品种六月早1号果实为材料,两个品种所用的果实均采集于福建农林大学园艺产品贮运保鲜研究所基地(117°14′5″E,24°13′58″N)。两个品种各取3株树龄(10 a左右)、树势和栽培管理条件基本一致的果树,每棵果树采集3个发育正常、均匀一致的果实,于完熟采收前60 d(DBH 60)开始取样,每隔10 d取1次样(三红蜜柚和六月早1号柚均以达到该品种完全成熟状态时采收,采收当天记为DBH0),并于采集当天对试验样品进行处理。每个品种设置3个生物学重复,将同一株树的3个果实汁胞取下混合均匀后标记分装,于液氮中速冻,储存于−80 ℃超低温冰箱中,用于后续的相关试验。
1.2 试验方法
1.2.1 转录组数据分析
从本实验室获得的暂未发表的三红蜜柚转录组数据中筛选得到CmCCRs基因的FPKM值和编码区序列,利用GraphPad Prism 9.5软件绘制热图,分析各个基因的表达丰度,筛选表达水平高、变化趋势与木质素含量变化显著相关的基因用于后续试验。
1.2.2 总RNA提取及cDNA合成
参照TaKaRa Trizol RNA提取试剂盒说明书提取三红蜜柚果实汁胞总RNA。参照全式金Easyscript® One-step gDNA Removal and cDNA Synthesis SuperMix试剂盒以及TaKaRa Perfect Real Time试剂盒提供的说明书逆转录合成cDNA,分别用于CmCCR基因的克隆和qRT-PCR试验。
1.2.3 CmCCR1基因的克隆及测序
参照全式金TransTaq® DNA Polymerase High Fidelity试剂盒说明书,以三红蜜柚cDNA为模板扩增CmCCR1基因序列。PCR反应体系包括:1 μL cDNA,0.5 μL 上下游引物,2 μL 2.5 mmol·L−1 dNTPs ,0.5 μL TransTaq® HiFi DNA Polymerase,2.5 μL TransTaq® HiFi Buffer Ⅱ(10×),以及18 μL ddH2O。PCR扩增程序如下:95 ℃预变性3 min;95 ℃变性60 s,56 ℃退火45 s,72 ℃延伸60 s,35个循环;72 ℃延伸3 min。所用引物序列见表1。参照全式金Gel/PCR Extraction Kit试剂盒的方法回收目的片段,克隆、转化感受态细胞,将PCR鉴定为阳性的克隆送至上海生工生物工程股份有限公司测序。
表 1 CmCCR基因ORF引物序列Table 1. Details of CmCCR ORF primers引物
Primers引物序列
Primer sequencesCmCCR1-Xba I-F GCTCTAGAATGACGGTAATTGATAGC CmCCR1-Kpn I-R GGGGTACCGAATTTTCACTGATTC 1.2.4 CmCCR1基因及其编码蛋白的生物信息学分析
利用ProtParam(https://web.expasy.org/protparam/)预测蛋白基本理化性质;利用TMHMM - 2.0(https://services.healthtech.dtu.dk/services/TMHMM-2.0/)进行跨膜结构预测;利用NetPhos - 3.1(https://services.healthtech.dtu.dk/services/NetPhos-3.1/)预测磷酸化位点;利用BUSCA(http://busca.biocomp.unibo.it)进行亚细胞定位预测;利用MEME(http://meme-suite.org/)预测及分析Motif。采用MEGA7.0.26最大似然估计法(maximum likelihood method)构建系统发育树。
1.2.5 CmCCR1基因亚细胞定位
将2 μL重组质粒p-super1300-CCR-GFP和2 μL对照质粒p-super-
1300 -GFP加入含有30 μL农杆菌感受态细胞的1.5 mL离心管中,轻轻混匀,冰浴10 min,液氮中速冻5 min,然后快速将离心管置于37 ℃水浴中保持5 min,再冰浴5 min,加入800 μL无抗生素的YEB培养基,28 ℃振荡培养4 h,取500 μL菌液涂布于含50 mg·L−1 Kana和50 mg·L−1 Rif 的YEB固体培养基上,28 ℃倒置培养48 h。挑取阳性克隆,28 ℃、200 r·min−1振荡培养至OD600为0.8~1.0。挑选正常生长4周左右的本氏烟草(Nicotiana benthamiana),用去掉针头的注射器,将菌液注射到烟草叶背至出现水渍状斑块。将注射好的烟草植株暗培养1 d、正常培养1 d后,于激光共聚焦显微镜下进行镜检。
1.2.6 CmCCR1基因实时荧光定量表达分析
引物设计软件为Beacon Designer 8.0,以甜橙(Citrus sinensis)Actin(XM_025093446)为内参基因,引物序列详见表2。荧光定量PCR仪为耶拿qTOWER 2.0。qRT-PCR反应体系如下: 12.5 μL 荧光染料, 2 μL cDNA, 8.5 μL ddH2O,2 μL上下游引物。qRT-PCR扩增程序如下:95 ℃预变性40 s;95 ℃变性20 s,58 ℃退火35 s,72 ℃延伸40 s,共40个循环。采用2−ΔΔCt法计算CmCCR1基因相对表达量[19],采用GraphPad Prism 9.5进行数据统计和差异显著性分析。每组试验均进行3次生物重复,3次技术重复。
表 2 qRT-PCR所用引物信息Table 2. qRT-PCR primers applied引物名称
Primer name引物序列
Primer sequencesq-CmCCR1 F: TTGATGAATCTTGCTGGAGTG R: GTCTTTGCCGAGCCATTTA Actin F: AGAACTATGAACTGCCTGATGGC R: GCTTGGAGCAAGTGCTGTGATT 1.2.7 CmCCR1在汁胞中的瞬时表达
在超净工作台中取50 μL金粉悬液至1.5 mL离心管,依次加入5 μL已构建好的p-CAMBIA1301-CmCCR1质粒(1 μg·μL−1),50 μL 2.5 mol·L−1的CaCl2和20 μL 0.1 mol·L−1亚精胺,振荡2~3 s;涡旋1 min,冰浴1 min,将离心管放置冰上30 min,
10000 r·min−1离心20 s,弃上清;用250 μL无水乙醇充分吹洗沉淀,再用60 μL无水乙醇重悬沉淀,得到金粉包裹的质粒。在载体膜中央滴8~10 μL携带金粉的质粒,待其风干后装上阻拦网并旋紧,以p-CAMBIA1301为对照组。将六月早1号柚汁胞放置在基因枪下,汁胞和阻挡网之间距离设置为9 cm,真空度设定在25~27,氦气压力设为1100 psi。轰击后的汁胞在25 ℃下暗培养12 h,正常培养24 h后用GUS染色确认瞬时转化效果。将瞬时转化p-CAMBIA1301-CmCCR1的汁胞横向切0.5 mm左右的薄片,用1%间苯三酚染色1~2 min 后,加入1~2滴浓盐酸,显色后迅速置于Leica普通生物显微镜观察、拍照。1.2.8 果实汁胞木质素含量的测定
参照Fukushima的方法[20]测定汁胞木质素含量。称取50 mg烘干的汁胞粉末置于15 mL离心管中,用5 mL去离子水洗涤3次后,再用5 mL 95%乙醇清洗3次,然后用5 mL丙酮和5 mL乙醚清洗3次,弃上清。将沉淀烘干后,加入2 mL 25%乙酰溴,在70 ℃水浴锅中加热30 min,冷却后加入1 mL 2 mol·L−1的NaOH(含0. 75 mol·L−1盐酸羟胺)溶液,测定OD值(280 nm),重复3次。
1.2.9 数据分析
采用GraphPad Prism 9.5对数据进行整理、分析和作图。用t检验进行差异显著性分析,用Pearson相关性检验进行相关性分析。*表示0.01<Ρ<0.05,**表示Ρ<0.01,Ρ<0.05具有统计学意义。
2. 结果与分析
2.1 三红蜜柚果实汁胞成熟期木质素含量与CmCCRs基因的转录水平变化
在三红蜜柚果实成熟期间,汁胞木质素的含量随着成熟度的增加而上升,果实成熟阶段的汁胞中,木质素含量显著高于其他时期(图1A),说明在汁胞发育的后期,木质素的不断合成积累导致了汁胞出现粒化现象。从三红蜜柚转录组数据中筛选得到5个注释为肉桂酰辅酶A还原酶基因(CCR)的成员。根据其FPKM值可以看出,这5个成员中,ID为Cg1g021310的表达水平随着汁胞的成熟显著上升,其表达规律与木质素的积累成正相关性,而其他4个成员的表达规律与木质素积累无相关性,表明Cg1g021310可能与木质素的合成积累有关(图1 B、C)。
![]() 图 1 三红蜜柚果实汁胞成熟期木质素含量与CmCCRs基因的转录水平变化A:汁胞成熟期间木质素含量变化,B:转录组数据中的5个CCRs基因在汁胞成熟期间的表达热图,C:木质素含量和5个CCRs基因表达水平的相关性。**表示Ρ<0.01,同图10。Figure 1. Changes on lignin content and CmCCR transcript of Sanhongmiyou juice sacs during maturationA: changes on lignin content in juice sacs during maturation; B: transcriptome-based expression heatmap on 5 CCRs in juice sacs during maturation; C: correlation between lignin content and expressions of 5 CCRs.** indicates P<0.01, same for Fig.10.
图 1 三红蜜柚果实汁胞成熟期木质素含量与CmCCRs基因的转录水平变化A:汁胞成熟期间木质素含量变化,B:转录组数据中的5个CCRs基因在汁胞成熟期间的表达热图,C:木质素含量和5个CCRs基因表达水平的相关性。**表示Ρ<0.01,同图10。Figure 1. Changes on lignin content and CmCCR transcript of Sanhongmiyou juice sacs during maturationA: changes on lignin content in juice sacs during maturation; B: transcriptome-based expression heatmap on 5 CCRs in juice sacs during maturation; C: correlation between lignin content and expressions of 5 CCRs.** indicates P<0.01, same for Fig.10.2.2 CmCCR1基因克隆及序列分析
根据三红蜜柚转录组数据中得到Cg1g021310基因开放阅读框(Open Reading Frame,ORF)序列设计引物进行PCR扩增,获得大小为
1017 bp的产物(图2),测序结果表明其序列与从转录组数据中获得的序列一致。Blastn对比结果表明,克隆得到的基因序列与克里曼丁橘(Citrus clementina)和甜橙的CCR1基因序列相似度均为100%,因此将该序列命名为CmCCR1基因,该基因序列编码338个氨基酸,起始密码子为ATG,终止密码子为TAA。2.3 CmCCR1生物信息学分析
2.3.1 CmCCR1蛋白质基本理化性质分析
软件预测结果表明,CmCCR1分子式为C
1675 H2645 N445O501S13,蛋白分子量为37.5 kDa,等电点(pI)7.06,带正电荷的残基(Arg + Lys)和带负电荷的残基(Asp + Glu)均为40,属于稳定亲水蛋白。CmCCR1蛋白中含量最高的氨基酸为缬氨酸(Val)含量为9.8%,其次是赖氨酸(Lys)和丙氨酸(Ala),含量分别为8.3%和8.0%。2.3.2 CmCCR1蛋白的跨膜信号和亚细胞定位预测
TMpred预测结果表明CmCCR1蛋白有多个跨膜区域,分值均超过500,说明是跨膜螺旋(图3)。磷酸化修饰位点预测分析结果表明,CmCCR1含有50个磷酸化修饰位点,主要发生在丝氨酸、L-苏氨酸和酪氨酸位点上。
亚细胞定位预测结果显示CmCCR1蛋白定位于内质网(膜)分为0.685,质膜分为0.640分,定位于高尔基体分0.460,综合以上预测结果,可以推断CmCCR1定位于内质网(膜)的可能性较大。
2.3.3 CmCCR1的结构域预测
通过MEME预测比较了CmCCR1和CcCCR1结构域,结合CDD对CmCCR1结构域的分析,结果显示CmCCR1和CcCCR1均含有NAD(P)H/NAD(P)(+) 结合(NADB)域,且包含多个活性位点(图4),属于NADB_Rossmann超家族成员,具有肉桂酰辅酶A还原酶的功能,具备该家族成员的典型特征。Motif预测结果显示CmCCR1与CcCCR1具有3个位置相近的Motif(图5)。
2.3.4 CmCCR1系统进化关系结果
将CmCCR1氨基酸序列与甜橙、克里曼丁橘等物种的CCRs氨基酸序列构建系统进化树,结果表明,CmCCR1与CsCCR1和CcCCR1聚为一类,单子叶植物和双子叶植物的CCR蛋白可分为两大类,双子叶植物中的CCR蛋白可分为CCR1和CCR2两类(图6)。
2.4 CmCCR1基因的亚细胞定位
本试验将构建好的重组载体p-super-1300-CmCCR-GFP和对照载体p-super-
1300 -GFP分别转化农杆菌GV3101,注射侵染烟草,暗培养1 d,正常培养1 d后,于激光共聚焦显微镜下镜检,发现对照载体的整个烟草下表皮细胞均被绿色荧光所覆盖,而p-super-1300 -CmCCR-GFP重组载体却只能在内质网附近看到绿色荧光(图7)。由此说明,三红蜜柚的CmCCR1基因定位于内质网(膜)上,与先前软件预测的结果相吻合。![]() 图 7 CmCCR1亚细胞定位A~C表示p-super1300-GFP在烟草叶片中的亚细胞定位图。其中A表示p-super1300-GFP在荧光下的状态;B表示烟草叶片细胞在明场下的状态;C表示叠加效果图。D~F表示p-super1300-CmCCR-GFP在烟草叶片中的亚细胞定位图。D表示p-super1300-CmCCR-GFP在荧光下的状态;E表示烟草叶片细胞在明场下的状态;F表示叠加效果图。Figure 7. Subcellular localization of CmCCR1 geneA–C show subcellular localizations of p-super1300-GFP; A represents state of p-super1300-GFP under fluorescence; B represents state of tobacco leaf cells under bright field; C represents overlay effect image. D–F show subcellular localizations of p-super1300-CmCCR-GFP; D represents state of p-super1300-CmCCR-GFP; E represents state of tobacco leaf cells under bright field; F represents overlay effect image.
图 7 CmCCR1亚细胞定位A~C表示p-super1300-GFP在烟草叶片中的亚细胞定位图。其中A表示p-super1300-GFP在荧光下的状态;B表示烟草叶片细胞在明场下的状态;C表示叠加效果图。D~F表示p-super1300-CmCCR-GFP在烟草叶片中的亚细胞定位图。D表示p-super1300-CmCCR-GFP在荧光下的状态;E表示烟草叶片细胞在明场下的状态;F表示叠加效果图。Figure 7. Subcellular localization of CmCCR1 geneA–C show subcellular localizations of p-super1300-GFP; A represents state of p-super1300-GFP under fluorescence; B represents state of tobacco leaf cells under bright field; C represents overlay effect image. D–F show subcellular localizations of p-super1300-CmCCR-GFP; D represents state of p-super1300-CmCCR-GFP; E represents state of tobacco leaf cells under bright field; F represents overlay effect image.2.5 CmCCR1基因在不同品种果实发育期间中的表达差异
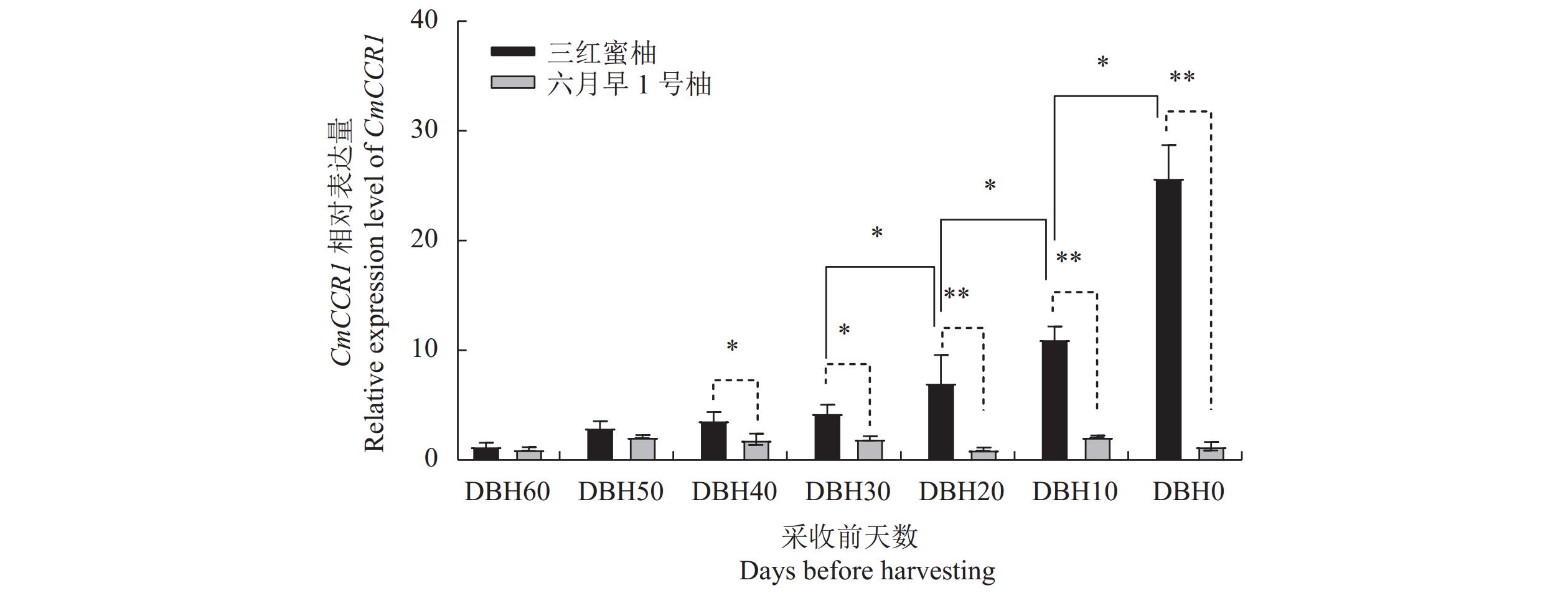
通过qRT-PCR分析了CmCCR1基因在三红蜜柚和六月早1号柚果实汁胞不同发育时期的相对表达量。结果表明(图8),CmCCR1基因的表达量随着三红蜜柚汁胞成熟度的增加而上升,在采收前30 d内,CmCCR1随着汁胞的发育其表达水平显著上升,在采收时达最高。CmCCR1基因在六月早1号柚各个时期汁胞中的相对表达量无显著差异,在采收前40 d之后显著低于同一发育阶段三红蜜柚汁胞的相对表达量,在采收前20 d差异极显著(P<0.01)。
2.6 CmCCR1基因在柚汁胞中的瞬时表达
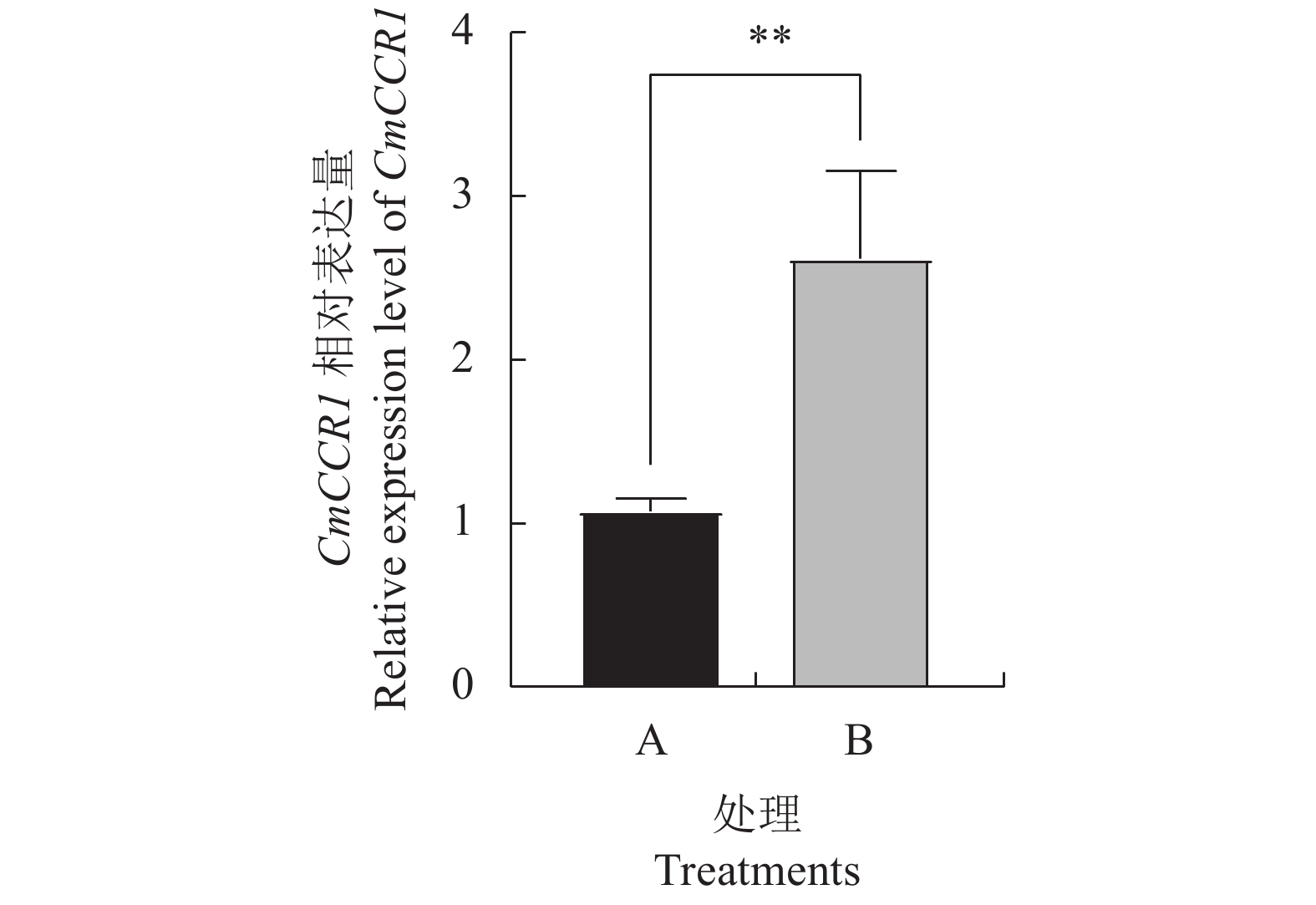
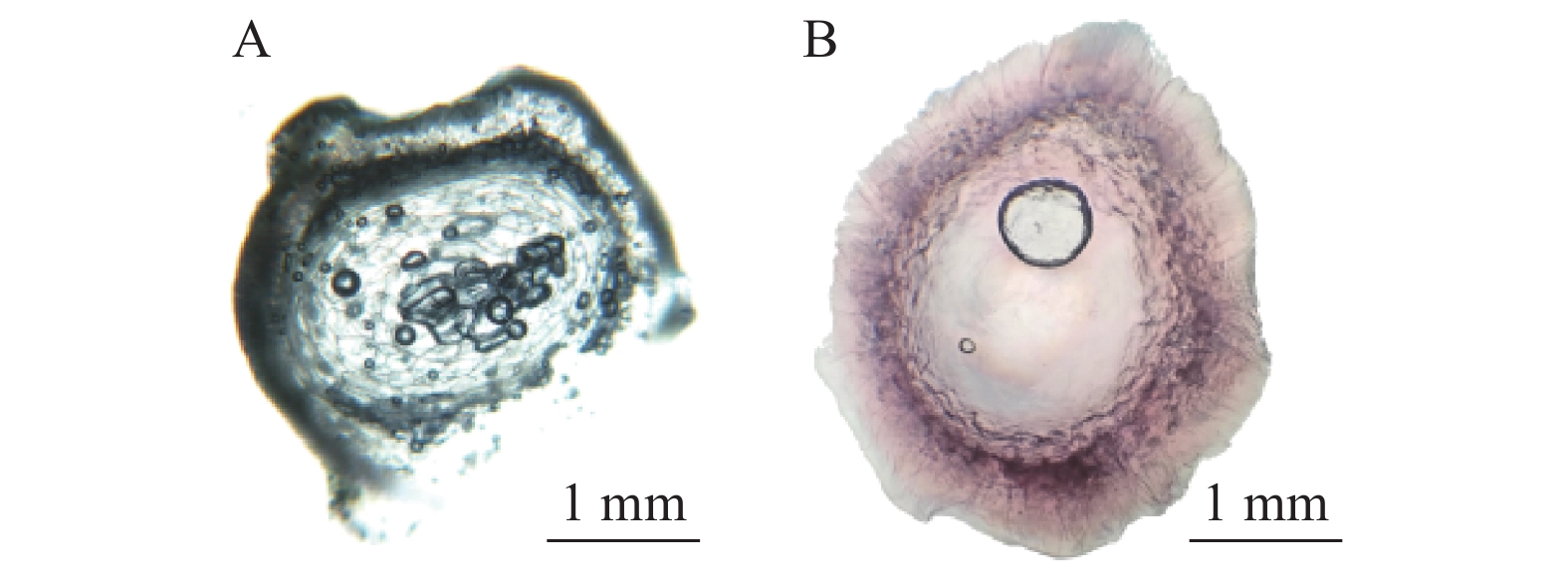
瞬时转化p-CAMBIA1301-CmCCR1质粒的六月早1号柚汁胞经GUS染色后呈蓝色(图9),RT-PCR结果显示,转化成功的汁胞中CmCCR1表达量显著高于对照组(图10),汁胞的横切面经间苯三酚染色后呈粉红色(图11),说明CmCCR1基因在六月早1号柚汁胞中表达后,导致了木质素的积累,表明CmCCR1基因是柚汁胞木质素合成途径中的关键酶。
![]() 图 11 间苯三酚染色的瞬转p-CAMBIA1301-CmCCR1六月早1号柚汁胞和正常汁胞A为对照组汁胞,未能被间苯三酚染色;B为试验组汁胞,间苯三酚染色后呈粉红色。Figure 11. Phloroglucinol-stained juice sacs of WT and transiently transformed p-CAMBIA1301-CmCCR1 LiuyuezaoyihaoA was control group not stainable by phloroglucinol; B, treatment group of pink juice sacs stained by phloroglucinol.
图 11 间苯三酚染色的瞬转p-CAMBIA1301-CmCCR1六月早1号柚汁胞和正常汁胞A为对照组汁胞,未能被间苯三酚染色;B为试验组汁胞,间苯三酚染色后呈粉红色。Figure 11. Phloroglucinol-stained juice sacs of WT and transiently transformed p-CAMBIA1301-CmCCR1 LiuyuezaoyihaoA was control group not stainable by phloroglucinol; B, treatment group of pink juice sacs stained by phloroglucinol.3. 讨论与结论
肉桂酰辅酶 A 还原酶(CCR)是木质素合成的关键酶之一,以香豆酰辅酶 A等5种肉桂酰辅酶A为底物,催化生成相应的肉桂醛,参与木质素的生物合成[21],是木质素特异合成途径中的第一个限速酶,并对碳流通过木质素生物合成途径具有潜在的调节作用[22]。CCR蛋白由CCRs基因家族编码,在拟南芥中和毛白杨(Populus tomentosa)中均有11个成员,在桉树中有10个成员[23~25],柚果实中的CCRs基因家族尚未见报道。本研究根据三红蜜柚转录组数据中得到5个CmCCRs成员,根据与果实中木质素含量的相关性筛选并克隆得到CmCCR1基因序列,该基因所编码的蛋白具有NAD(P)H/NAD(P)(+) 结合域,该结合域是CCRs的鉴别基序,高度保守[26]。植物CCR家族可分为双子叶植物和单子叶植物两大类[27],系统进化树分析发现CmCCR1与双子叶植物的CCR1的亲缘关系较近。已有研究表明,CCR1蛋白在木质素合成中起到关键作用[28],说明本研究克隆得到的CmCCR1编码的蛋白可能具有CCR1的功能,参与柚果实汁胞木质素的合成。
CCR基因的表达影响木质素的含量。Guojon等[15]发现抑制拟南芥AtCCR基因的表达减少了木质部中木质素的含量,CCR基因的突变体irx4的木质素含量降低了50%,影响植株的正常生长[29]。CCR基因的高表达导致香豆醛的积累,促进木质素积累[30]。CCR基因成员众多,根据其结构和功能可分为2类[31],不同成员的表达具有组织特异性,其中,仅有CCR1基因在所有木质化组织中表达[23, 32],而其他成员可能参与逆境胁迫的反应[33]。本研究从柚汁胞转录组数据中筛选得到5个CCRs基因成员,但仅有CmCCR1与汁胞发育过程中木质素含量的增加具有相关性,说明CmCCR1可能参与了汁胞木质素的合成,其他成员的作用还需要进一步确认。
CCR基因的表达与植物的生长发育过程中木质素的积累具有相关性。卢晓鹏[34]发现CCR基因在梨(Pyrus pyrifolia )果实中的表达与木质素含量变化趋势相同,在木质素含量高的品种中表达量显著高于木质素含量低的品种。在不同发育阶段辣椒(Capsicum annuum)中,CCR1基因的表达与木质素含量具有相关性,而且存在组织特异性,在茎中的表达量显著高于在叶片中的表达[27]。在本研究中,CmCCR1在三红蜜柚汁胞中的表达量随着果实的发育显著增加,木质素含量也伴随着增加;而在六月早1号柚汁胞发育过程中,CmCCR1的表达量无显著变化,而且没有测到木质素,但瞬时转化CmCCR1后,汁胞中的CmCCR1基因表达显著上升,且出现木质素的积累,因此,我们认为CmCCR1是三红蜜柚汁胞木质素的代谢的关键基因,可能参与木质素的积累。
综上,CmCCR1基因可能参与调控三红蜜柚汁胞木质素等次级代谢产物的生物合成。但对于CmCCR1基因的差异表达是否受到非生物胁迫以及启动子和相关转录因子的调控还需进一步的验证。
-
图 1 三红蜜柚果实汁胞成熟期木质素含量与CmCCRs基因的转录水平变化
A:汁胞成熟期间木质素含量变化,B:转录组数据中的5个CCRs基因在汁胞成熟期间的表达热图,C:木质素含量和5个CCRs基因表达水平的相关性。**表示Ρ<0.01,同图10。
Figure 1. Changes on lignin content and CmCCR transcript of Sanhongmiyou juice sacs during maturation
A: changes on lignin content in juice sacs during maturation; B: transcriptome-based expression heatmap on 5 CCRs in juice sacs during maturation; C: correlation between lignin content and expressions of 5 CCRs.** indicates P<0.01, same for Fig.10.
图 7 CmCCR1亚细胞定位
A~C表示p-super1300-GFP在烟草叶片中的亚细胞定位图。其中A表示p-super1300-GFP在荧光下的状态;B表示烟草叶片细胞在明场下的状态;C表示叠加效果图。D~F表示p-super1300-CmCCR-GFP在烟草叶片中的亚细胞定位图。D表示p-super1300-CmCCR-GFP在荧光下的状态;E表示烟草叶片细胞在明场下的状态;F表示叠加效果图。
Figure 7. Subcellular localization of CmCCR1 gene
A–C show subcellular localizations of p-super1300-GFP; A represents state of p-super1300-GFP under fluorescence; B represents state of tobacco leaf cells under bright field; C represents overlay effect image. D–F show subcellular localizations of p-super1300-CmCCR-GFP; D represents state of p-super1300-CmCCR-GFP; E represents state of tobacco leaf cells under bright field; F represents overlay effect image.
图 11 间苯三酚染色的瞬转p-CAMBIA1301-CmCCR1六月早1号柚汁胞和正常汁胞
A为对照组汁胞,未能被间苯三酚染色;B为试验组汁胞,间苯三酚染色后呈粉红色。
Figure 11. Phloroglucinol-stained juice sacs of WT and transiently transformed p-CAMBIA1301-CmCCR1 Liuyuezaoyihao
A was control group not stainable by phloroglucinol; B, treatment group of pink juice sacs stained by phloroglucinol.
表 1 CmCCR基因ORF引物序列
Table 1 Details of CmCCR ORF primers
引物
Primers引物序列
Primer sequencesCmCCR1-Xba I-F GCTCTAGAATGACGGTAATTGATAGC CmCCR1-Kpn I-R GGGGTACCGAATTTTCACTGATTC 表 2 qRT-PCR所用引物信息
Table 2 qRT-PCR primers applied
引物名称
Primer name引物序列
Primer sequencesq-CmCCR1 F: TTGATGAATCTTGCTGGAGTG R: GTCTTTGCCGAGCCATTTA Actin F: AGAACTATGAACTGCCTGATGGC R: GCTTGGAGCAAGTGCTGTGATT -
[1] 潘鹤立, 潘腾飞, 佘文琴, 等. 红绵蜜柚和三红蜜柚的园艺性状与丰产优质栽培技术 [J]. 中国南方果树, 2020, 49(4):143−148. PAN H L, PAN T F, SHE W Q, et al. Horticultural characters of hongmianmiyou and sanhongmiyou pomelos and cultivation techniques for high yield and good quality [J]. South China Fruits, 2020, 49(4): 143−148. (in Chinese)
[2] 潘东明, 郑国华, 陈桂信, 等. 琯溪蜜柚汁胞粒化原因分析 [J]. 果树科学, 1999, 16(3):202−209. PAN D M, ZHENG G H, CHEN G X, et al. Analysis of the reasons caused granulation of juice sacs in guanximiyou pomelo variety [J]. Journal of Fruit Science, 1999, 16(3): 202−209. (in Chinese)
[3] 潘腾飞, 朱学亮, 潘东明, 等. ‘琯溪蜜柚’贮藏期间汁胞粒化与木质素代谢的关系 [J]. 果树学报, 2013, 30(2):294−298. PAN T F, ZHU X L, PAN D M, et al. Relationship between granulation and lignin metabolism in ‘Guanximiyou’pummelo fruit during storage [J]. Journal of Fruit Science, 2013, 30(2): 294−298. (in Chinese)
[4] LI X T, HUANG H T, RIZWAN H M, et al. Transcriptome analysis reveals candidate lignin-related genes and transcription factors during fruit development in pomelo (Citrus maxima) [J]. Genes, 2022, 13(5): 845. DOI: 10.3390/genes13050845
[5] NEUTELINGS G. Lignin variability in plant cell walls: Contribution of new models [J]. Plant Science, 2011, 181(4): 379−386. DOI: 10.1016/j.plantsci.2011.06.012
[6] VANHOLME R, DE MEESTER B, RALPH J, et al. Lignin biosynthesis and its integration into metabolism [J]. Current Opinion in Biotechnology, 2019, 56: 230−239. DOI: 10.1016/j.copbio.2019.02.018
[7] YADAV S, CHATTOPADHYAY D. Lignin: The building block of defense responses to stress in plants [J]. Journal of Plant Growth Regulation, 2023, 42(10): 6652−6666. DOI: 10.1007/s00344-023-10926-z
[8] LEE M H, JEON H S, KIM S H, et al. Lignin-based barrier restricts pathogens to the infection site and confers resistance in plants [J]. The EMBO Journal, 2019, 38(23): e101948. DOI: 10.15252/embj.2019101948
[9] VANHOLME R, DEMEDTS B, MORREEL K, et al. Lignin biosynthesis and structure [J]. Plant Physiology, 2010, 153(3): 895−905. DOI: 10.1104/pp.110.155119
[10] VAN ACKER R, LEPLÉ J C, AERTS D, et al. Improved saccharification and ethanol yield from field-grown transgenic poplar deficient in cinnamoyl-CoA reductase [J]. Proceedings of the National Academy of Sciences of the United States of America, 2014, 111(2): 845−850.
[11] LI W, HAO Z Y, YANG L C, et al. Genome-wide identification and characterization of LcCCR13 reveals its potential role in lignin biosynthesis in Liriodendron chinense [J]. Frontiers in Plant Science, 2023, 13: 1110639. DOI: 10.3389/fpls.2022.1110639
[12] LACOMBE E, HAWKINS S, VAN DOORSSELAERE J, et al. Cinnamoyl CoA reductase, the first committed enzyme of the lignin branch biosynthetic pathway: Cloning, expression and phylogenetic relationships [J]. Plant Journal, 1997, 11(3): 429−441. DOI: 10.1046/j.1365-313X.1997.11030429.x
[13] CUI W T, ZHUANG Z H, JIANG P H, et al. Characterization, expression profiling, and biochemical analyses of the Cinnamoyl-CoA reductase gene family for lignin synthesis in alfalfa plants [J]. International Journal of Molecular Sciences, 2022, 23(14): 7762. DOI: 10.3390/ijms23147762
[14] LEPLÉ J C, DAUWE R, MORREEL K, et al. Downregulation of cinnamoyl-coenzyme A reductase in poplar: Multiple-level phenotyping reveals effects on cell wall polymer metabolism and structure [J]. The Plant Cell, 2007, 19(11): 3669−3691. DOI: 10.1105/tpc.107.054148
[15] GOUJON T, FERRET V, MILA I, et al. Down-regulation of the AtCCR1 gene in Arabidopsis thaliana: Effects on phenotype, lignins and cell wall degradability [J]. Planta, 2003, 217(2): 218−228. DOI: 10.1007/s00425-003-0987-6
[16] CHEN F, DIXON R A. Lignin modification improves fermentable sugar yields for biofuel production [J]. Nature Biotechnology, 2007, 25(7): 759−761. DOI: 10.1038/nbt1316
[17] 谢诗. 缺硼对两种柑橘砧木木质素含量及其合成关键基因表达的影响[D]. 武汉: 华中农业大学, 2014. XIE S. Effects of boron deficiency on lignin content and the key gene expression of lignin biosynthesis in two kinds citrus rootstocks[D]. Wuhan: Huazhong Agricultural University, 2014. (in Chinese)
[18] ANTEROLA A M, LEWIS N G. Trends in lignin modification: A comprehensive analysis of the effects of genetic manipulations/mutations on lignification and vascular integrity [J]. Phytochemistry, 2002, 61(3): 221−294. DOI: 10.1016/S0031-9422(02)00211-X
[19] LIVAK K J, SCHMITTGEN T D. Analysis of relative gene expression data using real-time quantitative PCR and the 2 (-Delta Delta C(T)) Method [J]. Methods, 2001, 25(4): 402−408. DOI: 10.1006/meth.2001.1262
[20] FUKUSHIMA R S, KERLEY M S. Use of lignin extracted from different plant sources as standards in the spectrophotometric acetyl bromide lignin method [J]. Journal of Agricultural and Food Chemistry, 2011, 59(8): 3505−3509. DOI: 10.1021/jf104826n
[21] BARAKAT A, YASSIN N B M, PARK J S, et al. Comparative and phylogenomic analyses of cinnamoyl-CoA reductase and cinnamoyl-CoA-reductase-like gene family in land plants [J]. Plant Science, 2011, 181(3): 249−257. DOI: 10.1016/j.plantsci.2011.05.012
[22] BARROS J, SERK H, GRANLUND I, et al. The cell biology of lignification in higher plants [J]. Annals of Botany, 2015, 115(7): 1053−1074. DOI: 10.1093/aob/mcv046
[23] LAUVERGEAT V, LACOMME C, LACOMBE E, et al. Two cinnamoyl-CoA reductase (CCR) genes from Arabidopsis thaliana are differentially expressed during development and in response to infection with pathogenic bacteria [J]. Phytochemistry, 2001, 57(7): 1187−1195. DOI: 10.1016/S0031-9422(01)00053-X
[24] CHAO N, LI N, QI Q, et al. Characterization of the cinnamoyl-CoA reductase (CCR) gene family in Populus tomentosa reveals the enzymatic active sites and evolution of CCR [J]. Planta, 2017, 245(1): 61−75. DOI: 10.1007/s00425-016-2591-6
[25] MANDROU E, HEIN P R G, VILLAR E, et al. A candidate gene for lignin composition in Eucalyptus: Cinnamoyl-CoA reductase (CCR) [J]. Tree Genetics & Genomes, 2012, 8(2): 353−364.
[26] CHAO N, JIANG W T, WANG X C, et al. Novel motif is capable of determining CCR and CCR-like proteins based on the divergence of CCRs in plants [J]. Tree Physiology, 2019, 39(12): 2019−2026. DOI: 10.1093/treephys/tpz098
[27] WU D, NI M, LEI X, et al. Analyses of pepper cinnamoyl-CoA reductase gene family and cloning of CcCCR1/2 and their function identification in the formation of pungency [J]. Horticulturae, 2022, 8(6): 537. DOI: 10.3390/horticulturae8060537
[28] WANG X, ZHANG Z X, WANG W X, et al. Functional identification of CCR1 gene in apple (Malus halliana) demonstrates that it enhances saline-alkali stress tolerance [J]. Chemical and Biological Technologies in Agriculture, 2024, 11(1): 45. DOI: 10.1186/s40538-024-00565-1
[29] JONES L, ENNOS A R, TURNER S R. Cloning and characterization of irregular xylem4 (irx4): A severely lignin-deficient mutant of Arabidopsis [J]. Plant Journal, 2001, 26(2): 205−216. DOI: 10.1046/j.1365-313x.2001.01021.x
[30] YANG Z, LIU Z H, XU H, et al. ArecaceaeMDB: A comprehensive multi-omics database for Arecaceae breeding and functional genomics studies [J]. Plant Biotechnology Journal, 2023, 21(1): 11−13. DOI: 10.1111/pbi.13945
[31] YIN N W,LI B,LIU X,et al. Two types of cinnamoyl-CoA reductase function divergently in accumulation of lignins, flavonoids and glucosinolates and enhance lodging resistance in Brassica napus [J]. The Crop Journal, 2022, 10(3): 647−660.
[32] PICHON M, COURBOU I, BECKERT M, et al. Cloning and characterization of two maize cDNAs encoding cinnamoyl-CoA reductase (CCR) and differential expression of the corresponding genes [J]. Plant Molecular Biology, 1998, 38(4): 671−676. DOI: 10.1023/A:1006060101866
[33] LI J, FAN F, WANG L, et al. Cloning and expression analysis of cinnamoyl-CoA reductase (CCR) genes in sorghum[J]. PeerJ, 2016, 4(5): e2005.
[34] 卢晓鹏. 砂梨(Pyrus pyrifolia Nakai)果实石细胞、褐色果皮和柠檬酸形成机制研究[D]. 武汉: 华中农业大学, 2011 LU X P. Mechanism researches on the formation of stone cell, russet skin and citric acid of pear (Pyrus pyrifolia Nakai) fruit[D]. Wuhan: Huazhong Agricultural University, 2011. (in Chinese)




 下载:
下载: